9.3 Test t de Student
Nous allons également pouvoir utiliser la distribution t de Student comme distribution de référence pour comparer une moyenne par rapport à une valeur cible ou pour comparer deux moyennes. C’est le test t de Student… ou plutôt les tests de Student puisqu’il en existe plusieurs variantes.
Partons d’un exemple concret. Imaginez que vous êtes des biologistes ouest-australiens travaillant à Freemantle. Vous y étudiez le crabe Leptographus variegatus (Fabricius, 1793). C’est un crabe qui peut se trouver en populations abondantes sur les côtes rocheuses fortement battues. Il a un régime alimentaire partiellement détritivore et partiellement carnivore.

Crabe Leptograpsus variegatus par Johnragla.
Ce crabe est rapide et difficile à capturer… mais vous avez quand même réussi à en attraper et mesurer 200 d’entre eux, ce qui constitue un échantillon de taille raisonnable. Comme deux variétés co-existent, la variété bleue (B) et la variété orange (O) sur votre site d’étude, vous vous demandez si elles diffèrent d’un point de vue morphométrique. Naturellement, nous pouvons également supposer des différences entre mâles et femelles. Vous avez donc décidé de réaliser un échantillonnage stratifié consistant à capturer et mesurer autant de bleus que d’oranges et autant de mâles que de femelles. Vous avez donc 50 mâles bleus, 50 femelles bleues, 50 mâles oranges et 50 femelles oranges.
crabs <- read("crabs", package = "MASS", lang = "fr")
skimr::skim(crabs)# Skim summary statistics
# n obs: 200
# n variables: 8
#
# ── Variable type:factor ───────────────────────────────────────────────────────────────────────────────────────────────
# variable missing complete n n_unique top_counts ordered
# sex 0 200 200 2 F: 100, M: 100, NA: 0 FALSE
# species 0 200 200 2 B: 100, O: 100, NA: 0 FALSE
#
# ── Variable type:integer ──────────────────────────────────────────────────────────────────────────────────────────────
# variable missing complete n mean sd p0 p25 p50 p75 p100 hist
# index 0 200 200 25.5 14.47 1 13 25.5 38 50 ▇▇▇▇▇▇▇▇
#
# ── Variable type:numeric ──────────────────────────────────────────────────────────────────────────────────────────────
# variable missing complete n mean sd p0 p25 p50 p75 p100
# depth 0 200 200 14.03 3.42 6.1 11.4 13.9 16.6 21.6
# front 0 200 200 15.58 3.5 7.2 12.9 15.55 18.05 23.1
# length 0 200 200 32.11 7.12 14.7 27.28 32.1 37.23 47.6
# rear 0 200 200 12.74 2.57 6.5 11 12.8 14.3 20.2
# width 0 200 200 36.41 7.87 17.1 31.5 36.8 42 54.6
# hist
# ▂▃▆▇▇▆▅▂
# ▂▃▇▇▇▇▅▃
# ▁▃▅▇▇▆▅▂
# ▂▃▇▆▇▃▂▁
# ▁▃▅▇▇▆▃▁Toutes les variables qualtitatives sont des mesures effectuées sur la carapace des crabes. Nous nous posons la question suivante :
- Les femelles ont-elle une carapace plus large à l’arrière, en moyenne que les mâles ?
Voici une comparaison graphique :
chart(data = crabs, rear ~ sex) +
geom_boxplot()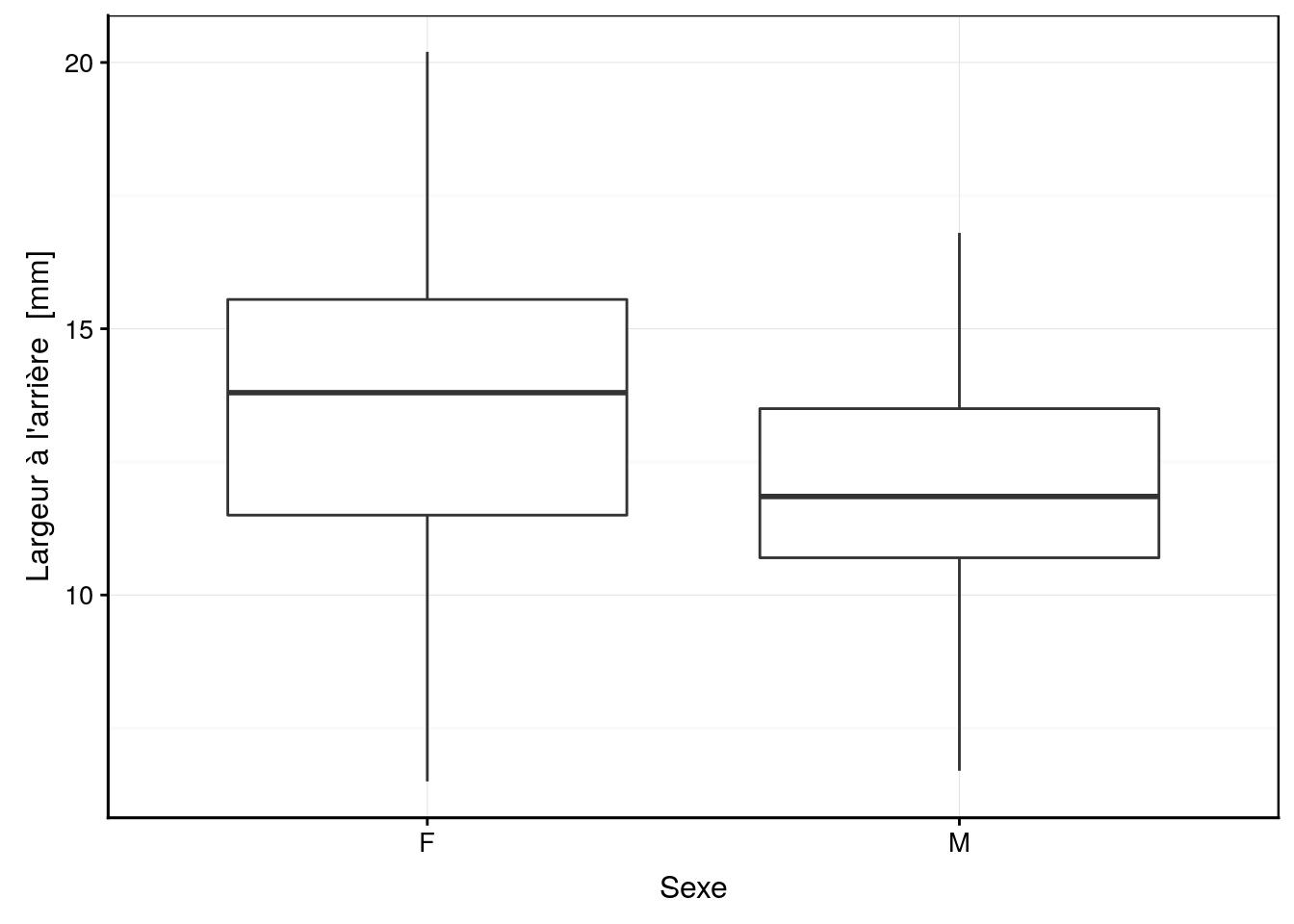
Sur le graphique, il semble que les femelles (sex == "F") tendent à avoir une carapace plus large à l’arrière -variable rear- que les mâles (sex == "M"), mais cette différence est-elle significative ou est-elle juste liée au hasard de l’échantillonnage ? Pour y répondre, nous devons élaborer un test d’hypothèse qui confrontera les hypothèses suivantes (en se basant sur les moyennes) :
- \(H_0: \overline{rear_F} = \overline{rear_M}\)
- \(H_1: \overline{rear_F} \neq \overline{rear_M}\)
Ici, nous n’avons aucune idée a priori pour \(H_1\) si les femelles sont sensées avoir une carapace plus large ou non que les mâles à l’arrière. Donc, nous considérons qu’elle peut être aussi bien plus grande que plus petite. On parle ici de test bilatéral car la différence peut apparaître des deux côtés. Pour ce test, nous pouvons partir de la notion d’intervalle de confiance et de notre idée de calculer les quantiles de part et d’autre de la distribution théorique à parts égales, comme dans la Fig. 9.2.
Une idée serait de calculer \(\overline{rear_F} - \overline{rear_M}\), la différence des moyennes entre mesures pour les femelles et pour les mâles. Les hypothèses deviennent alors :
- \(H_0: \overline{rear_F} - \overline{rear_M} = 0\)
- \(H_1: \overline{rear_F} - \overline{rear_M} \neq 0\)
Appelons cette différence \(\Delta rear\). Nous pouvons définir un intervalle de confiance pour \(\Delta rear\) si nous pouvons calculer la valeur t ainsi que l’erreur standard \(SE_{\Delta rear}\) associées à cette variables calculée. Après avoir interrogé des statisticiens chevronnés, ceux-ci nous proposent l’équation suivante pour \(SE_{\Delta rear}\) (avec \(n_F\) le nombre de femelles et \(n_M\) le nombre de mâles) :
\[SE_{\Delta rear} = \sqrt{SE_{rear_F}^2 + SE_{rear_M}^2} = \sqrt{\frac{s_{rear_F}^2}{n_F} + \frac{s_{rear_M}^2}{n_M}}\]
Il nous reste à déterminer les degrés de liberté associés à la distribution t. Les statisticiens nous disent qu’il s’agit de n moins deux degrés de libertés. Nous obtenons alors l’équation suivante pour l’intervalle de confiance :
\[\mathrm{IC}(1 - \alpha)_{\Delta rear} \simeq \Delta rear \pm t_{\alpha/2}^{n-2} \cdot SE_{\Delta rear}\]
Dans notre cas, cela donne :
crabs %>.%
group_by(., sex) %>.%
summarise(., mean = mean(rear), var = var(rear), n = n()) ->
crabs_stats
crabs_stats# # A tibble: 2 x 4
# sex mean var n
# <fct> <dbl> <dbl> <int>
# 1 F 13.5 7.51 100
# 2 M 12.0 4.67 100# Calcul de Delta rear et de son intervalle de confiance à 95%
(delta_rear <- crabs_stats$mean[1] - crabs_stats$mean[2])# [1] 1.497(t <- qt(0.025, nrow(crabs) - 2))# [1] -1.972017(se <- sqrt(crabs_stats$var[1] / crabs_stats$n[1] + crabs_stats$var[2] / crabs_stats$n[2]))# [1] 0.3489874(ic_95 <- c(delta_rear + t * se, delta_rear - t * se))# [1] 0.8087907 2.1852093Un premier raisonnement consiste à dire que si la valeur attendue sous \(H_0\) est comprise dans l’intervalle de confiance, nous ne pouvons pas rejetter l’hypothèse nulle, puisqu’elle représente une des valeurs plausibles à l’intérieur l’IC. Dans le cas présent, l’intervalle de confiance à 95% sur \(\Delta rear\) va de 0.81 à 2.19. Il ne contient donc pas zéro. Donc, nous pouvons rejetter \(H_0\) au seuil \(\alpha\) de 5%.
Nous pouvons effectivement interpréter le test de cette façon, mais le test t de Student se définit de manière plus classique en comparant la valeur \(t_{obs}\) à la distribution théorique, et en renvoyant une valeur P associée au test. Ainsi, le lecteur peut interpréter les résultats avec son propre seuil \(\alpha\) éventuellement différent de celui choisi par l’auteur de l’analyse.
Le raisonnement est le suivant. Sous \(H_0\), la distribution de \(\Delta rear\) est connue. Elle suit une distribution t de Student de moyenne égale à la vraie valeur de la différence des moyennes, d’écart type égal à l’erreur standard sur cette différence, et avec \(n - 2\) degrés de liberté. En pratique, nous remplaçons les valeurs de la population pour la différence des moyennes et pour les erreurs standard par celles estimées par l’intermédiaire de l’échantillon. Comme dans le cas du test \(\chi^2\), nous définissons les zones de rejet et de non rejet par rapport à cette distribution théorique. Dans le cas du test de Student bilatéral, l’aire \(\alpha\) est répartie à moitié à gauche et à moitié à droite (Fig. 9.6).
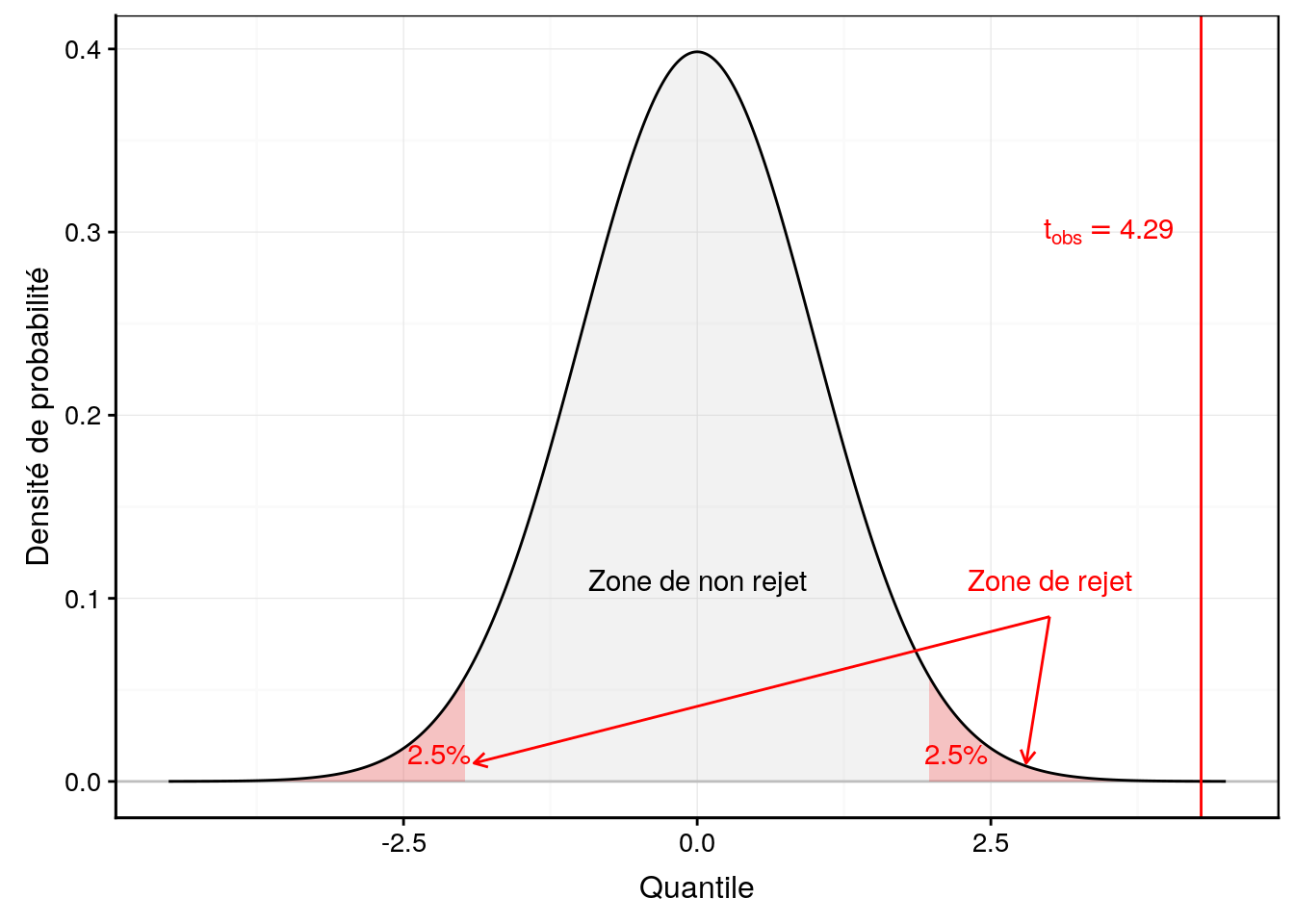
Figure 9.6: Visualisation de la distribution de Student réduite sous l’hypothèse nulle du test bilatéral au seuil de 5%.
Nous pouvons calculer la valeur P nous-même comme ceci, sachant la valeur de \(t_{obs} = \frac{\Delta rear}{SE_{\Delta rear}}\) parce que nous travaillons avec une distribution t réduite :
(t_obs <- delta_rear / se)# [1] 4.289553(p_value <- pt(t_obs, df = 198, lower.tail = FALSE) * 2)# [1] 2.797369e-05Ne pas oublier de multiplier la probabilité obtenue par deux, car nous avons un test bilatéral qui considère une probabilité égale à gauche et à droite de la distribution !
Naturellement, R propose une fonction toute faite pour réaliser ce test afin que nous ne devions pas détailler les calculs à chaque fois. Il s’agit de la fonction t.test(). Dans la SciViews Box, le snippet équivalent est accessible depuis .hm pour hypothesis tests: means. Dans le menu qui apparait, vous choisissez independant Student's t-test. Les arguments de la fonction sont les suivants. Le jeu de données dans data =, une formule qui reprend le nom de la variable quantitative à gauche (rear) et celui de la variable qualitative à deux niveaux à droite (sex), l’idication du type d’hypothèse alternative, ici alternative = "two-sided" pour un test bilatéral, le niveau de confiance égal à \(1 - \alpha\), donc conf.level = 0.95 et enfin si nous considérons les variances comme égales pour les deux sous-populations var.equal = TRUE.
t.test(data = crabs, rear ~ sex,
alternative = "two.sided", conf.level = 0.95, var.equal = TRUE)#
# Two Sample t-test
#
# data: rear by sex
# t = 4.2896, df = 198, p-value = 2.797e-05
# alternative hypothesis: true difference in means is not equal to 0
# 95 percent confidence interval:
# 0.8087907 2.1852093
# sample estimates:
# mean in group F mean in group M
# 13.487 11.990Nous retrouvons exactement toutes les valeurs que nous avons calculées à la main. Dans le cas présent, rappelez-vous la façon d’interpréter le test. Nous comparons la valeur P à \(\alpha\). Si elle est plus petit, nous rejettons \(H_0\), sinon, nous ne la rejettons pas. Ici, nous rejettons \(H_0\) et pourrons dire que la largeur à l’arrière de la carapace de L. variegatus diffère de manière significative entre les mâles et les femelles au seuil \(\alpha\) de 5% (test t bilatéral, t = 4,29, ddl = 198, valeur P << 10-3).
Conditions d’application
- échantillon représentatif (échantillonnage aléatoire et individus indépendants les uns des autres),
- observations indépendantes les unes des autres,
- une variable numérique et une variable facteur à deux niveaux,
- distribution de la population…
- normale, alors le test basé sur la distribution t de Student sera exact,
- approximativement normale, le test sera approximativement exact,
- non normale, le test sera approximativement exact si \(n\) est grand.
Petite astuce… les mesures morphométriques sont dépendantes de la taille globale de l’animal qui varie d’un individu à l’autre, il vaut donc mieux étudier des rapports de tailles plutôt que des mesures absolues. Refaites le calcul sur base du ratio rear / length comme exercice et déterminez si la différence est plus ou moins nette entre les mâles et les femelles que dans le cas de rear seul.
Vous pouvez également comparer les crabes bleus (species = “B”) avec les crabes oranges (species = “O”) à l’aide du même test.
A vous de jouer !
Appliquez les test de student dans votre projet portant sur la biométrie humaine.
Pour en savoir plus
- Une vidéo en anglais qui explique le test t de Student un peu différemment.