4.5 Différents moteurs graphiques

Prolifération des standards d’après xkcd.
Depuis le début, l’ensemble des graphiques que nous vous avons proposés utilise la fonction chart() du package chart. Cependant, il ne s’agit pas de la seule fonction permettant de réaliser des graphiques dans R, loin de là. En fait, chart est tout récent et a été développé pour homogénéiser autant que possible les graphiques issus de trois moteurs graphiques différents : ggplot2, lattice et les graphiques base. La fonction chart() a d’autres avantages également :
- Un thème par défaut qui est le plus proche possible d’un rendu typique d’une publication scientifique.
- La possibilité d’utiliser l’interface formule avec ggplot2.
- La cohérence des objets graphiques obtenus qui peuvent tous êtres combinés en une figure composée, même si ils sont produits avec des moteurs graphiques différents.
- Un libellé automatique des axes et autres éléments du graphique en fonction des attributs
labeletunitsdes variables (pour l’instant, seulement les graphiques de type ggplot2).
# Importation des données
(urchin <- read("urchin_bio", package = "data.io", lang = "FR"))# # A tibble: 421 x 19
# origin diameter1 diameter2 height buoyant_weight weight solid_parts
# <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# 1 Pêche… 9.9 10.2 5 NA 0.522 0.478
# 2 Pêche… 10.5 10.6 5.7 NA 0.642 0.589
# 3 Pêche… 10.8 10.8 5.2 NA 0.734 0.677
# 4 Pêche… 9.6 9.3 4.6 NA 0.370 0.344
# 5 Pêche… 10.4 10.7 4.8 NA 0.610 0.559
# 6 Pêche… 10.5 11.1 5 NA 0.610 0.551
# 7 Pêche… 11 11 5.2 NA 0.672 0.605
# 8 Pêche… 11.1 11.2 5.7 NA 0.703 0.628
# 9 Pêche… 9.4 9.2 4.6 NA 0.413 0.375
# 10 Pêche… 10.1 9.5 4.7 NA 0.449 0.398
# # … with 411 more rows, and 12 more variables: integuments <dbl>,
# # dry_integuments <dbl>, digestive_tract <dbl>,
# # dry_digestive_tract <dbl>, gonads <dbl>, dry_gonads <dbl>,
# # skeleton <dbl>, lantern <dbl>, test <dbl>, spines <dbl>,
# # maturity <int>, sex <fct># Réalisation du graphique
chart(data = urchin, height ~ weight %col=% origin) +
geom_point() 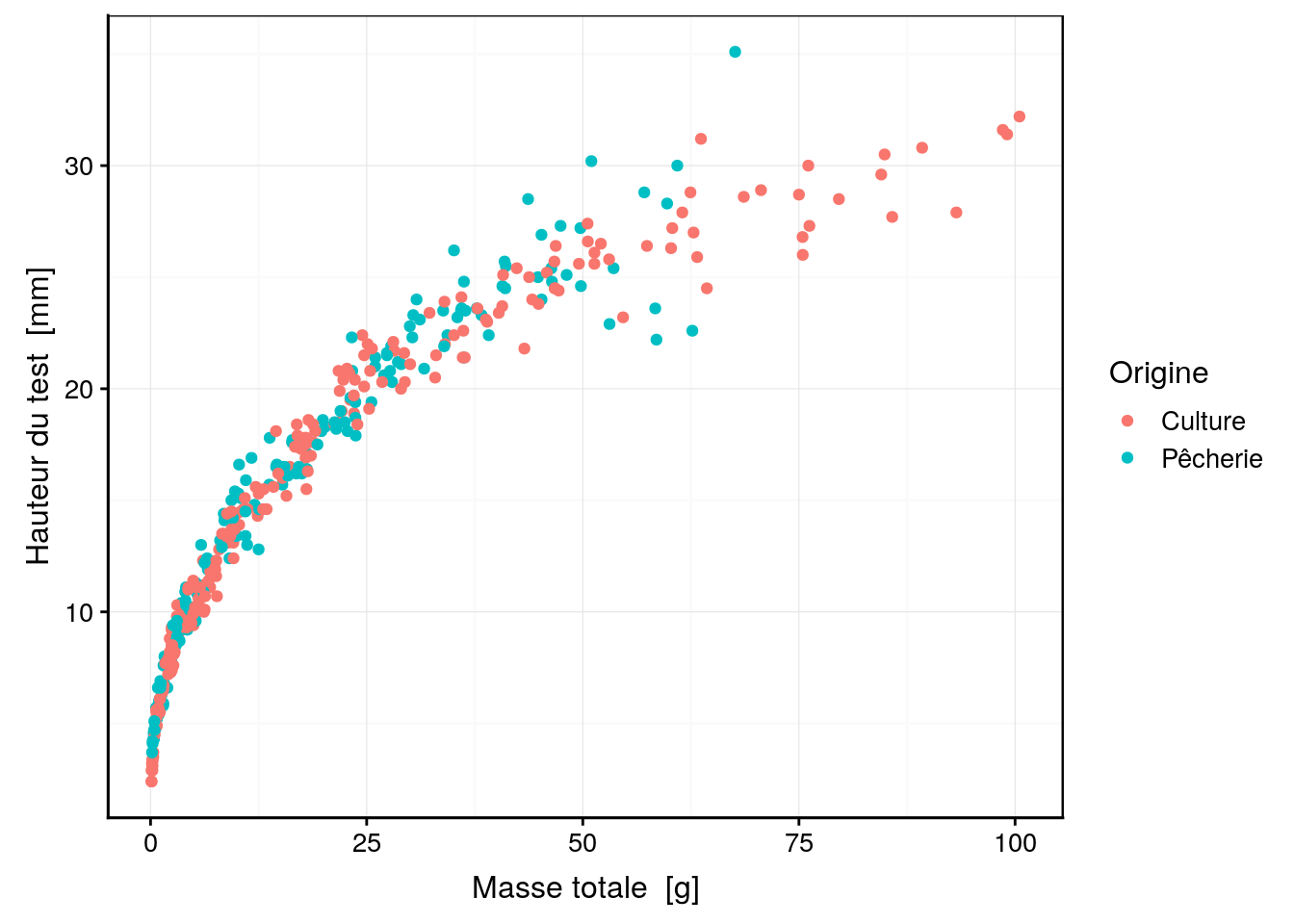
Figure 4.22: Graphique typique obtenu avec chart() : rendu par défaut publiable tel quel, et libellé automatique des axes avec les unités.
4.5.1 ggplot2
Le moteur graphique ggplot2 est écrit pas Hadley Wickham, un personnage emblématique de la “révolution tidyverse” qui propose une surcouche moderne au dessus de R. ggplot2 implémente une “grammaire graphique” particulièrement puissante et flexible, proposée et popularisée par le statisticien Leland Wilkinson. Par défaut, chart() crée en réalité un graphique ggplot2 adapté. Voici la version ggplot2 standard du même graphique représenté à la Fig. 4.22 :
ggplot(data = urchin, mapping = aes(x = weight, y = height, col = origin)) +
geom_point() 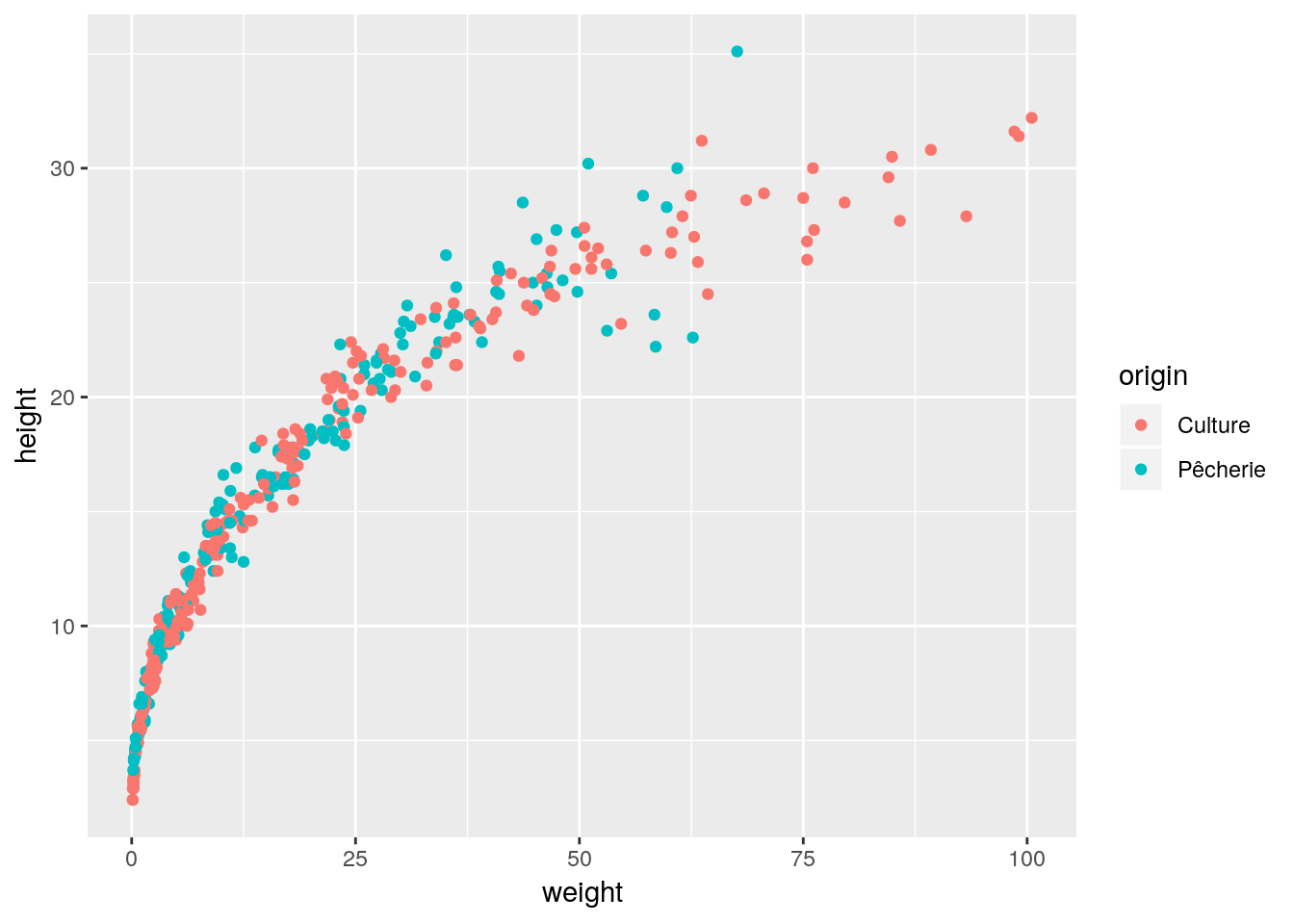
Figure 4.23: Graphique typique obtenu avec ggplot() (moteur graphique ggplot2).
En comparant les Figs. 4.22 et 4.23 (en faisant abstraction des instructions R utilisées pour l’instant), plusieurs points sautent immédiatement aux yeux:
Le thème par défaut de ggplot2 est très reconnaissable avec un quadrillage blanc sur fond gris clair. On aime ou on n’aime pas, mais il est clair que (1) ce n’est pas une présentation “standard” d’un graphique scientifique, et (2) le thème tord un peu le cou à une règle importante pour réaliser un graphique de qualité : minimiser la quantité d’“encre” nécessaire pour représenter un graphique, autrement dit, plus le graphique est simple et sobre, mieux c’est. Le thème par défaut de
chart()respecte mieux tout ceci15.La taille des caractères est légèrement plus grande dans la Fig. 4.22 réalisée avec
chart(). Le manque de lisibilité des parties textuelles dans un graphique est un défaut fréquent, dépendant de la résolution et de la taille de reproduction du graphique dans le document final. Le choix dechart()recule un peu ce risque.chart()est capable d’aller lire les métadonnées (libellés en français et unités des variables) et les utilisent automatiquement pour proposer des libellés corrects et complets des axes par défaut.ggplot()ne peut pas le faire, et il faut utiliser la fonctionlabs()pour l’indiquer manuellement.
ggplot(), chart() a été conçu pour produire le graphique le plus proche d’un rendu final impeccable avec tous les paramètres par défaut.
Quelques règles simples vous permettent de passer des instructions ggplot() à chart() et vice versa16 :
On peut toujours remplacer
ggplot()parchart()dans les instructions R (à condition que le package chart soit chargé bien sûr, par exemple viaSciViews::R). Dans ce cas, le thème par défaut diffère, et le libellé automatique des axes (non disponible avecggplot()) est activé.Avec
chart()on peut utiliseraes()pour spécifier les “esthétiques” (éléments à visualiser sur le graphique) comme pourggplot(), mais on peut aussi utiliser une interface formule plus compacte. Cette interface formule rapproche la versionchart()des graphiques ggplot2 d’un autre moteur de graphique dans R : lattice.Outre les esthétiques classiques
xety, l’interface formule dechart()permet d’en inclure d’autres directement dans la formule à l’aide d’opérateurs spécifiques%<esth>%=. Par exemple,aes(x = weight, y = height, col = origin)dans la Fig. 4.23 se traduit en la formule plus conciseheight ~ weight %col=% originavecchart()(notez la position inversée dexetydans la formule puisqu’on ay ~ x). Tous les esthétiques de ggplot2 sont supportés de cette manière.Partout où
aes()est utilisé pour les instructions ggplot2, on peut utiliser à la placef_aes()et y spécifier plutôt une formule de typechart().Avec
ggplot()les facettes doivent être spécifiées à l’aide defacet_XXX(). A condition d’utiliserchart(), il est possible d’inclure les spécifications des facettes les plus utilisées directement dans la formule en utilisant l’opérateur|. Cette façon de procéder est, encore une fois, identique à ce qui se fait dans lattice (voir plus loin).
Le point (5) mérite une petite démonstration pour comparaison :
a <- chart(data = urchin, height ~ weight | origin) +
geom_point()
b <- ggplot(data = urchin, mapping = aes(x = weight, y = height)) +
geom_point() +
facet_grid( ~ origin)
combine_charts(list(a, b))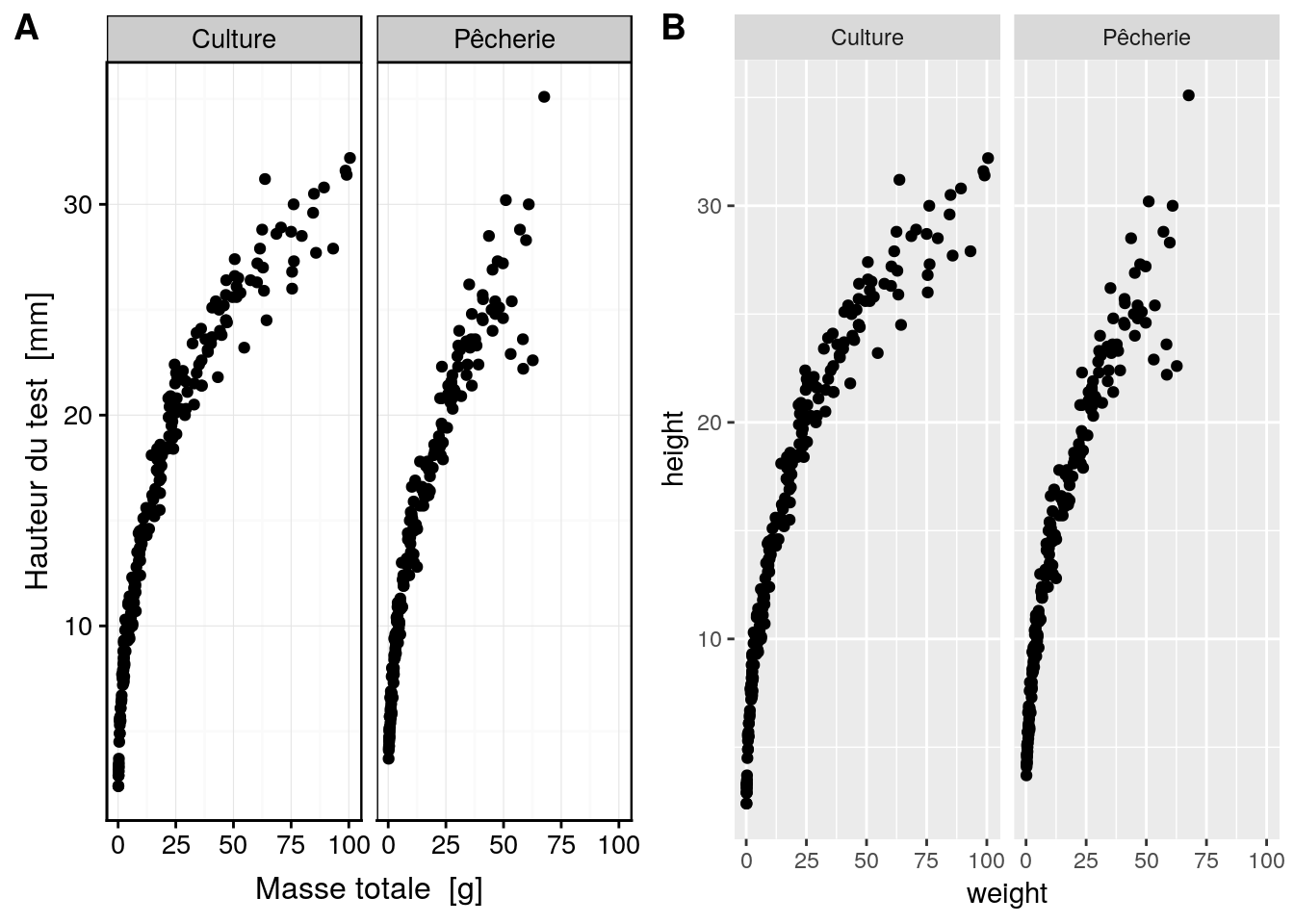
Figure 4.24: Graphique à facettes. A. version chart(), B. version ggplot().
4.5.2 lattice
Autant ggplot2 est complètement modulable en ajoutant littéralement à l’aide de l’opérateur + des couches successives sur le graphique, autant lattice vise à réaliser les graphiques en une seule instruction. lattice utilise également abondamment l’interface formule pour spécifier les variables à utiliser dans le graphique. La version lattice du graphique d’exemple est présentée à la Fig. 4.25.
xyplot(height ~ weight, data = urchin, groups = origin, auto.key = TRUE)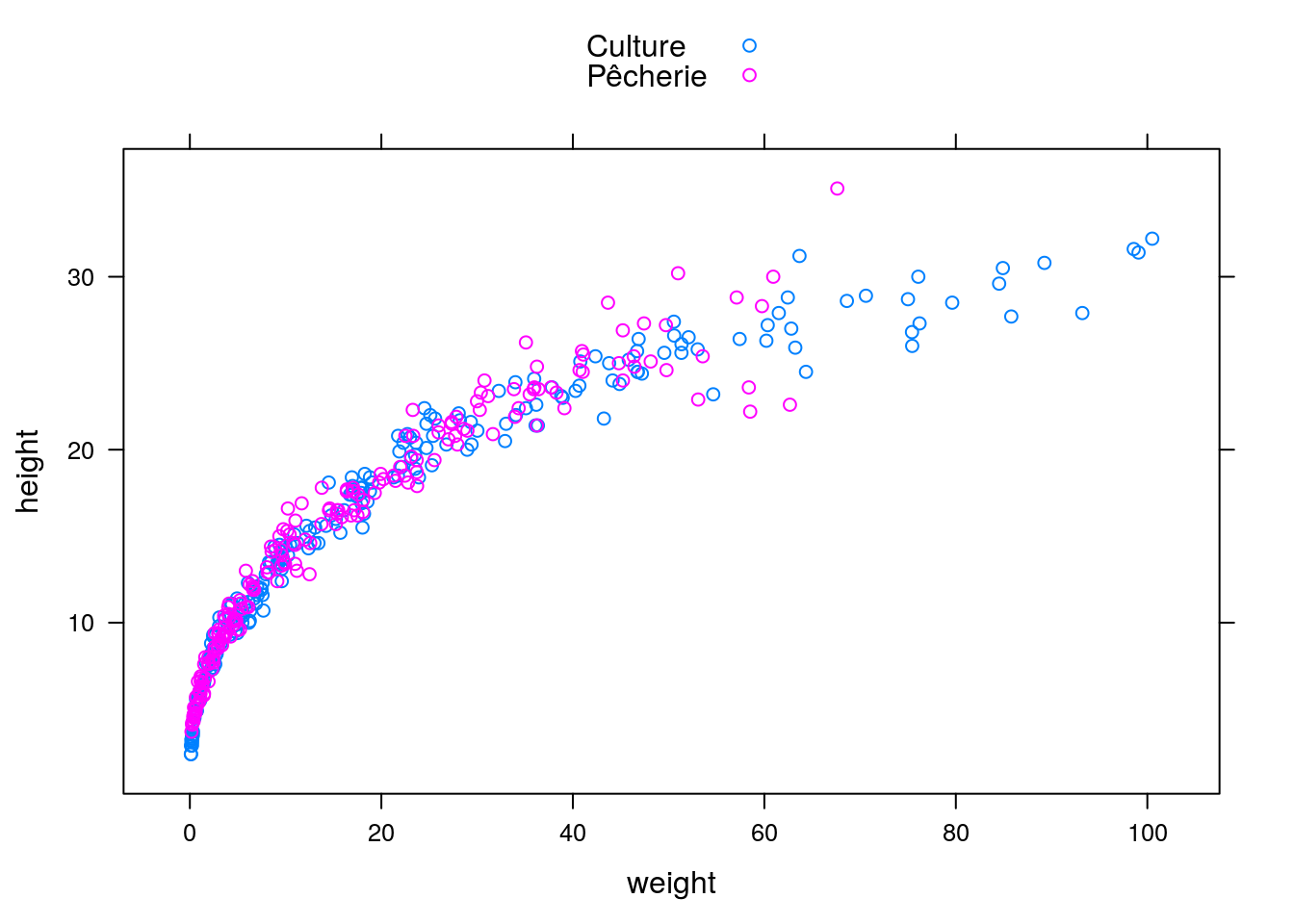
Figure 4.25: Graphique exemple réalisé avec lattice.
Et voici la version chart() utilisant le moteur lattice. Notez la façon d’appeler la fonction xyplot() de lattice via chart$xyplot() :
theme_sciviews_lattice(n = 2)
a <- chart$xyplot(height ~ weight, data = urchin, groups = origin,
auto.key = list(space = "right", title = "Origine", cex.title = 1, columns = 1),
ylab = "Hauteur du test [mm]", xlab = "Masse totale [g]",
par.settings = list(superpose.symbol = list(col = scales::hue_pal()(2))))
b <- chart(data = urchin, height ~ weight %col=% origin) +
geom_point()
combine_charts(list(a, b))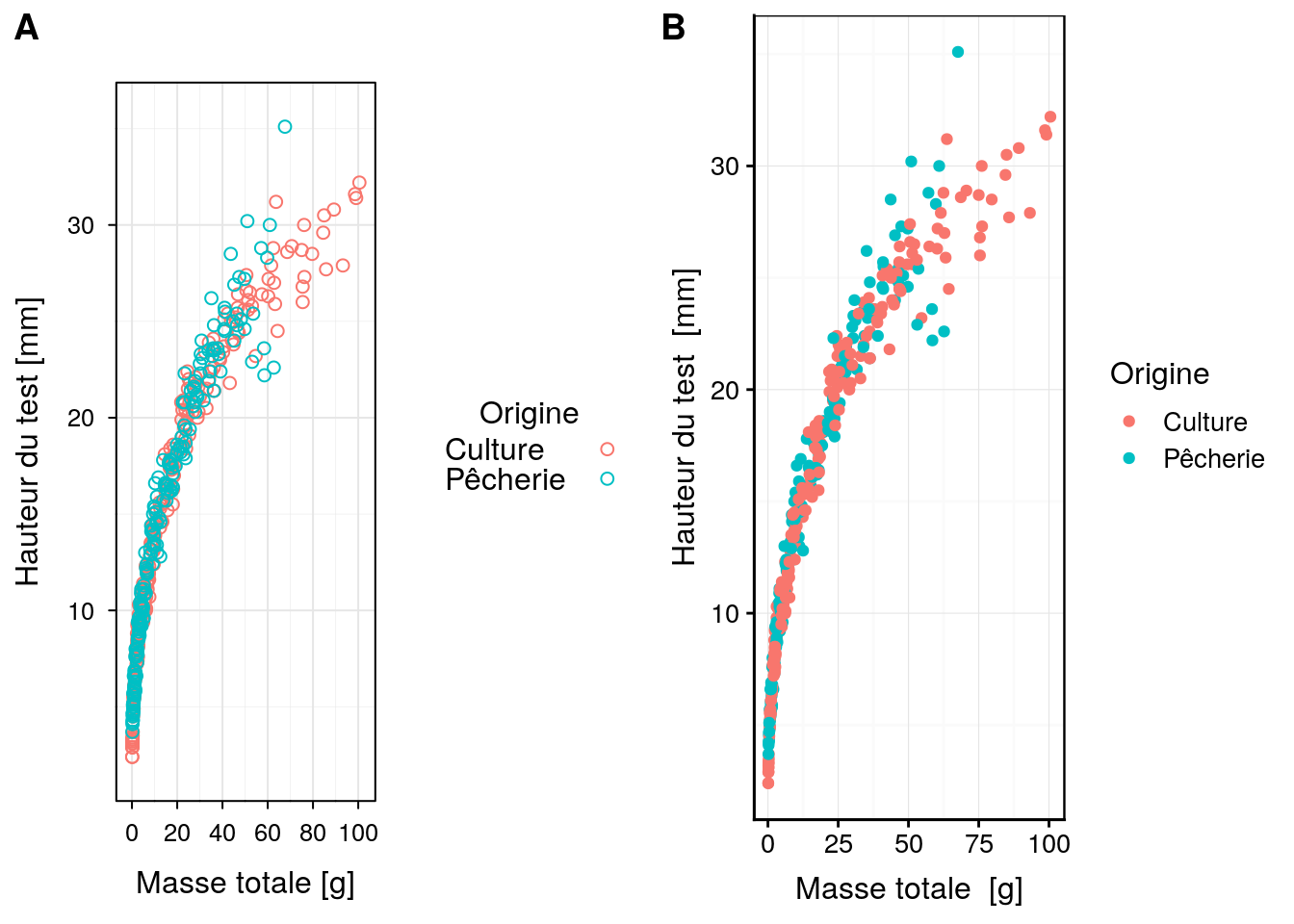
Figure 4.26: Graphique exemple réalisé avec chart() A. avec le moteur lattice, B. avec le moteur ggplot2.
La quantité d’instructions nécessaires pour rendre la version lattice proche de la version ggplot2 devrait disparaître dans les prochaines versions de chart(). Un autre objectif est aussi de gommer le plus possible les différences entre les rendus des différents moteurs de graphiques R, et en particuliers entre ggplot2 et lattice. Comparez la Fig. 4.26A avec la Fig. 4.25 pour apprécier le gain déjà obtenu en matière d’homogénéisation.
Voici un graphique à facettes réalisé avec chart() et le moteur lattice. Notez que la formule utilisée est identique à cette employée pour la version ggplot2 avec chart().
chart$xyplot(data = urchin, height ~ weight | origin,
scales = list(alternating = 1),
xlab = "Masse totale [g]", ylab = "Hauteur du test [mm]")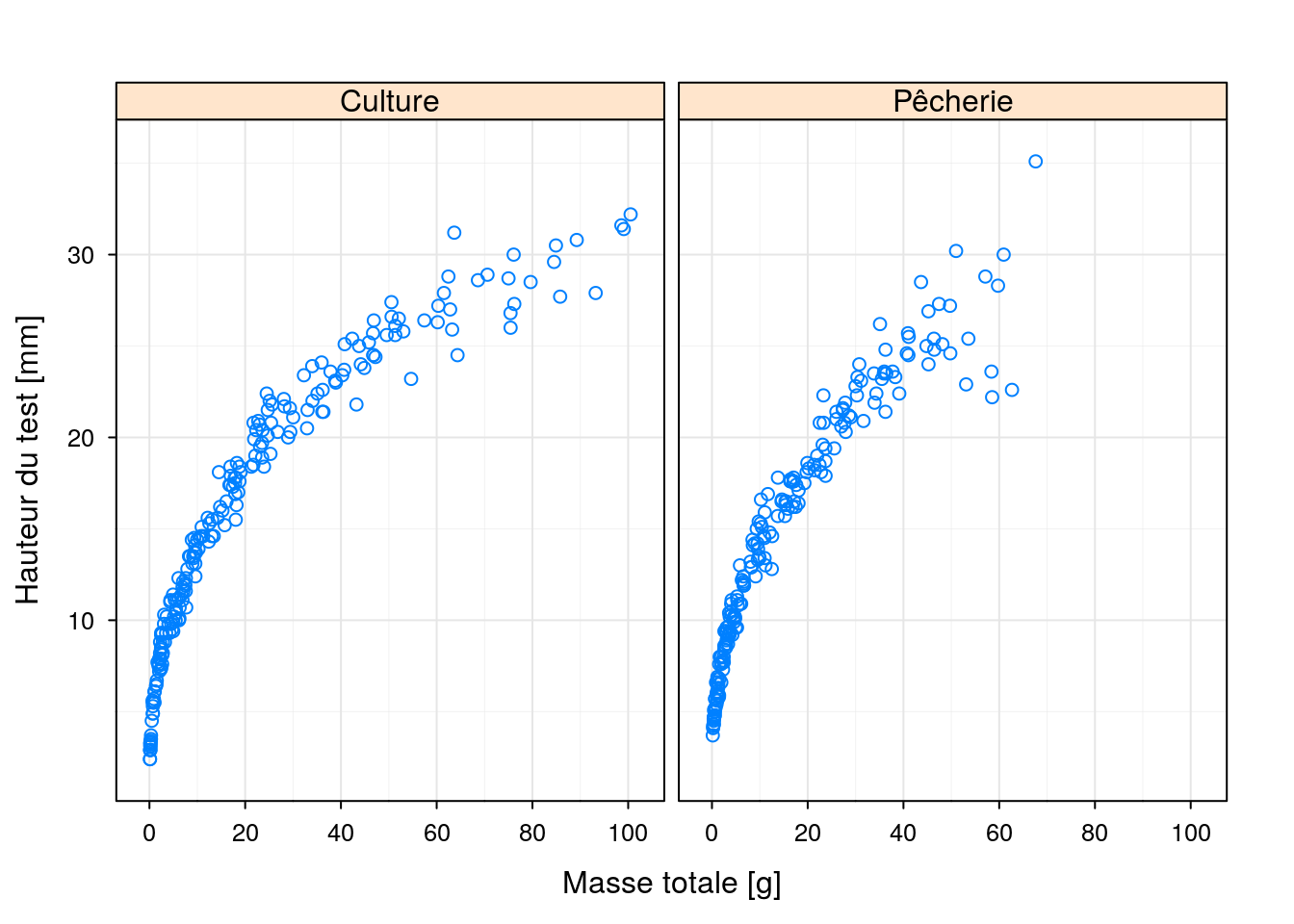
Figure 4.27: Graphique à facettes, avec chart() version lattice.
Mise à part les instructions additionnelles encore nécessaires dans cette version de chart(), l’appel et le rendu sont très similaires par rapport à la version ggplot2 du même graphique avec chart() :
chart(data = urchin, height ~ weight | origin) +
geom_point()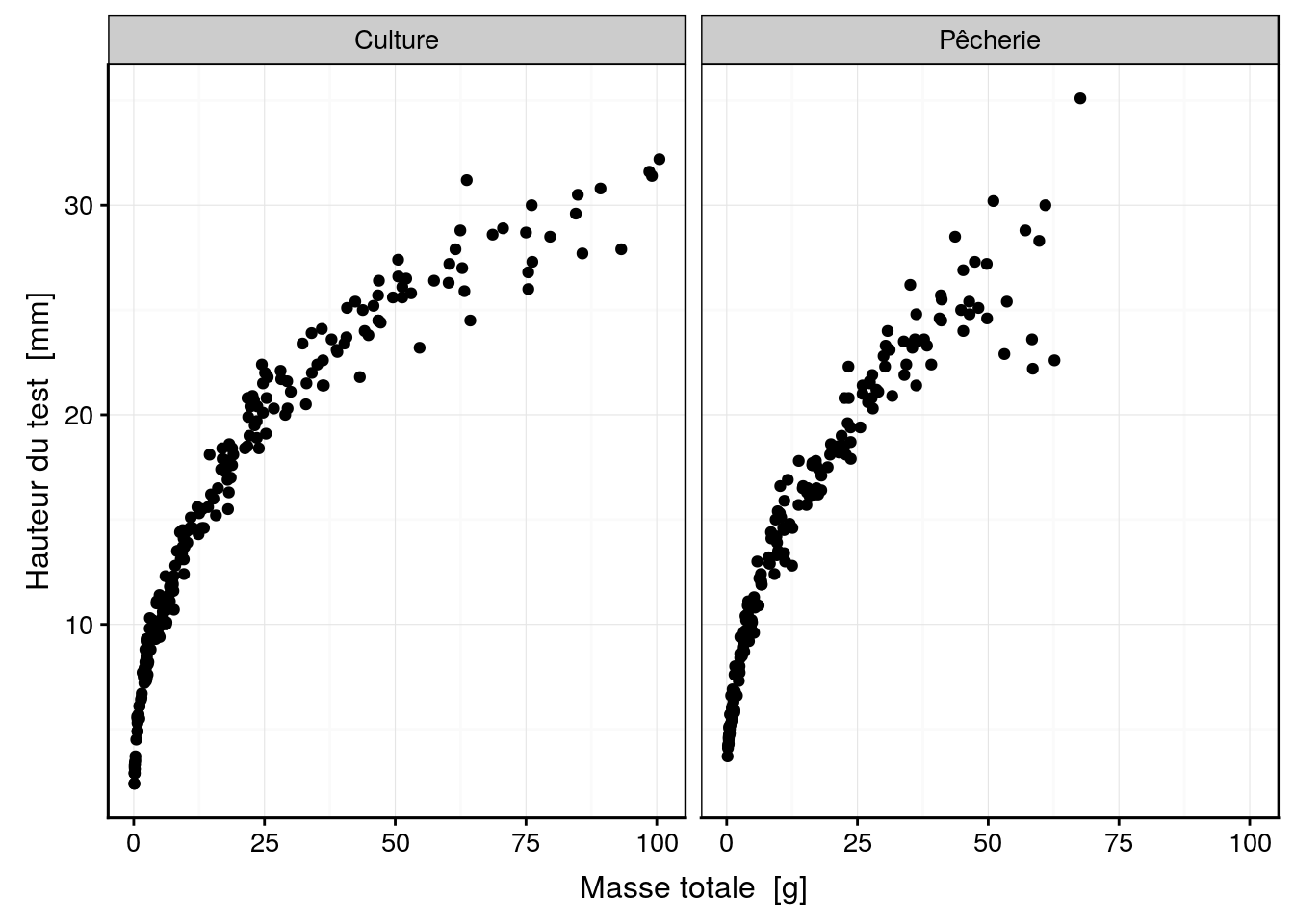
Figure 4.28: Graphique à facettes, avec chart() version ggplot2.
4.5.3 Graphiques de base
Comme son nom le suggère, le moteur graphique de base est celui qui est implémenté de manière natif dans R. Il est donc utilisé un peu partout. Il est vieillissant et est plus difficile à manipuler que ggplot2 certainement, et même que lattice. Néanmoins, il est très flexible et rapide, … mais son rendu par défaut n’est plus vraiment au goût du jour. Voici notre graphique d’exemple rendu avec le moteur graphique R de base :
plot(urchin$weight, urchin$height,
col = c("red", "darkgreen")[urchin$origin], pch = 1)
legend(x = 80, y = 10, legend = c("Culture", "Pêcherie"),
col = c("red", "darkgreen"), pch = 1)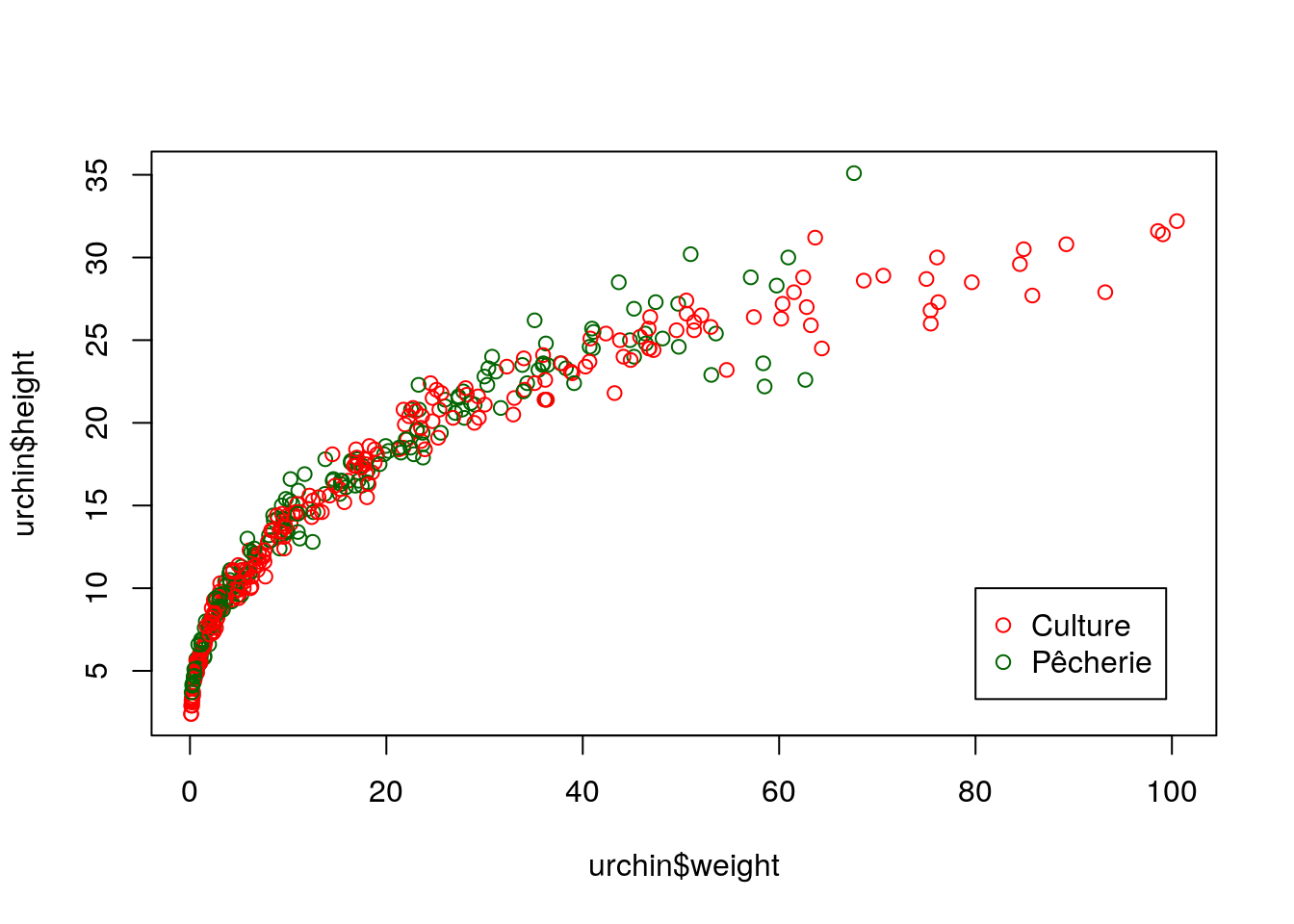
Figure 4.29: Graphique exemple réalisé avec le moteur graphique R de base.
Vous rencontrerez très fréquemment la fonction plot(). C’est une fonction dite générique dont le comportement change en fonction de l’objet fourni en premier argument. Ainsi, elle réalise le graphique le plus pertinent à chaque fois en fonction du contexte. Notez tout de suite les instructions un peu confuses nécessaires pour spécifier la couleur souhaitée en fonction de l’origine des oursins. Le moteur graphique de base ne gère pas automatiquement des aspects plus complexes du graphique, telle que le positionnement d’une légende. Donc, à moins d’avoir prévu la place suffisante avant de tracer le graphique, nous ne pouvons que l’inclure à l’intérieur du cadre du graphique dans un second temps à l’aide de la fonction legend(). Comme cette dernière ne comprend rien à ce qui a été réalisé jusqu’ici, il faut lui respécifier les couleurs, formes et tailles de points utilisés ! C’est un des aspects pénibles du moteur graphique R de base.
Voici maintenant une version chart() de ce graphique de base :
chart$base({
par(mar = c(5.1, 4.1, 4.1, 6.1))
plot(urchin$weight, urchin$height,
col = scales::hue_pal()(2)[urchin$origin], pch = 19, cex = 0.8,
xlab = "Masse totale [g]", ylab = "Hauteur du test [mm]")
legend(x = 105, y = 20, legend = c("Culture", "Pêcherie"), title = "Origine",
col = scales::hue_pal()(2), pch = 19, bty = "n", cex = 0.8, y.intersp = 2)
})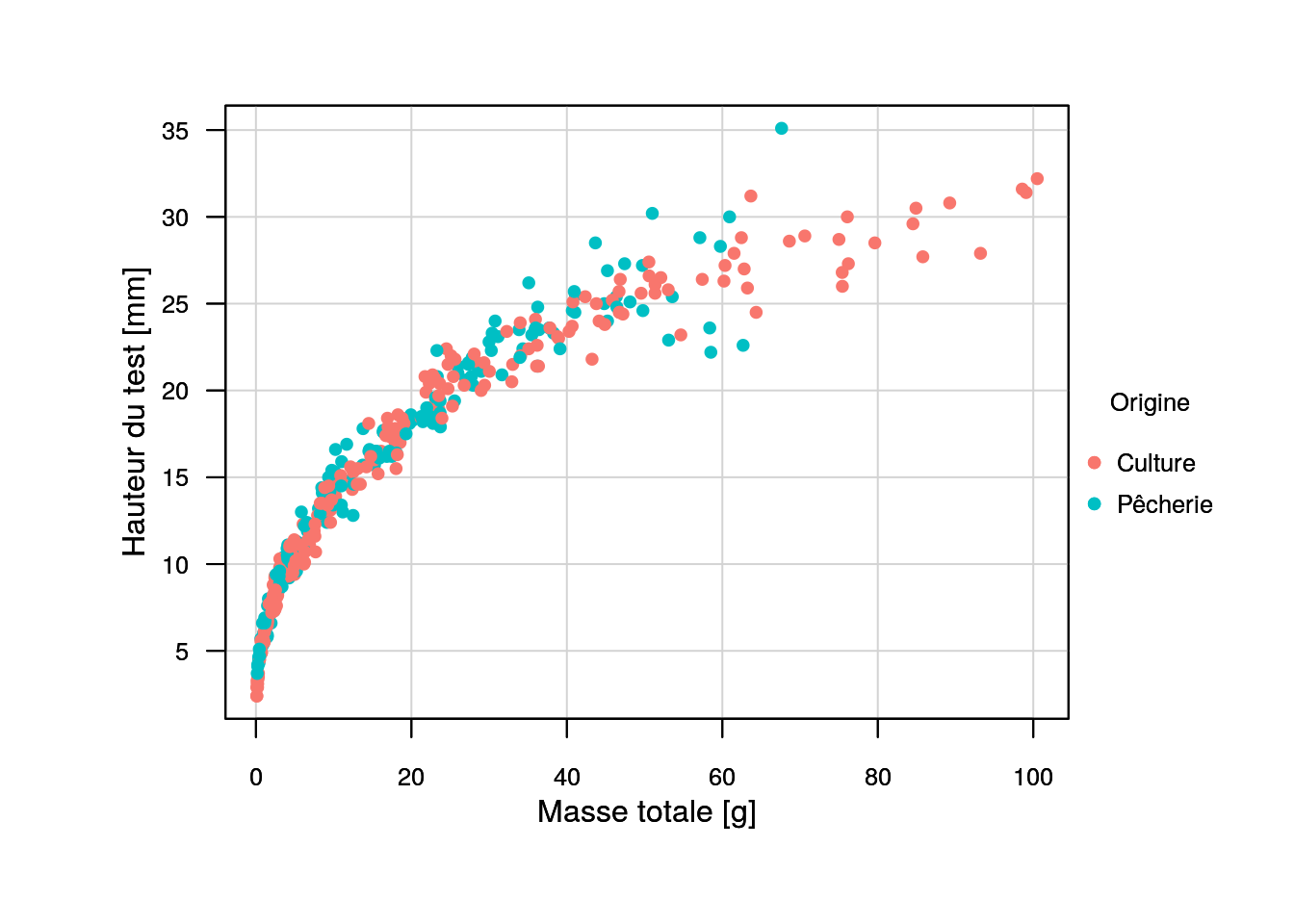
Figure 4.30: Graphique exemple réalisé avec le moteur graphique de base et la fonction chart().
fig.keep = 2 pour éviter d’imprimer la première version dans le rapport lorsqu’on utilise chart$base().
Pour l’instant, le seul avantage de chart() avec les graphiques de base est qu’il les convertit en une forme combinable avec les autres graphiques dans une figure composite (sinon, ce n’est pas possible). A part cela, il faut fournir à chart$base() tout le code nécessaire pour tracer et personnaliser le graphique. Comme on peut le voir sur cet exemple, cela demande une quantité considérable de code. C’est aussi un autre aspect pénible de ce moteur graphique : il est très flexible, mais l’interface n’est pas optimale. Pour finir, les graphiques de base ont plus de mal avec les facettes, mais il peuvent quand même générer les versions les plus simples, par exemple à l’aide de la fonction coplot() qui accepte une formule très similaire à ce qui s’utilise avec lattice :
coplot(data = urchin, height ~ weight | origin)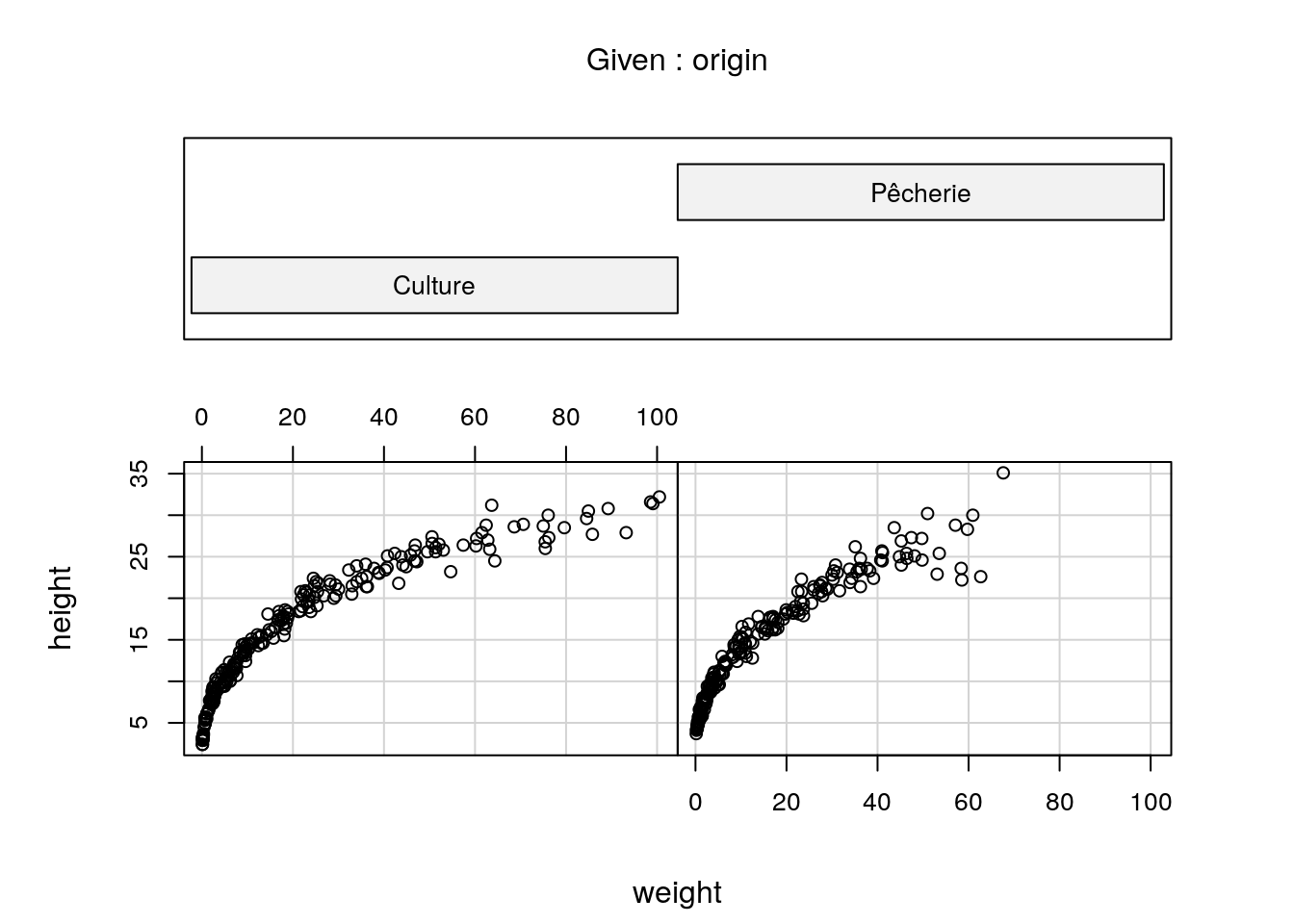
Figure 4.31: Graphique à facettes avec le moteur graphique de base.
A l’issue de cette comparaison, vous pourrez décider du moteur graphique que vous préférerez utiliser. Dans le cadre de ce cours, nous n’utiliserons en tous cas que quasi-exclusivement des graphiques ggplot2 créés à l’aide la fonction chart().
A vous de jouer
Proposez cinq graphiques inédits (qui n’ont pas été vu jusqu’ici) dans vos différents projets. Employez par exemple les liens suivants pour vous inspirer :
Terminez ce module en vérifiant que vous avez acquis l’ensemble des notions abordées.
Ouvrez RStudio dans votre SciViews Box, puis exécutez l’instruction suivante dans la fenêtre console :
BioDataScience::run("04a_test")Pour en savoir plus
Chapitre Data visualisation de R for Data Science qui utilise
ggplot().Site rassemblant des extensions pour ggplot2
Comparaison de lattice et ggplot2. Cette page fait aussi référence à un ensemble de graphiques différents générés en lattice et en ggplot2 pour comparaison (en anglais).
Divers exemples de graphiques réalisés avec le moteur de base
ggplot2 comparé aux graphiques R de base. Un point de vue différent d’un utilisateur habitué aux graphiques R de base (en anglais).
Notez que plusieurs thèmes existent dans ggplot2. Il est facile d’en changer et des les personnaliser… mais c’est toujours appréciable d’avoir un rendu impeccable dès le premier essai.↩
Etant donné l’abondante littérature écrite sur ggplot2, il est utile de pouvoir convertir des exemples ggplot2 en graphiques
chart(), si vous êtes convaincu par cette nouvelle interface.↩