3.1 Histogramme
Vous souhaitez visualiser l’étalement de vos données sur un axe (on parle de distribution4 en statistique) pour l’une des variables étudiées. L’histogramme est l’un des outils pouvant vous apporter cette information. Ce graphique représente sous forme de barres un découpage en plusieurs classes5 d’une variable numérique.
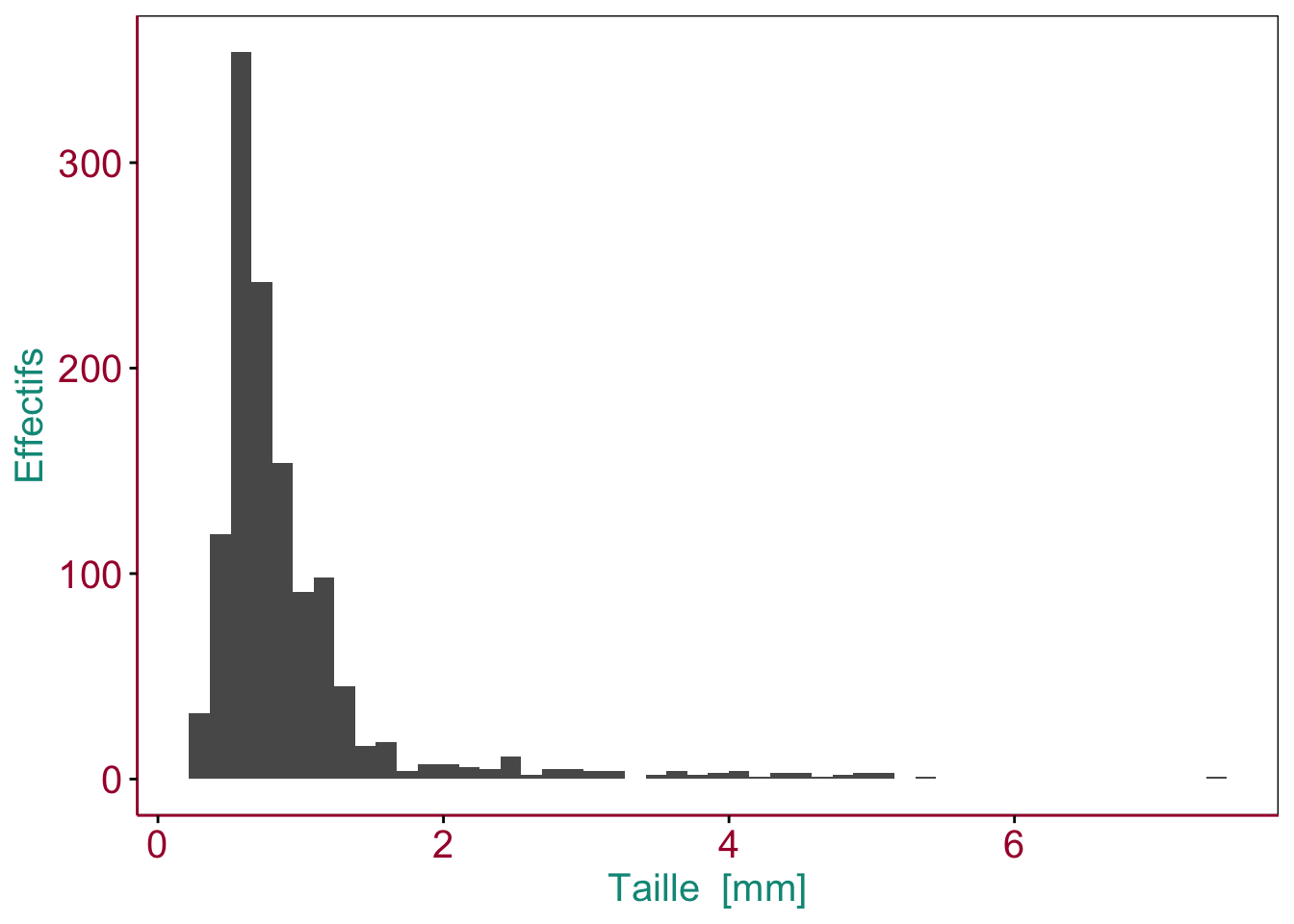
Figure 3.1: Exemple d’histogramme montrant la distribution de la taille dans un échantillon de zooplancton. Des couleurs sont utilisées pour mettre en évidence différentes parties du graphique (en pratique, les axes et leurs labels seront noirs).
Outre l’histogramme lui-même, représenté par des barres de hauteur équivalentes au nombre d’observations réalisées dans les différentes classes, les éléments suivants sont également indispensables à la compréhension du graphique (ici mis en évidence en couleur) :
- Les axes avec les graduations (en rouge). Su l’axe des abscisses, les classes de taille, et sur l’axe des ordonnées, les effectifs
- les labels des axes et l’unité (pour l’axe des abscisses uniquement ici) (en bleu)
Les instructions dans R afin de produire un histogramme à l’aide de la fonction chart() sont :
# Importation du jeu de données
(zooplankton <- read("zooplankton", package = "data.io", lang = "FR"))# # A tibble: 1,262 x 20
# ecd area perimeter feret major minor mean mode min max std_dev range
# <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# 1 0.770 0.465 4.45 1.32 1.16 0.509 0.363 0.036 0.004 0.908 0.231 0.904
# 2 0.700 0.385 2.32 0.728 0.713 0.688 0.361 0.492 0.024 0.676 0.183 0.652
# 3 0.815 0.521 4.15 1.33 1.11 0.598 0.308 0.032 0.008 0.696 0.204 0.688
# 4 0.785 0.484 4.44 1.78 1.56 0.394 0.332 0.036 0.004 0.728 0.218 0.724
# 5 0.361 0.103 1.71 0.739 0.694 0.188 0.153 0.016 0.008 0.452 0.110 0.444
# 6 0.832 0.544 5.27 1.66 1.36 0.511 0.371 0.02 0.004 0.844 0.268 0.84
# 7 1.23 1.20 15.7 3.92 1.37 1.11 0.217 0.012 0.004 0.784 0.214 0.78
# 8 0.620 0.302 3.98 1.19 1.04 0.370 0.316 0.012 0.004 0.756 0.246 0.752
# 9 1.19 1.12 15.3 3.85 1.34 1.06 0.176 0.012 0.004 0.728 0.172 0.724
# 10 1.04 0.856 7.60 1.89 1.66 0.656 0.404 0.044 0.004 0.88 0.264 0.876
# # … with 1,252 more rows, and 8 more variables: size <dbl>, aspect <dbl>,
# # elongation <dbl>, compactness <dbl>, transparency <dbl>, circularity <dbl>,
# # density <dbl>, class <fct># Réalisation du graphique
chart(data = zooplankton, ~ size) +
geom_histogram(bins = 50) +
ylab("Effectifs")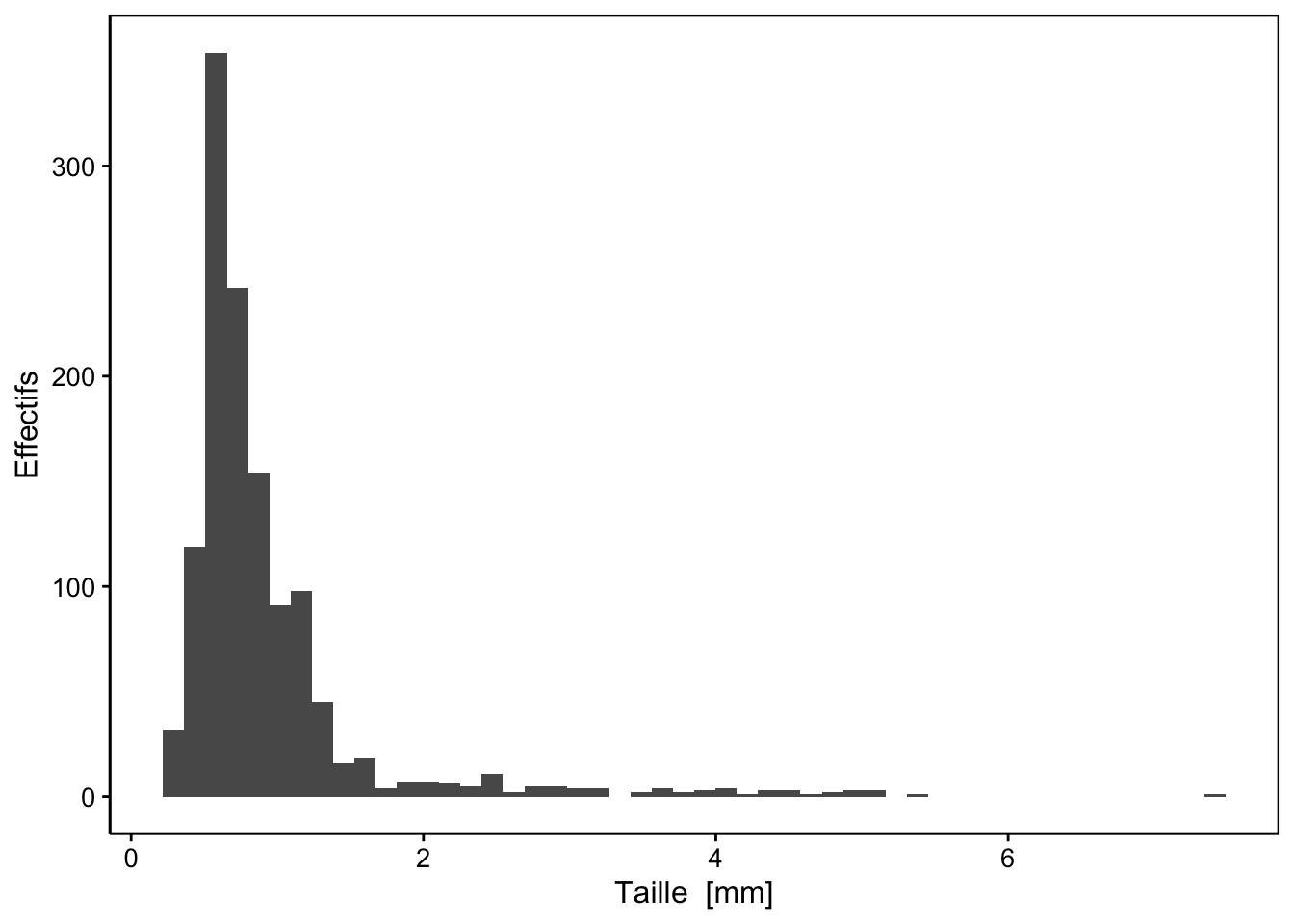
Figure 3.2: Distribution des tailles au sein d’un échantillon de zooplancton
La fonction chart() requiert comme argument le jeu de donnée (data = zooplankton), ainsi que la formule à employer dans laquelle vous avez indiqué le nom de la variable que vous voulez sur l’axe des abscisses à droite de la formule, après le tilde ~. Parmi toutes les variables du jeu de données, nous avons choisi ici de représenter size. Jusqu’ici, nous avons spécifié ce que nous voulons représenter, mais pas encore comment (sous quelle apparence), nous voulons matérialiser cela sur le graphique. Avec les graphiques de type ggplot() que nous réalisons ici à l’aide de chart(), nous ajoutons des couches au graphique à l’aide de l’opérateur +. Pour un histogramme, nous devons ajouter une couche avec la fonction geom_histogram(), tout comme pour le nuage de point, nous utilisions geom_point(). L’argument bins= dans cette fonction permet de préciser le nombre de classes souhaitées. Le découpage en classe de la variable size se fait ici automatiquement.
À vous de jouer !
Vous pouvez décrypter votre histogramme sur base des modes6 et de la symétrie7 de ces derniers. Un histogramme peut être unimodal (un seul mode), bimodal (deux modes) ou multimodal (plus de deux modes). En général, s’il y a plus d’un mode, nous pouvons suspecter que des sous-populations distinctes existent dans les données (par exemple : des différences morphométriques entre mâles et femelles pour une espèce au dimorphisme sexuel marqué).
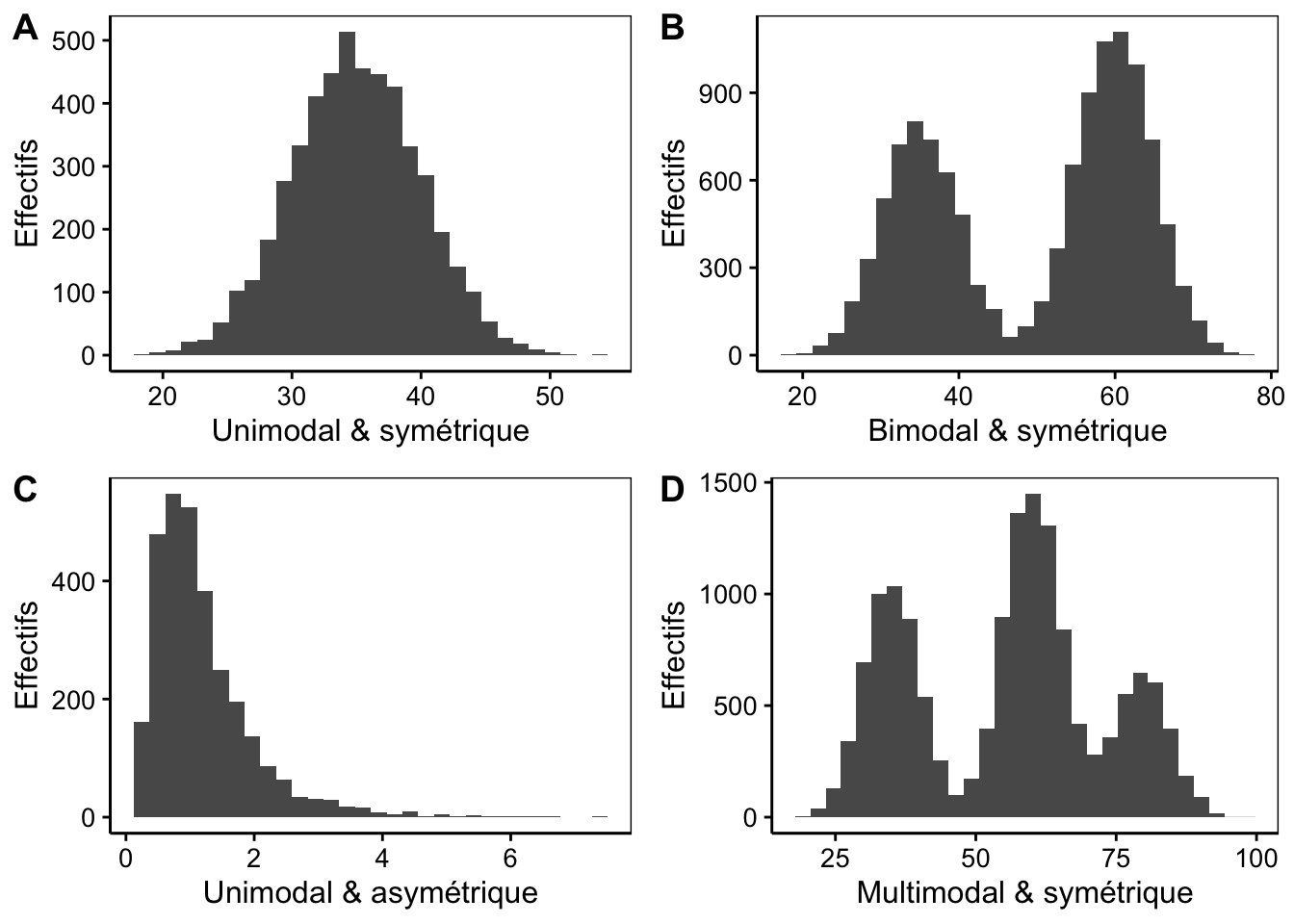
Figure 3.3: Histogrammes montrant les modes et symétries : A. histogramme unimodal et symétrique, B. histogramme bimodal et asymétrique, C. histogramme unimodal et asymétrique, D. histogramme multimodal et symétrique.
3.1.1 Nombre de classes
Vous devez être particulièrement vigilant lors de la réalisation d’un histogramme aux classes définies pour ce dernier.
# Réalisation du graphique précédent
a <- chart(data = zooplankton, ~ size) +
geom_histogram(bins = 50) +
ylab("Effectifs")
# Modification du nombre de classes
b <- chart(data = zooplankton, ~ size) +
geom_histogram(bins = 20) +
ylab("Effectifs")
c <- chart(data = zooplankton, ~ size) +
geom_histogram(bins = 10) +
ylab("Effectifs")
d <- chart(data = zooplankton, ~ size) +
geom_histogram(bins = 5) +
ylab("Effectifs")
# Assemblage des graphiques
combine_charts(list(a, b, c, d))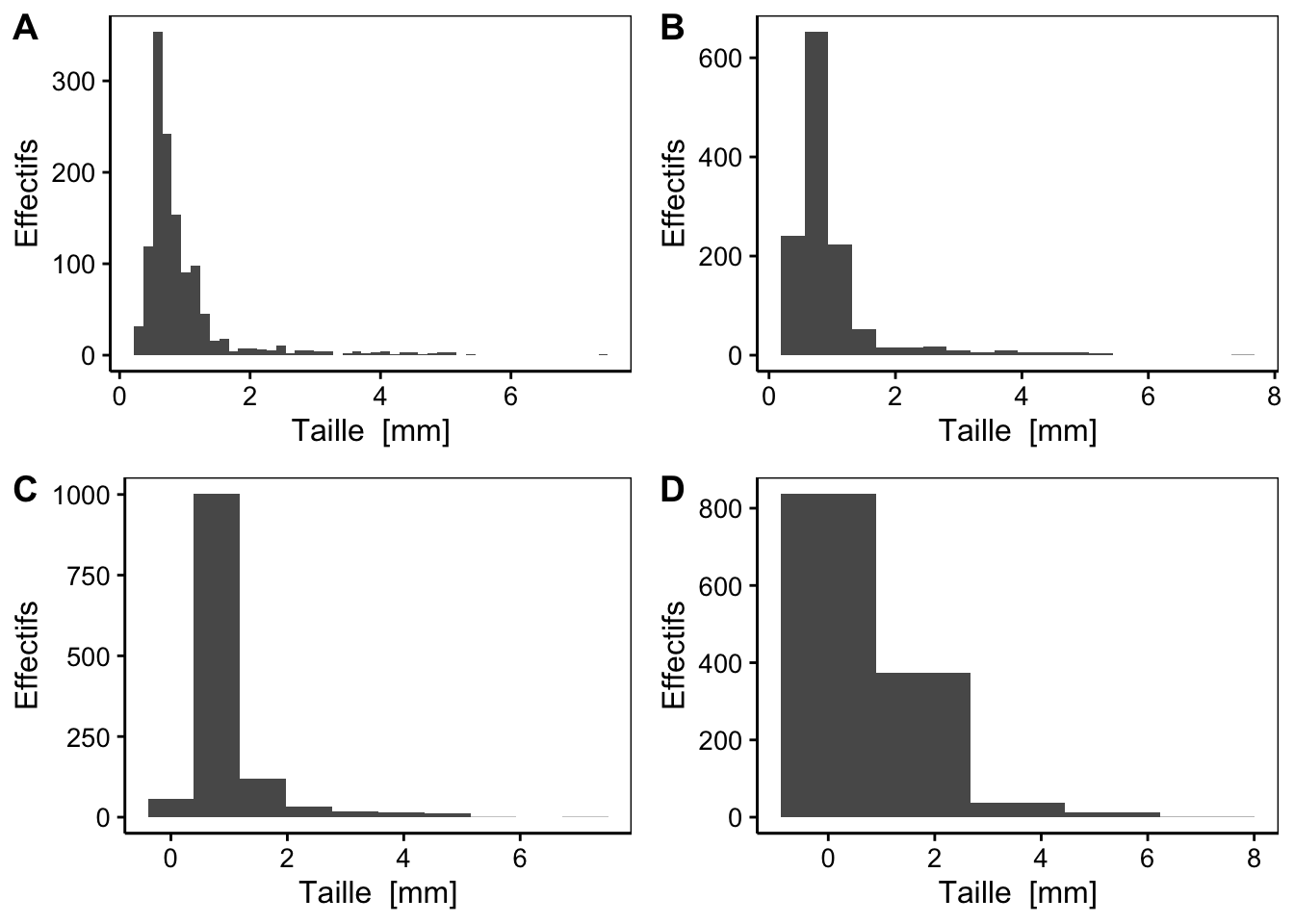
Figure 3.4: Choix des classes. A. histogramme initial montrant la répartition des tailles au sein d’organismes planctoniques. B., C., D. Même histogramme que A, mais en modifiant le nombre de classes.
Comme vous pouvez le voir à la Fig. 3.4, le changement du nombre de classes peut modifier complètement la perception des données via l’histogramme (notez l’utilisation de la fonction combine_charts() pour réaliser une figure composite, nous étudierons cette fonction plus en détail dans le prochain module). Le choix idéal est un compromis entre plus de classes (donc plus de détails), et un découpage raisonnable en fonction de la quantité de données disponibles. Si l’intervalle des classes est trop petit, l’histogramme sera illisible. Si l’intervalle des classes est trop grand, il sera impossible de visualiser correctement les différents modes. Dans la figure en exemple, les variantes A et B sont acceptables, mais les C et D manquent de détails.
À vous de jouer !
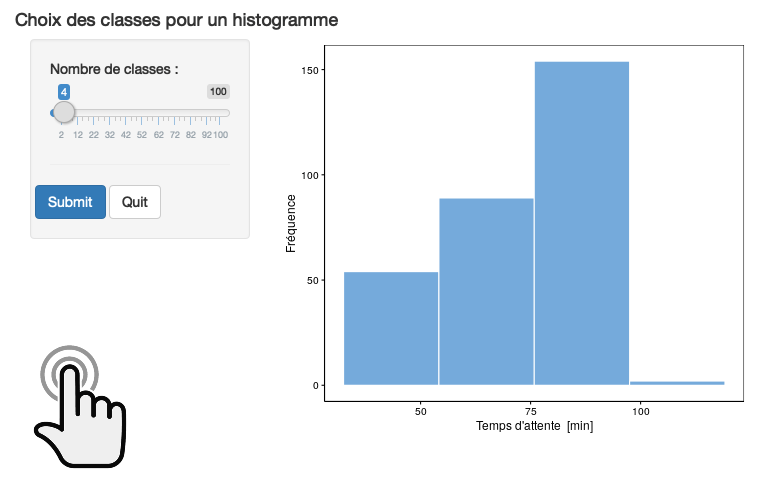 Cliquez pour lancer ou exécutez dans RStudio
Cliquez pour lancer ou exécutez dans RStudio BioDataScience1::run_app("A03Sa_histogram").
Pièges et astuces
La SciViews Box propose un snippet RStudio pour réaliser un histogramme. Il s’appelle .cuhist (pour chart -> univariate -> histogram). Entrez ce code dans une zone d’édition R et appuyez ensuite sur la tabulation, et vous verrez le code remplacé par ceci :
chart(data = DF, ~VARNUM) +
geom_histogram(binwidth = 30)L’argument binwidth= permet de préciser la largeur des classes. C’est une autre façon de spécifier le découpage en classes, mais vous pouvez naturellement le remplacer par l’argument bins= si vous préférez.
Vous avez à votre disposition un ensemble de snippets que vous pouvez retrouver dans l’aide-mémoire sur SciViews. N’oubliez pas que vous avez également à votre disposition l’aide-mémoire sur la visualisation des données (Data Visualization Cheat Sheet), via la fonction ggplot() à laquelle vous pouvez simplement substituer chart().
3.1.2 Histogramme par facteur
Lors de l’analyse de jeux de données, vous serez amené à réaliser un histogramme par facteur (c’est-à-dire, en fonction de différents niveaux d’une variable qualitative qui divise le jeu de données en sous-groupes).
Une variable qualitative ou variable facteur est une variable qui représente des catégories. Par exemple, une couleur, le genre, une classe taxonomique, … Les différentes catégories possibles pour la variable sont appelées niveaux ou modalités (levels en anglais). Pour le genre, nous auront donc deux niveaux : “homme” ou “femme”.
Les variables facteurs peuvent aussi représenter un petit nombre de classes différentes8 réalisées par découpage d’une variable numérique. Par exemple, si au lieu de reprendre la taille d’un animal, nous nous contentons de considérer s’il est “petit”, “moyen” ou “grand”. Dans ce cas, il existe un ordre logique entre les niveaux : petit < moyen < grand. La variable sera alors dite “qualitative ordonnée” et sera représentée par un objet ordered dans R. Sinon, la variable sera qualitative non ordonnée et sera un objet factor dans R.
numeric ou integer (des entiers) dans R. Attention que les variables facteur peuvent très bien être importées comme chaînes de caractères (objet character), et il faudra peut-être les convertir à l’aide des fonctions factor() ou ordered() avant de les utiliser.
Par exemple, dans un jeu de données sur des fleurs d’iris, la variable species9 représente l’espèce d’iris étudiée (trois espèces différentes : I. setosa, I. versicolor et I. virginica).
# # A tibble: 150 x 5
# sepal_length sepal_width petal_length petal_width species
# <dbl> <dbl> <dbl> <dbl> <fct>
# 1 5.1 3.5 1.4 0.2 setosa
# 2 4.9 3 1.4 0.2 setosa
# 3 4.7 3.2 1.3 0.2 setosa
# 4 4.6 3.1 1.5 0.2 setosa
# 5 5 3.6 1.4 0.2 setosa
# 6 5.4 3.9 1.7 0.4 setosa
# 7 4.6 3.4 1.4 0.3 setosa
# 8 5 3.4 1.5 0.2 setosa
# 9 4.4 2.9 1.4 0.2 setosa
# 10 4.9 3.1 1.5 0.1 setosa
# # … with 140 more rows# Réalisation de l'histogramme par facteur
chart(data = iris, ~ sepal_length %fill=% species) +
geom_histogram(bins = 25) +
ylab("Effectifs") +
scale_fill_viridis_d() # palette de couleur harmonieuse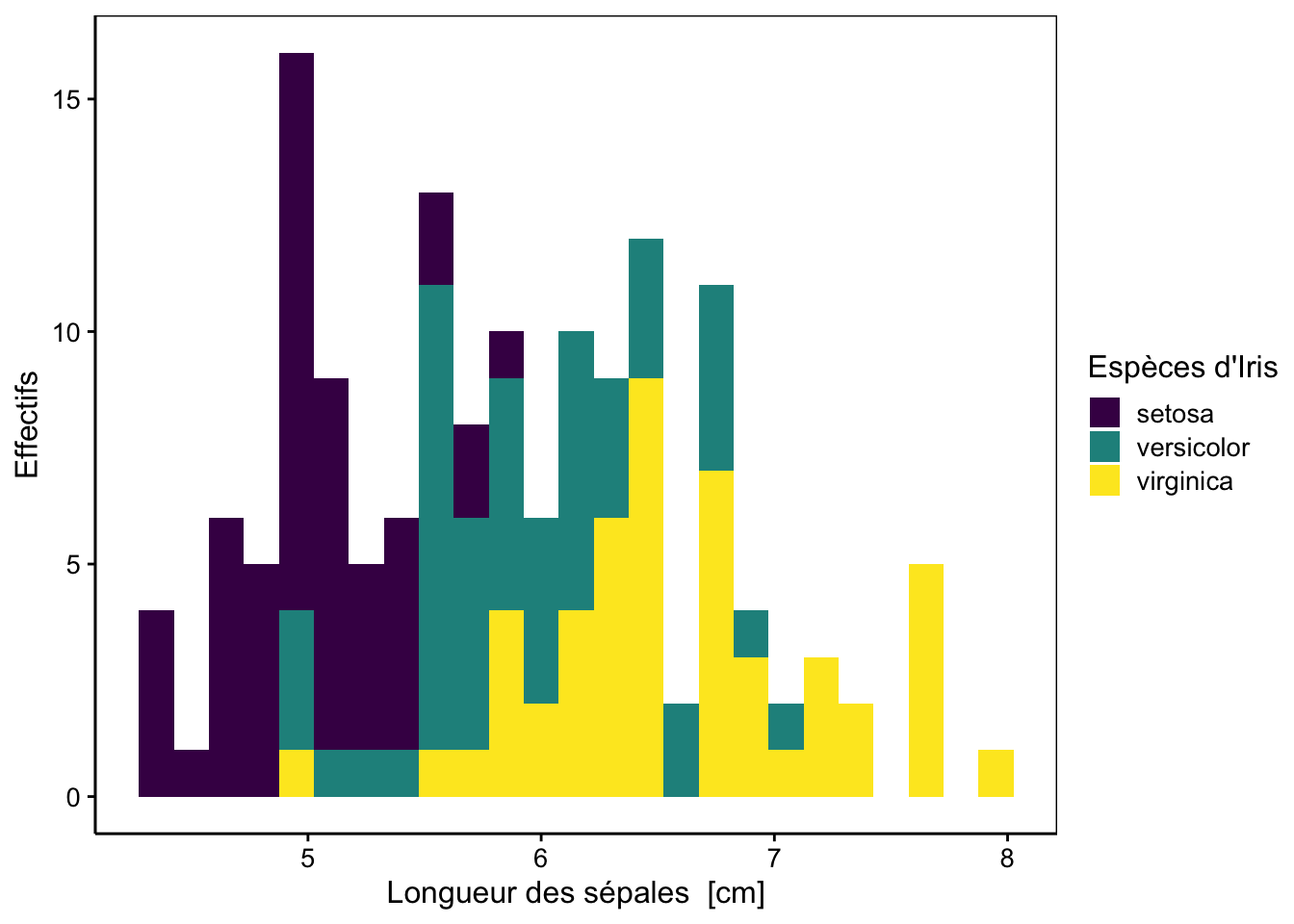
Figure 3.5: Distribution des longueurs de sépales de trois espèces d’iris.
Ici, nous avons tracé un histogramme unique, mais en prenant soin de colorier les barres en fonction de l’espèce. La formule fait toujours intervenir la variable numérique à découper en classes à la droite du tilde ~, ici sepal_length, mais nous y avons ajouté une directive supplémentaire pour indiquer que le remplissage des barres (%fill=%) doit se faire en fonction du contenu de la variable species.
Nous avons ici un bon exemple d’histogramme multimodal lié à la présence de trois sous-populations (les trois espèces différentes) au sein d’un jeu de données unique. Le rendu du graphique n’est pas optimal. Voici deux astuces pour l’améliorer. La première consiste à représenter trois histogrammes séparés, mais rassemblés dans une même figure. Pour cela, nous utilisons des facettes (facets) au lieu de l’argument %fill=%. Dans chart(), les facettes peuvent être spécifiées an utilisant l’opérateur | dans la formule.
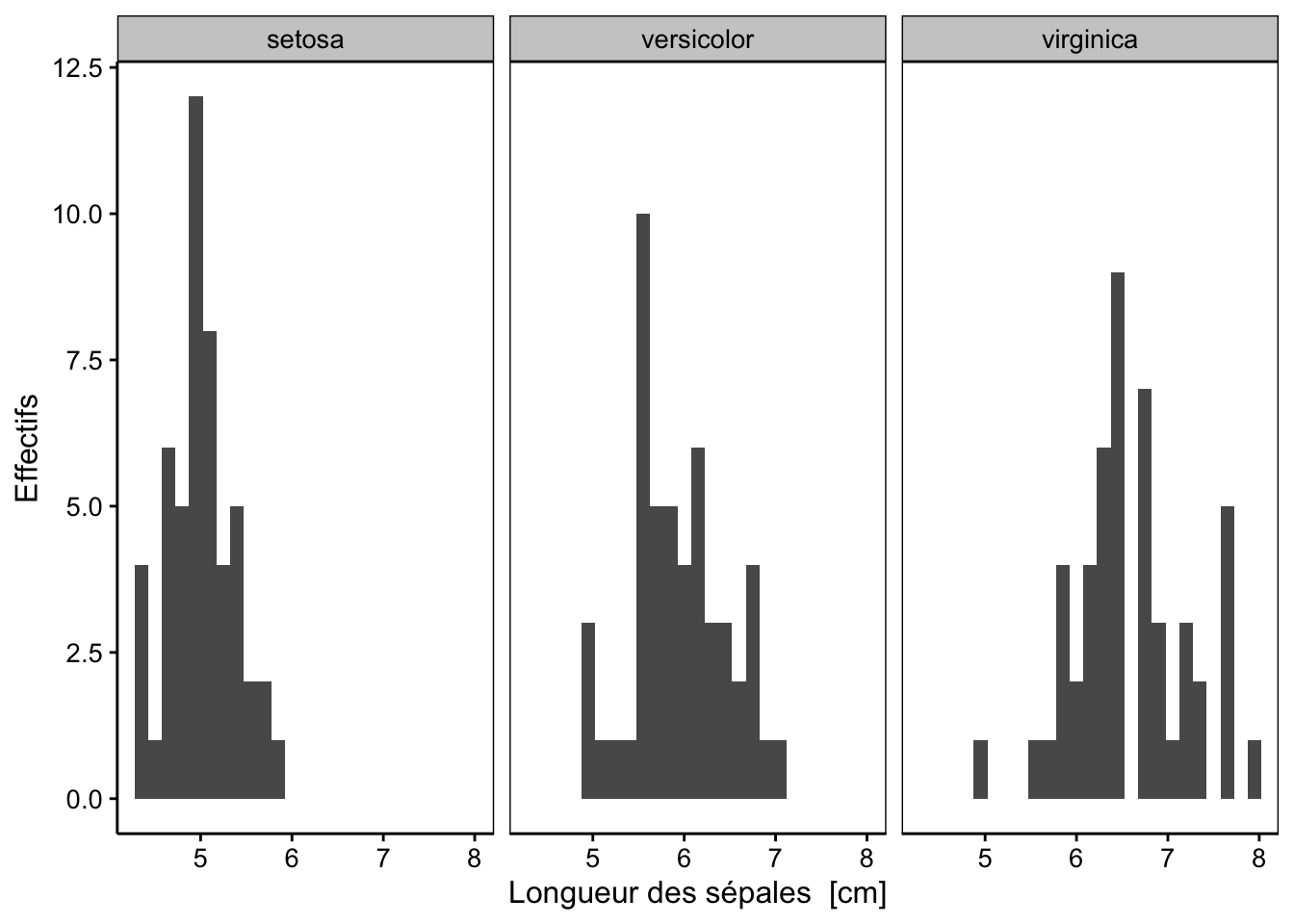
Figure 3.6: Distribution de la longueur des sépales de trois espèces d’iris (en employant les facettes pour séparer les espèces).
L’histogramme est maintenant séparé en trois en fonction des niveaux de la variable facteur species. Cela rend la lecture plus aisée. Une seconde solution combine les facettes avec | et l’argument %fill=%10. Il faut ensuite ajouter par-derrière un histogramme grisé de l’ensemble des données.
nbins <- 25
chart(data = iris, ~ sepal_length %fill=% species | species) +
# histogramme d'arrière-plan en gris ne tenant pas compte de la variable species
geom_histogram(data = select(iris, -species), fill = "grey", bins = nbins) +
geom_histogram(show.legend = FALSE, bins = nbins) +
ylab("Effectifs") +
scale_fill_viridis_d()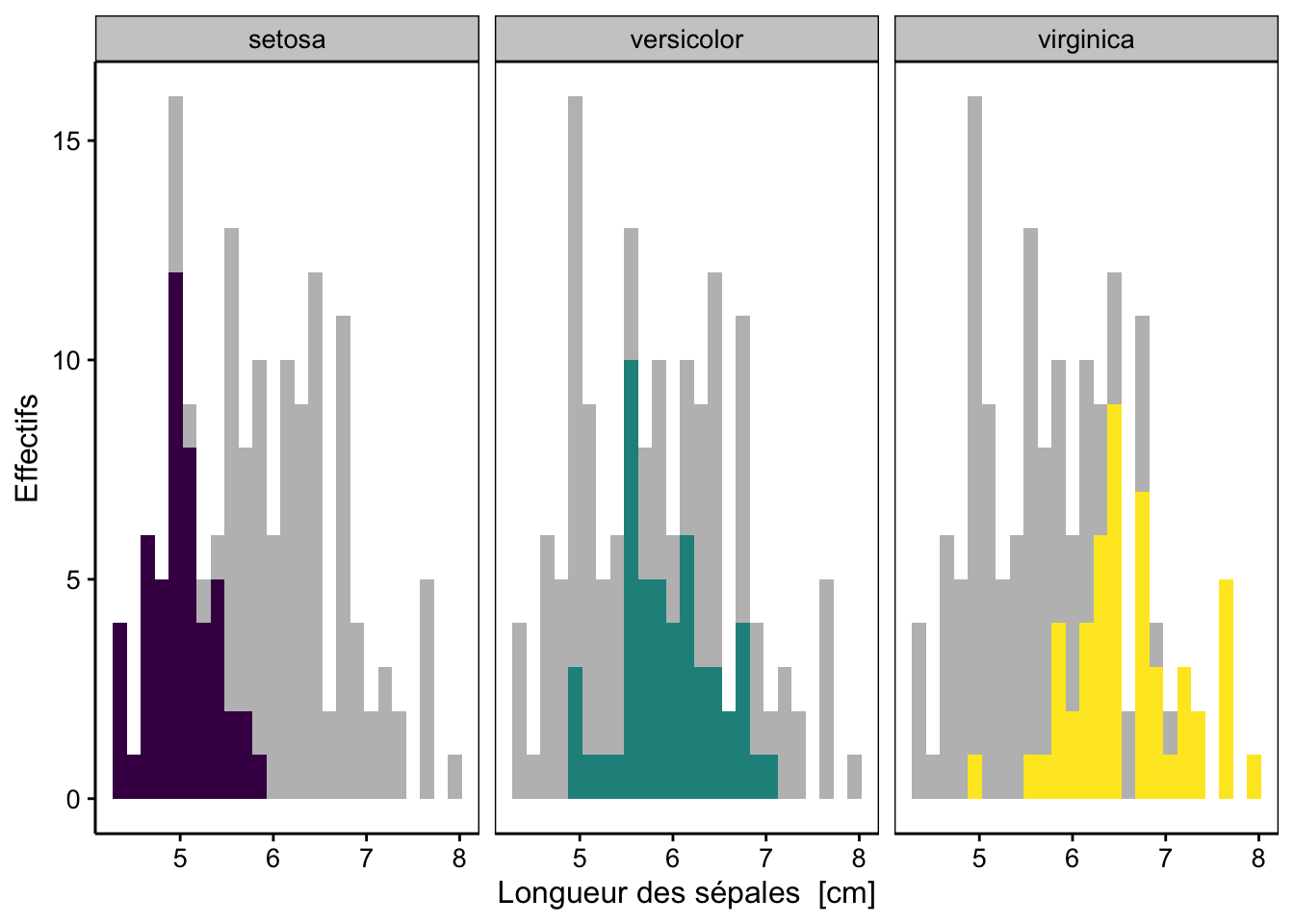
Figure 3.7: Distribution des longueurs de sépales de trois espèces d’iris (avec facettes et histogrammes complets grisés en arrière-plans).
Vous découvrez sans doute que les graphiques réalisables avec R sont modulables à souhait en ajoutant une série d’instructions successives qui créent autant de couches superposées dans le graphique. Cette approche permet de réaliser quasiment une infinité de graphiques différents en combinant seulement quelques dizaines d’instructions. Pour s’y retrouver, les fonctions qui ajoutent des couches commencent toutes par geom_, et celles qui manipulent les couleurs par scale_, par exemple. Vous découvrirez encore d’autres fonctions graphiques plus loin.
À vous de jouer !
Effectuez maintenant les exercices du tutoriel A03La_univaries (Graphiques univariés).
BioDataScience1::run("A03La_univaries")note: Ce tutoriel couvre l’ensemble de la matière de ce module. N’hésitez pas à le réaliser en parallèle de votre lecture.