3.2 Graphique de densité
L’histogramme n’est pas le seul outil à votre disposition. Vous pouvez également employer le graphique de densité qui se présente un peu comme un histogramme lissé. Le passage d’un histogramme vers un graphe de densité se base sur une estimation par noyau gaussien11
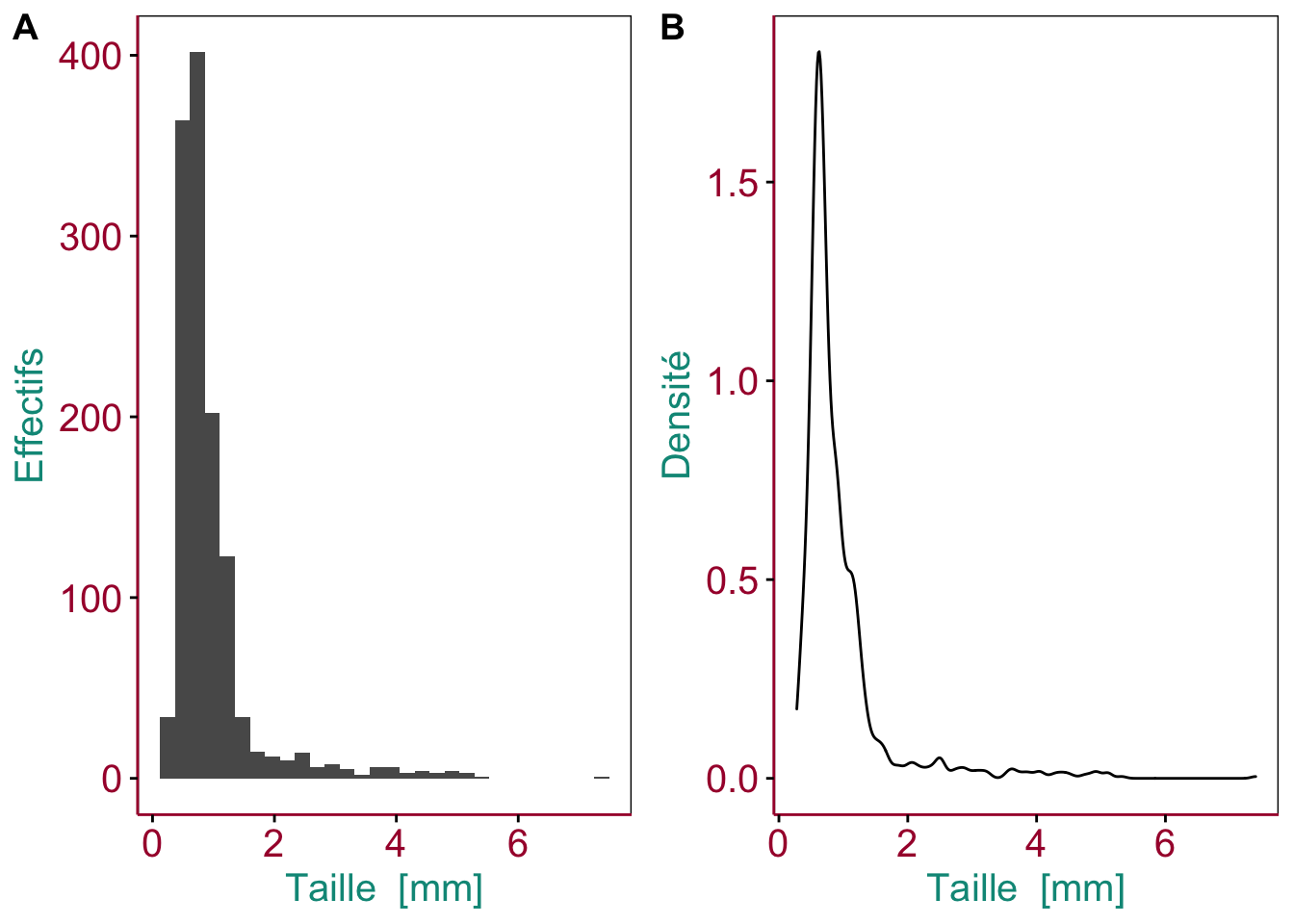
Figure 3.8: A. Histogramme et B. graphique de densité montrant la distribution de la taille de zooplancton étudié par analyse d’image. Les couleurs pour les axes et les labels servent à les mettre en évidence, mais en pratique ils seront noirs.
Comme pour les autres graphiques, veillez à soigner les indications qui permettent d’interpréter le graphique. Outre la courbe de densité, il faut :
- Les axes avec les graduations (en rouge)
- les labels des axes, et l’unité pour l’axe des abscisses (en bleu)
Les instructions en R pour produire un graphique de densité avec la fonction chart() sont :
# Importation du jeu de données
(zooplankton <- read("zooplankton", package = "data.io", lang = "FR"))# # A tibble: 1,262 x 20
# ecd area perimeter feret major minor mean mode min max std_dev range
# <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# 1 0.770 0.465 4.45 1.32 1.16 0.509 0.363 0.036 0.004 0.908 0.231 0.904
# 2 0.700 0.385 2.32 0.728 0.713 0.688 0.361 0.492 0.024 0.676 0.183 0.652
# 3 0.815 0.521 4.15 1.33 1.11 0.598 0.308 0.032 0.008 0.696 0.204 0.688
# 4 0.785 0.484 4.44 1.78 1.56 0.394 0.332 0.036 0.004 0.728 0.218 0.724
# 5 0.361 0.103 1.71 0.739 0.694 0.188 0.153 0.016 0.008 0.452 0.110 0.444
# 6 0.832 0.544 5.27 1.66 1.36 0.511 0.371 0.02 0.004 0.844 0.268 0.84
# 7 1.23 1.20 15.7 3.92 1.37 1.11 0.217 0.012 0.004 0.784 0.214 0.78
# 8 0.620 0.302 3.98 1.19 1.04 0.370 0.316 0.012 0.004 0.756 0.246 0.752
# 9 1.19 1.12 15.3 3.85 1.34 1.06 0.176 0.012 0.004 0.728 0.172 0.724
# 10 1.04 0.856 7.60 1.89 1.66 0.656 0.404 0.044 0.004 0.88 0.264 0.876
# # … with 1,252 more rows, and 8 more variables: size <dbl>, aspect <dbl>,
# # elongation <dbl>, compactness <dbl>, transparency <dbl>, circularity <dbl>,
# # density <dbl>, class <fct>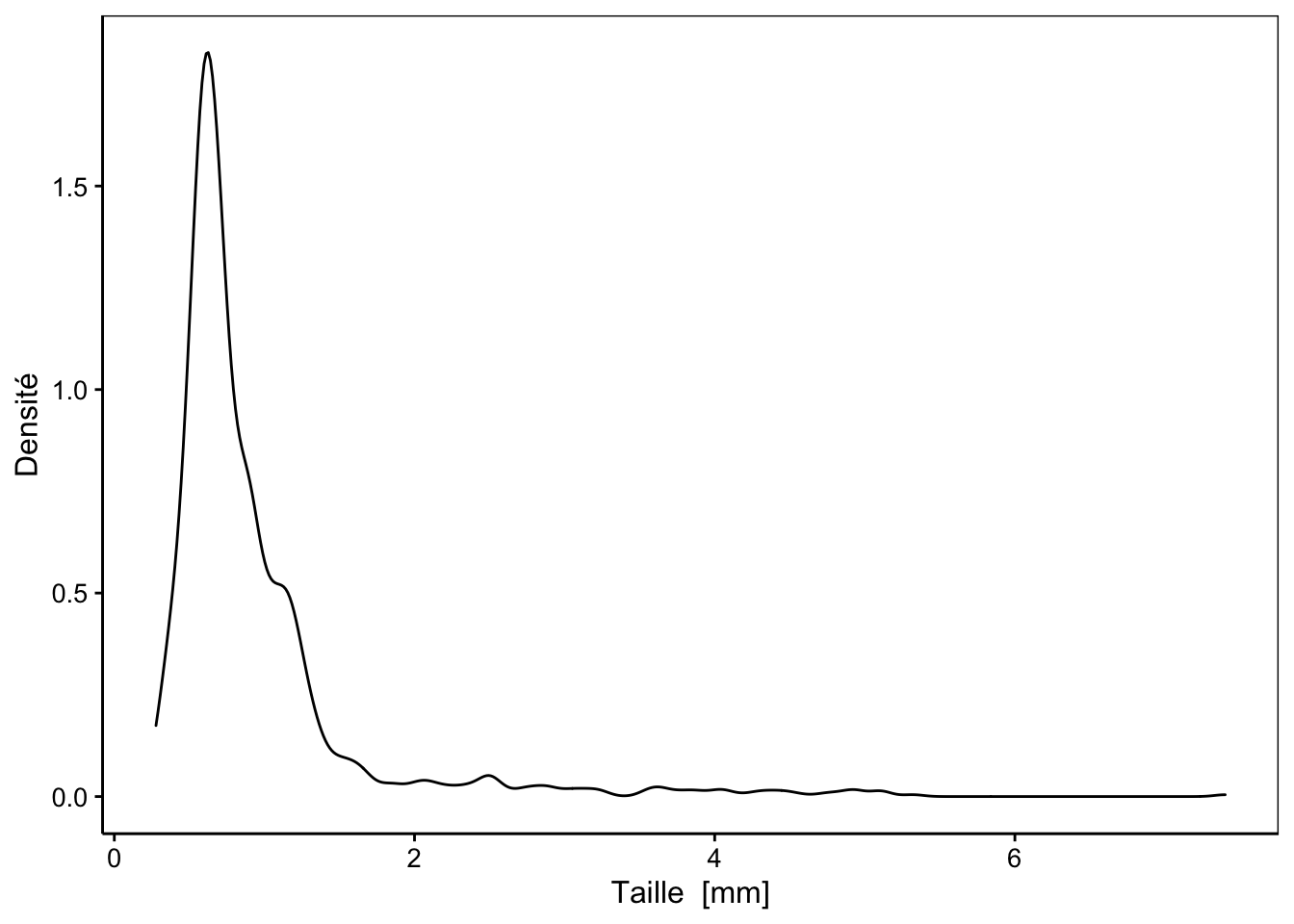
Figure 3.9: Distribution des tailles au sein de l’échantillon de zooplancton.
Ici, nous utilisons donc la fonction geom_density().
À vous de jouer !
Note : ce projet GitHub Classroom individuel couvre l’ensemble de la matière de ce module. N’hésitez pas à la réaliser en parallèle de votre lecture.
Réalisez le travail A03Ia_distributions.
Travail individuel pour les étudiants inscrits au cours de Science des Données Biologiques I : visualisation à l’UMONS à terminer avant le 2021-12-24 23:59:59.
Initiez votre projet GitHub Classroom
Voyez les explications dans le fichier README.md.