4.1 Importation des données
Il est possible d’encoder de très petits jeux de données directement dans R. La fonction dtx_rows() permet de le faire facilement. Notez que les noms des colonnes du tableau sont à rentrer sous forme de formules (~var), que chaque entrée est séparée par une virgule, et que les chaînes de caractères sont entourées de guillemets. Les espaces sont optionnels et peuvent être utilisés pour aligner les données afin que le tout soit plus lisible. Des commentaires peuvent être utilisés éventuellement en fin de ligne (un dièse # suivi du commentaire).
small_dataset <- dtx_rows(
~treatment, ~dose, ~response,
"control", 0.5, 18.35,
"control", 1.0, 26.43, # This value needs to be double-checked
"control", 2.0, 51.08,
"test" , 0.5, 10.29,
"test" , 1.0, 19.92,
"test" , 2.0, 41.06)
# Print the table
small_dataset# treatment dose response
# <char> <num> <num>
# 1: control 0.5 18.35
# 2: control 1.0 26.43
# 3: control 2.0 51.08
# 4: test 0.5 10.29
# 5: test 1.0 19.92
# 6: test 2.0 41.06Dans la plupart des cas, vous utiliserez ou collecterez des données stockées dans des formats divers : feuilles Excel, fichiers CSV (“comma-separated-values”, un format standard d’encodage d’un tableau de données sous forme textuelle), formats spécifiques à divers logiciels statistiques comme SAS, Stata, Systat… Ces données peuvent être sur un disque local ou disponibles depuis un lien URL sur le net18. De nombreuses fonctions existent dans R pour importer toutes ces données. La fonction read() du package {data.io} est l’une des plus simples et conviviales d’entre-elles. Vous l’avez déjà utilisée, mais reprenons un exemple pour en discuter les détails.
Genre | Date de naissance | Masse [kg] | Taille [cm] | Tour de poignet [mm] | Année de la mesure | Age [année] |
|---|---|---|---|---|---|---|
M | 1995-03-11 | 69.0 | 182 | 15.0 | 2 013 | 18 |
M | 1998-04-03 | 74.0 | 190 | 16.0 | 2 013 | 15 |
M | 1967-04-04 | 83.0 | 185 | 17.5 | 2 013 | 46 |
M | 1994-02-10 | 60.0 | 175 | 15.0 | 2 013 | 19 |
W | 1990-12-02 | 48.0 | 167 | 14.0 | 2 013 | 23 |
... | ... | ... | ... | ... | ... | |
M | 1998-08-12 | 80.0 | 177 | 18.0 | 2 017 | 19 |
W | 1999-08-12 | 67.3 | 173 | 15.0 | 2 017 | 18 |
M | 1968-08-18 | 84.0 | 187 | 18.0 | 2 017 | 49 |
W | 1971-08-18 | 64.9 | 166 | 15.0 | 2 017 | 46 |
M | 1999-08-12 | 68.7 | 182 | 15.0 | 2 017 | 18 |
First and last 5 rows of a total of 395 | ||||||
Le jeu de données biometry est disponible dans le package R {BioDataScience} indiqué dans l’argument package =. Dans ce cas, il ne faut pas spécifier de chemin d’accès au fichier : R sait où le trouver tout seul. Il est également spécifié ici que la langue souhaitée est le français avec l’argument lang = "fr". Le résultat de l’importation est assigné à la variable biometry (mais elle pourrait tout aussi bien porter un autre nom). Pour finir, visualisez le résultat de l’importation, ici en faisant un tableau des premières et dernières lignes avec tabularise$headtail().
Visualisez toujours votre tableau de données juste après l’importation pour vérifier que l’opération s’est bien déroulée. Vérifiez que les différentes colonnes ont été importées au bon format. Vous pouvez ouvrir l’onglet Environnement de RStudio et cliquer sur la petite icône bleue devant le nom de l’objet pour voir de quoi il est composé. En particulier,
Les données numériques sont-elle bien comprises par R comme des nombres (
<num>,<dbl>ou<int>) ?Les variables qualitatives ou semi-quantitatives sont parfois importées comme chaînes de caractères (
<chr>) et doivent éventuellement être converties en variables de type facteur à l’aide deas.factor()ou facteur ordonné avecas.ordered(), voir plus loin.
View(biometry), glimpse(biometry), str(biometry), skimr::skim(biometry).
Avant d’importer vos données dans R, vous devez vous poser les deux questions suivantes :
- Où ces données sont-elles stockées ?
Vous venez d’importer des données depuis un package R. Vous pouvez également les lire depuis un fichier sur le disque ou en utilisant une URL pour les récupérer depuis le Web. Tous ces cas sont gérés par read() qui unifie donc de manière simple vos accès aux données.
- Quel est le format de vos données ?
Souvent ce format est renseigné par l’extension du fichier. Par exemple .xlsx pour un Microsoft Excel ou .csv pour du “comma-separated-value”. Attention ! L’extension du fichier est cachée sous Windows, et parfois sous macOS. Visualisez vos fichiers dans l’onglet Files dans RStudio pour voir leurs noms complets avec les extensions. Pour l’instant, read() supporte 32 formats de fichiers différents, mais cette liste est amenée à s’agrandir à l’avenir. Pour découvrir les formats supportés, et les fonctions d’importation spécifiques appelées à chaque fois, utilisez :
# # A tibble: 32 × 5
# type read_fun read_header write_fun comment
# <chr> <chr> <chr> <chr> <chr>
# 1 csv readr::read_csv data.io::hread_text readr::write_csv comma …
# 2 csv2 readr::read_csv2 data.io::hread_text <NA> semico…
# 3 xlcsv readr::read_csv data.io::hread_text readr::write_excel_csv write …
# 4 tsv readr::read_tsv data.io::hread_text readr::write_tsv tab se…
# 5 fwf readr::read_fwf data.io::hread_text <NA> fixed …
# 6 log readr::read_log <NA> <NA> standa…
# 7 rds readr::read_rds <NA> readr::write_rds R data…
# 8 txt readr::read_file <NA> readr::write_file text f…
# 9 raw readr::read_file_raw <NA> <NA> binary…
# 10 ssv readr::read_table data.io::hread_text <NA> space …
# # ℹ 22 more rowsPar la suite, vous allez apprendre à importer vos données depuis différentes sources.
4.1.1 Données sur le disque
Lorsque l’extension du fichier reflète le format des données, il vous suffit juste d’indiquer le chemin d’accès au fichier dans read(), en utilisant préférentiellement un chemin relatif. La plupart du temps, cela suffit pour importer correctement les données.
Très important : le chemin d’accès à votre fichier
peut s’écrire de manière absolue (chemin partant de la racine d’un
disque sous Windows ou de la hiérarchie de dossiers sous macOS ou Linux)
ou bien de manière relative (par rapport au dossier courant, la plupart
du temps, le dossier du projet dans RStudio). Vous devez autant
que possible employer des chemins relatifs pour que votre
projet soit portable. Si vous avez du mal à déterminer
le chemin relatif par rapport à vos données, le snippet
filerelchoose vous sera très utile :
-
Assurez-vous que le chemin actif dans l’onglet Console est (A) le dossier racine de votre projet RStudio, ou si vous travaillez dans un fichier Quarto ou R Markdown, (B) le même que le répertoire contenant le fichier édité. Pour cela, utilisez l’entrée de menu RStudio
Session -> Définir le répertoire de travail -> Vers le répertoire du projet(situation A) ouSession -> Définir le répertoire de travail -> Vers l’emplacement du fichier source(situation B). -
Utilisez le snippet
filerelchooseque vous activez dans une zone de code R (dans un script R, ou à l’intérieur d’un chunk dans un document Quarto/R Markdown). Entrezfile, attendez que le menu contextuel de complétion apparaisse, sélectionnezfilerelchoosedans la liste et tapezEntrée. Une boite de dialogue de sélection de fichier apparaît. Sélectionnez le fichier qui vous intéresse et …fileest remplacé par le chemin relatif vers votre fichier dans l’éditeur.
Les explications détaillées concernant l’organisation de vos projets dans RStudio pour qu’ils soient portables, ainsi que la gestion des chemins d’accès aux fichiers et les chemins relatifs sont détaillés dans l’annexe B, à la section B.1.1. C’est le moment de vérifier que vous avez bien compris et assimilé son contenu.
Pièges et astuces
Si l’extension est incorrecte, vous pouvez forcer un format de fichier particulier à l’importation en l’indiquant dans l’appel à
read()commeread$<ext>(). Par exemple, pour forcer l’importation d’un fichier de type “comma-separated-values” pour un fichier qui se nommeraitmy_data.txtet qui se situe dans le répertoire courant, vous écrirezread$csv("my_data.txt").Si les données ne sont pas importées correctement, cela signifie que les arguments d’importation par défaut ne sont pas adaptés. Les arguments à spécifier sont différents d’un format à l’autre. Voyez d’abord la fonction appelée en interne par
read()dans le tableau obtenu grâce àgetOption("read_write"). Par exemple, pour un fichierxlsx, il s’agit de la fonctionreadxl::read_excel()qui est utilisée. Ensuite, voyez l’aide de cette dernière fonction pour en découvrir les différents arguments (?readxl::read_excel). Là, vous pourrez découvrir l’argumentsheet =qui indique la feuille à importer depuis le fichier (première feuille par défaut), ourange =qui indique la plage de données dans la feuille à utiliser (par défaut, depuis la cellule A1 en haut à gauche jusqu’à la fin du tableau). Donc, si votre fichiermy_data.xlsxcontient les feuillessheet1,sheet2etsheet3, et que les données qui vous intéressent sont dans la plageC5:E34desheet2, vous pourrez écrire :read("my_data.xlsx", sheet = "sheet2", range = "C5:E34").
4.1.2 Données depuis Internet
Il existe différents logiciels qui permettent d’éditer des jeux de données en ligne et de les partager sur le Net. Google Sheets est l’un d’entre eux, tout comme Excel Online. Des stockages spécifiques pour les données scientifiques existent aussi comme Figshare ou Zenodo. Ces sites permettent de partager facilement des jeux de données sur Internet.
La science est de plus en plus ouverte, et les pratiques de données ouvertes (Open Data) de plus en plus fréquentes et même imposées par des programmes de recherche comme les programmes européens ou le FNRS en Belgique. Vous serez donc certainement amenés à accéder à des données depuis des dépôts spécialisés sur Internet.
Concentrez-vous sur les outils spécifiques à la gestion de ce type de données. il s’agit, en effet, d’une compétence clé qu’un bon scientifique des données se doit de maîtriser parfaitement. En recherchant à chaque fois la meilleure façon d’accéder à des données sur Internet, vous développerez cette compétence progressivement par la pratique… et vous pourrez faire valoir un atout encore rare mais apprécié lors d’un entretien d’embauche plus tard.Voici un exemple de feuille de données Google Sheets : https://docs.google.com/spreadsheets/d/1iEuGrMk4IcCkq7gMNzy04DkSaPeWH35Psb0E56KEQMw. Il est possible d’importer ce genre de données directement dans R, mais il faut d’abord déterminer l’URL à utiliser pour obtenir les données dans un format reconnu. Dans le cas de Google Sheets, il suffit d’indiquer que l’on souhaite exporter les données au format CSV en rajoutant /export?format=csv à la fin de l’URL.
Cette URL est très longue. Elle est peu pratique et par ailleurs, elle a toujours la même structure : "https://docs.google.com/spreadsheets/d/{id}/export?format=csv" avec {id} qui est l’identifiant unique de la feuille Google Sheets (ici 1iEuGrMk4IcCkq7gMNzy04DkSaPeWH35Psb0E56KEQMw). Vous pouvez indiquer explicitement ceci dans votre code et profiter des capacités de remplacement de texte dans des chaînes de caractères de la fonction glue::glue() pour effectuer un travail impeccable.
googlesheets_as_csv <- "https://docs.google.com/spreadsheets/d/{id}/export?format=csv"
coral_data_id <- "1iEuGrMk4IcCkq7gMNzy04DkSaPeWH35Psb0E56KEQMw"
(coral_url <- glue::glue(googlesheets_as_csv, id = coral_data_id))# https://docs.google.com/spreadsheets/d/1iEuGrMk4IcCkq7gMNzy04DkSaPeWH35Psb0E56KEQMw/export?format=csvVous n’aurez alors plus qu’à lire les données depuis cette URL. N’oubliez pas non plus de spécifier à read() que les données sont à lire au format CSV en utilisant read$csv() :
# Rows: 98 Columns: 9
# ── Column specification ────────────────────────────────────────────────────────
# Delimiter: ","
# chr (2): localisation, species
# dbl (6): id, salinity, temperature, time, gain, gain_std
# dttm (1): date
#
# ℹ Use `spec()` to retrieve the full column specification for this data.
# ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.localisation | species | id | salinity | temperature | date | time | gain | gain_std |
|---|---|---|---|---|---|---|---|---|
A0 | s.hystrix | 1 | 34.7 | 24.5 | 2018-04-24 09:10:00 | 7 | 0.08 | 0.05897017 |
A0 | s.hystrix | 2 | 34.7 | 24.5 | 2018-04-24 09:10:00 | 7 | 0.06 | 0.03669477 |
A0 | s.hystrix | 3 | 34.7 | 24.5 | 2018-04-24 09:10:00 | 7 | 0.03 | 0.01904207 |
A0 | s.hystrix | 4 | 34.7 | 24.5 | 2018-04-24 09:10:00 | 7 | 0.11 | 0.04722930 |
A0 | s.hystrix | 5 | 34.7 | 24.5 | 2018-04-24 09:10:00 | 7 | 0.12 | 0.06810833 |
... | ... | ... | ... | ... | ... | ... | ... | |
B0 | s.hystrix | 96 | 35.0 | 25.2 | 2018-03-19 13:03:00 | 7 | 0.07 | 0.05942559 |
B0 | s.hystrix | 97 | 35.0 | 25.2 | 2018-03-19 13:03:00 | 7 | 0.05 | 0.04447699 |
B0 | s.hystrix | 98 | 35.0 | 25.2 | 2018-03-19 13:03:00 | 7 | 0.09 | 0.05969835 |
B0 | s.hystrix | 99 | 35.0 | 25.2 | 2018-03-19 13:03:00 | 7 | 0.06 | 0.05176493 |
B0 | s.hystrix | 100 | 35.0 | 25.2 | 2018-03-19 13:03:00 | 7 | 0.10 | 0.07131212 |
First and last 5 rows of a total of 98 | ||||||||
Lorsque vous travaillez sur des données issues d’une source externe, et donc susceptibles d’être modifiées ou même pire, de disparaître. Il est préférable d’enregistrer une copie locale de ces données dans votre projet (dans le sous-dossier data de préférence). Si vous voulez enregistrer une copie locale du fichier non modifié téléchargé depuis Internet, vous l’indiquez dans l’argument cache_file = de read() (pensez à utiliser autant que possible un chemin relatif). Ainsi, le code suivant ne téléchargera le fichier depuis Internet qu’une seule fois et en sauvera automatiquement une copie dans data/coral.csv. Ensuite, les exécutions suivantes du même code liront les données depuis la version du jeu de données sauvé en local dans data/coral.csv.
Faites bien attention que le dossier dans lequel vous souhaitez stocker une copie de vos données doit être présent sur votre disque. Dans le cas contraire, un message d’erreur sera généré et vous ne pourrez pas importer les données.
Vous pouvez aussi vous passer de cette fonctionnalité, mais importer vos données dans un script à part qui remanie aussi ces données à la suite. Vous pouvez alors terminer ce script en écrivant les données remaniées dans un dossier local. Si vous travaillez exclusivement avec R, l’un des meilleurs formats est RDS, un format natif qui conservera toutes les caractéristiques de votre objet, y compris sa classe, et d’éventuels attributs19. Par défaut, les données seront stockées non compressées, mais vous pourrez aussi décider de compresser avec les algorithmes "gz" (plus rapide et répandu), "bz2" (intermédiaire), ou "xz" (le plus efficace en taux de compression, mais aussi le plus lent et gourmand en ressources CPU). Par exemple, pour enregistrer les données avec compression "gz", dans le sous-dossier data de votre projet, vous écrirez (le dossier data doit être préexistant) :
Cette instruction est valable si elle est exécutée depuis un script R dans la fenêtre Console où le répertoire actif est le dossier racine du projet. Pour une instruction dans un document Quarto ou R Markdown présent dans un sous-dossier du projet, vous écrirez file = "../data/coral.rds". Dans les deux cas, vous pouvez aussi utiliser file = here:here("data", "coral.rds") qui fonctionnera dans tous les cas de figure (conseillé, donc). Cette forme est également utilisable pour cache_file = dans read(). Ensuite, vous pourrez simplement charger ces données plus loin depuis la version locale dans votre Quarto ou R Markdown comme ceci (cas d’un document Quarto dans un sous-dossier du projet, par exemple docs) :
Attention, ne supprimez jamais l’instruction permettant de retrouver vos données sur Internet sous prétexte que vous avez maintenant une copie locale à disposition. C’est le lien, le fil conducteur vers les données originales. Vous pouvez soit mettre l’instruction en commentaire en ajoutant un dièse devant, soit soustraire le chunk de l’évaluation en indiquant eval=FALSE dans son entête. Faites-en de même avec l’instruction write(). Ainsi, le traitement de vos données commencera à l’instruction read() et vous partirez de la copie locale. Si jamais vous voulez effectuer une mise à jour depuis la source initiale, il sera toujours possible de dé-commenter les instructions, ou de passer le chunk à eval=TRUE temporairement (ou encore plus simplement, forcez l’exécution du chunk dans l’éditeur en cliquant sur la petite flèche verte en haut à gauche de ce chunk).
Pièges et astuces
Comme il s’agit seulement d’une copie des données originelles, vous pouvez choisir de ne pas inclure le fichier
.rdsdans le système de gestion de version de Git. Il suffit d’ajouter une entrée.rdsdans le fichier.gitignoreà la racine de votre dépôt, et tous les fichiers avec cette extension seront ignorés. Notez toutefois que, si vous partagez votre projet sur GitHub, les données locales n’y apparaîtront pas non plus. D’une part, cela décharge le système de gestion de version, et d’autre part, les gros fichiers de données n’ont pas vraiment leur place dans GitHub. Cependant, soyez conscient que quelqu’un qui réalise un clone ou un fork de votre dépôt devra d’abord réimporter lui aussi localement les données avant de pouvoir travailler, ce qui implique de bien comprendre le mécanisme que vous avez mis en place. Documentez-le correctement, avec une note explicite dans le fichierREADME.md, par exemple.Les données originales ne sont peut-être pas présentées de la façon qui vous convient. Cela peut nécessiter un travail important de préparation du tableau de données. Au fur et à mesure que le ou les chunks d’importation/préparation des données augmentent en taille, ils deviennent de plus en plus gênants dans un document consacré à l’analyse de ces données. Si c’est le cas, vous avez deux options possibles :
- Séparer votre Quarto/R Markdown en deux. Un premier document dédié à l’importation/préparation des données et un second qui se concentre sur l’analyse. Une bonne pratique consiste à numéroter les fichiers en tête pour qu’ils apparaissent par ordre logique lorsqu’ils sont listés par ordre alphabétique (
01import.qmd,02analysis.qmd). - Effectuer le travail d’importation/préparation du tableau de données dans un script R. Dans le R Markdown, vous pouvez ajouter l’instruction (commentée ou placée dans un chunk
eval=FALSE) pour “sourcer” ce script R afin de réimporter/retraiter vos données (ici, cas d’un fichier Quarto qui se situerait dans un sous-dossier docs) :
Si le travail de préparation des données est lourd (et donc, prends beaucoup de temps) il peut être avantageux d’enregistrer localement la version nettoyée de vos données plutôt que la version originale. Mais alors, indiquez-le explicitement.
- Séparer votre Quarto/R Markdown en deux. Un premier document dédié à l’importation/préparation des données et un second qui se concentre sur l’analyse. Une bonne pratique consiste à numéroter les fichiers en tête pour qu’ils apparaissent par ordre logique lorsqu’ils sont listés par ordre alphabétique (
Faites toujours la distinction entre données brutes et données nettoyées. Ne les mélangez jamais et documentez toujours de manière reproductible le processus qui mène des unes aux autres ! C’est tout aussi important que de garder un lien vers la source originale des données dans votre code et d’utiliser toujours des chemins relatifs vers vos fichiers pour une analyse portable et reproductible.
Nous vous conseillons de placer la version brute des données dans le
sous-dossier data/raw et la version nettoyée dans le
sous-dossier data de votre projet RStudio. Ainsi, la
structure des dossiers est elle-même très suggestive.
4.1.3 Données depuis un package
Les packages R comme {data.io}, {chart} ou encore {svFlow}, fournissent une série de fonctions supplémentaires. Certains d’entre eux proposent également des jeux de données. Ici aussi, read() permet de les récupérer, même si c’est la fonction data() qui est souvent utilisée à cet effet dans R. Comparons read() et data() dans le cas des données issues de packages R. Avec data(), vous n’assignez pas le jeu de données à un nom. Ce nom vous est imposé comme le nom initial du jeu de données :
Le jeu de données urchin_bio n’est pas véritablement chargé dans l’environnement utilisateur avec data(). Seulement une “promesse” de chargement (Promise) est enregistrée. Voyez dans l’onglet Environnement ce qui apparaît. Ce n’est qu’à la première utilisation du jeu de données que le tableau est véritablement chargé. Par exemple :
# origin diameter1 diameter2 height buoyant_weight weight solid_parts
# 1 Fishery 9.9 10.2 5.0 NA 0.5215 0.4777
# 2 Fishery 10.5 10.6 5.7 NA 0.6418 0.5891
# 3 Fishery 10.8 10.8 5.2 NA 0.7336 0.6770
# 4 Fishery 9.6 9.3 4.6 NA 0.3697 0.3438
# 5 Fishery 10.4 10.7 4.8 NA 0.6097 0.5587
# 6 Fishery 10.5 11.1 5.0 NA 0.6096 0.5509
# integuments dry_integuments digestive_tract dry_digestive_tract gonads
# 1 0.3658 NA 0.0525 0.0079 0
# 2 0.4447 NA 0.0482 0.0090 0
# 3 0.5326 NA 0.0758 0.0134 0
# 4 0.2661 NA 0.0442 0.0064 0
# 5 0.4058 NA 0.0743 0.0117 0
# 6 0.4269 NA 0.0492 0.0097 0
# dry_gonads skeleton lantern test spines maturity sex
# 1 0 0.1793 0.0211 0.0587 0.0995 0 <NA>
# 2 0 0.1880 0.0205 0.0622 0.1053 0 <NA>
# 3 0 0.2354 0.0254 0.0836 0.1263 0 <NA>
# 4 0 0.0630 0.0167 0.0180 0.0283 0 <NA>
# 5 0 NA NA NA NA 0 <NA>
# 6 0 NA NA NA NA 0 <NA>Regardez à nouveau dans l’onglet Environnement. Ce coup-ci urchin_bio apparaît bien dans la section Data et l’icône en forme de petit tableau à la droite qui permet de le visualiser est enfin accessible.
La fonction read() permet de choisir librement le nom que nous souhaitons donner à notre jeu de données. Si nous voulons l’appeler urchin au lieu de urchin_bio, pas de problèmes. De plus, il est directement chargé et accessible dans l’onglet Environnement (en effet, si on utilise une instruction qui charge un jeu de données, c’est très vraisemblablement parce que l’on souhaite ensuite le manipuler depuis R, non ?)
Nous avons déjà vu que read() donne accès également dans certains cas à des métadonnées (par exemple le label et les unités des jeux de données) dans différentes langues, ce que ne permet pas data(). Enfin, la syntaxe et la fonction utilisée sont pratiquement identiques pour charger des données depuis un fichier, depuis Internet ou depuis un package avec read(). C’est logique et facile à retenir. data() ne permet que de récupérer des données liées à un package R, et c’est tout ! Pour toutes ces raisons, nous préférons utiliser ici read() à data().
4.1.3.1 Langue du jeu de données
La fonction read() est également capable de lire un fichier annexe permettant de rajouter des métadonnées (données complémentaires) à notre tableau, comme les labels et les unités des variables en différentes langues. Lorsque le jeu de données est importé avec la fonction data(), ces métadonnées ne sont pas employées.
data("urchin_bio", package = "data.io")
# Visualisation des données
chart(urchin_bio, height ~ weight %col=% origin) +
geom_point()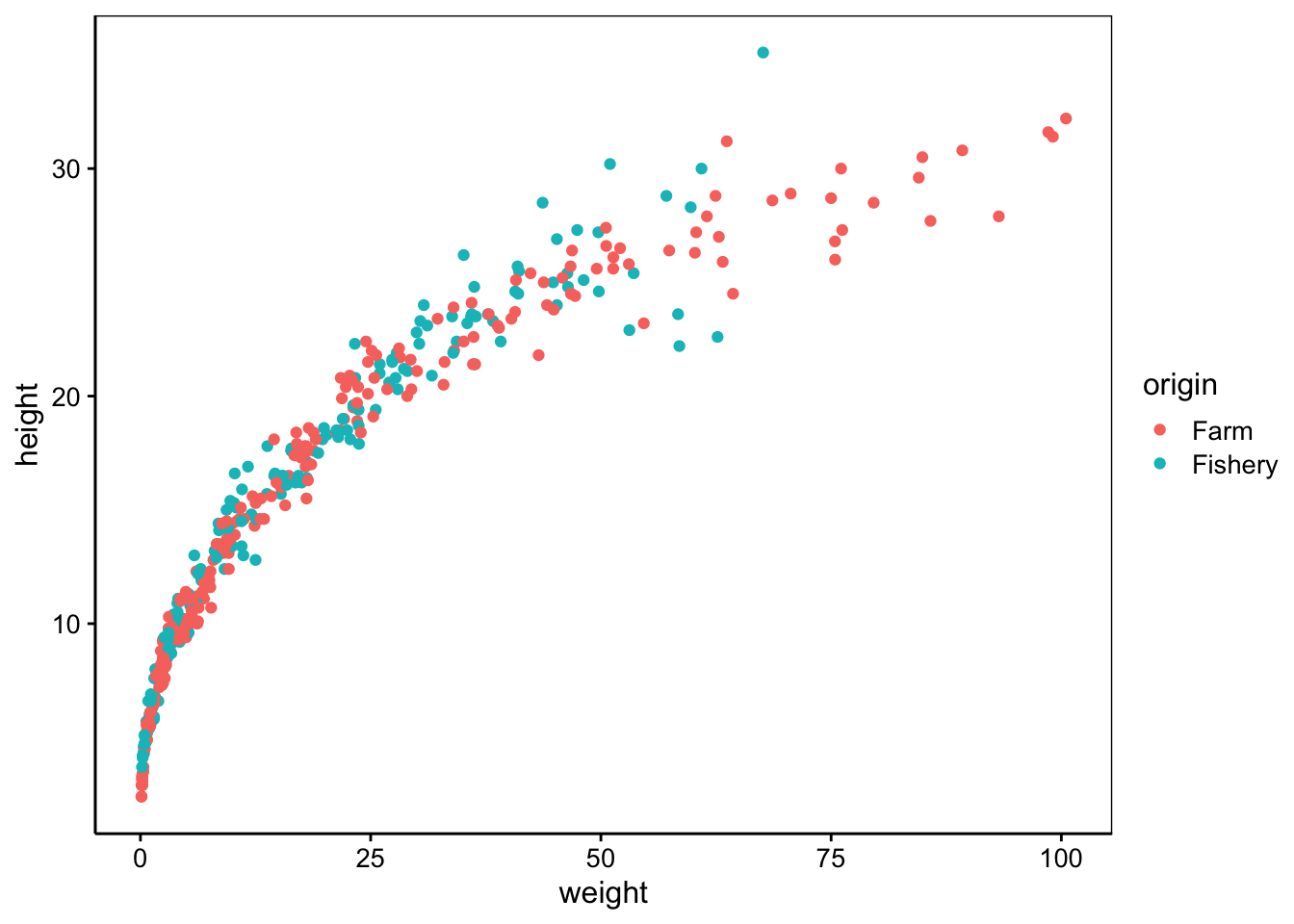
Comparez ceci avec le même graphique, mais obtenu à partir de différentes versions du jeu de données urchin_bio importé à l’aide de read() avec des valeurs différentes pour l’argument lang =.
urchin <- read("urchin_bio", package = "data.io") # langage par défaut défini dans SciViews::R()
urchin_en <- read("urchin_bio", package = "data.io", lang = "en")
urchin_fr <- read("urchin_bio", package = "data.io", lang = "fr")
urchin_FR <- read("urchin_bio", package = "data.io", lang = "FR")Les différences dans les labels sont observables sur le graphique ci-dessous.
a <- chart(urchin, height ~ weight %col=% origin) +
geom_point()
b <- chart(urchin_en, height ~ weight %col=% origin) +
geom_point()
c <- chart(urchin_fr, height ~ weight %col=% origin) +
geom_point()
d <- chart(urchin_FR, height ~ weight %col=% origin) +
geom_point()
combine_charts(list(a, b, c, d))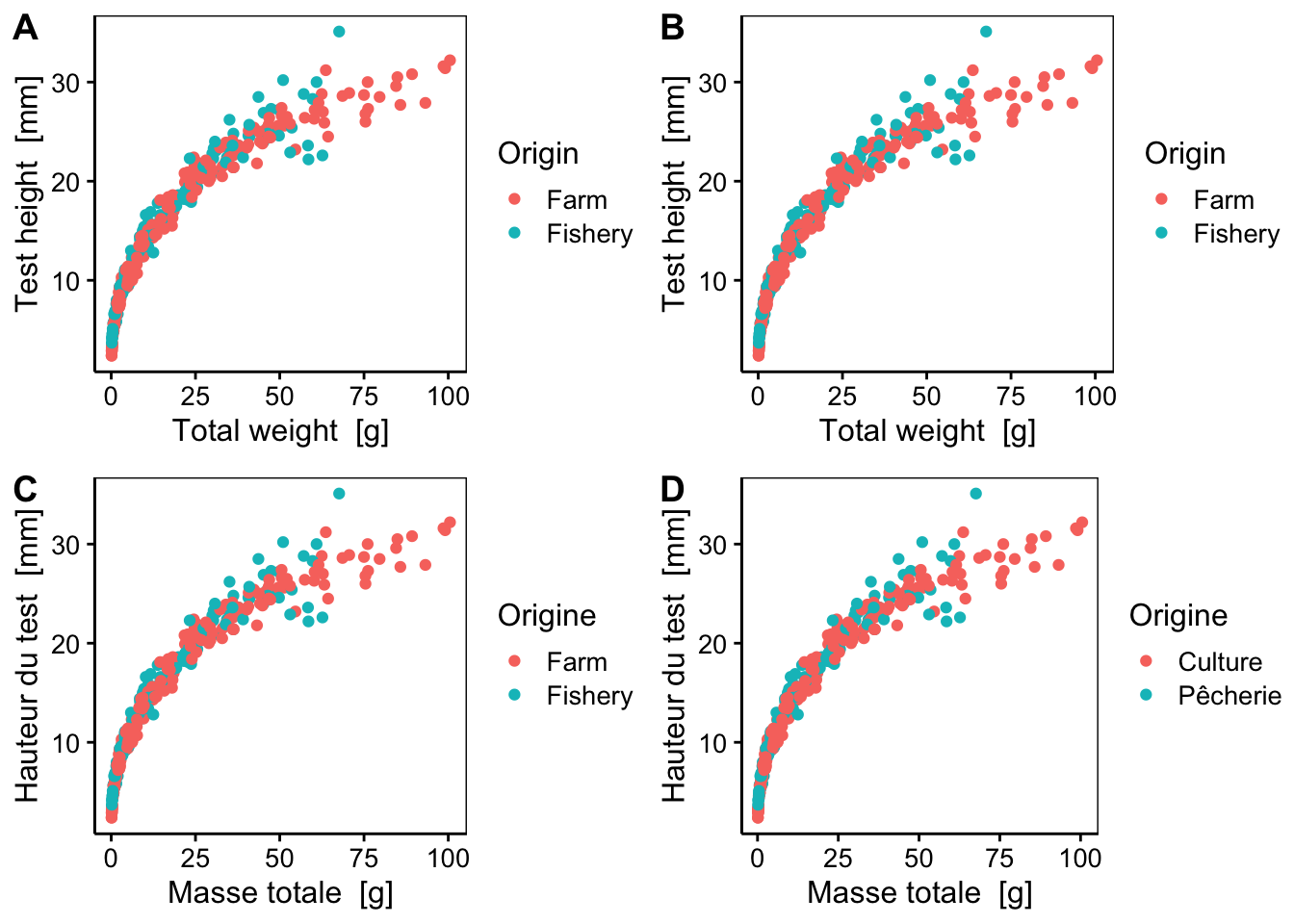
a&b: l’argumentlang =par défaut estlang = "en". Il utilise les labels et unités en anglais avec les unités dans le système international.c: l’argumentlang = "fr"utilise les labels et unités en français. Il laisse cependant les niveaux des variables facteurs en anglais (FarmetFishery) afin d’éviter de devoir changer les instructions de manipulation des données qui feraient référence à ces niveaux.d: l’argumentlang = "FR"ajoute les labels et unités en français. De plus, il traduit également les niveaux des variables facteurs (CultureetPêcherie).
Il vous est conseillé d’employer l’argument lang = "fr" lors de vos différents travaux. La langue internationale en science est l’anglais et vous serez très certainement amené dans votre carrière scientifique à produire des documents en français et en anglais. L’utilisation de lang = "fr"rend le même code réutilisable sur la version française ou anglaise, contrairement à lang = "FR". Observez les exemples ci-dessous.
# origin diameter1 diameter2 height buoyant_weight weight solid_parts
# <fctr> <num> <num> <num> <num> <num> <num>
# 1: Farm 53.1 54.5 26.3 9.5707 60.21 41.72
# 2: Farm 52.7 52.7 25.9 10.8380 63.23 46.58
# 3: Farm 54.0 54.2 24.5 10.6961 64.35 44.26
# 4: Farm 51.1 51.3 28.8 11.2132 62.45 44.99
# 5: Farm 52.1 53.6 31.2 11.0814 63.67 44.01
# 6: Farm 52.3 51.4 28.6 12.3791 68.62 53.88
# integuments dry_integuments digestive_tract dry_digestive_tract gonads
# <num> <num> <num> <num> <num>
# 1: 31.13 16.59 4.32 0.50 5.75
# 2: 29.91 17.83 5.83 0.59 9.97
# 3: 30.16 17.50 4.05 0.53 8.80
# 4: 30.91 18.23 5.63 0.74 7.60
# 5: 32.53 18.52 6.53 0.69 4.36
# 6: 33.72 20.05 8.42 0.87 9.92
# dry_gonads skeleton lantern test spines maturity sex
# <num> <num> <num> <num> <num> <int> <fctr>
# 1: 1.85 14.65 0.93 6.87 6.85 1 M
# 2: 3.03 16.09 1.02 7.48 7.59 2 F
# 3: 2.87 15.67 0.91 6.94 7.82 0 M
# 4: 2.55 16.63 1.08 6.62 8.93 0 M
# 5: 1.46 NA NA NA NA 0 M
# 6: 3.35 NA NA NA NA 0 F# origin diameter1 diameter2 height buoyant_weight weight solid_parts
# <fctr> <num> <num> <num> <num> <num> <num>
# 1: Farm 53.1 54.5 26.3 9.5707 60.21 41.72
# 2: Farm 52.7 52.7 25.9 10.8380 63.23 46.58
# 3: Farm 54.0 54.2 24.5 10.6961 64.35 44.26
# 4: Farm 51.1 51.3 28.8 11.2132 62.45 44.99
# 5: Farm 52.1 53.6 31.2 11.0814 63.67 44.01
# 6: Farm 52.3 51.4 28.6 12.3791 68.62 53.88
# integuments dry_integuments digestive_tract dry_digestive_tract gonads
# <num> <num> <num> <num> <num>
# 1: 31.13 16.59 4.32 0.50 5.75
# 2: 29.91 17.83 5.83 0.59 9.97
# 3: 30.16 17.50 4.05 0.53 8.80
# 4: 30.91 18.23 5.63 0.74 7.60
# 5: 32.53 18.52 6.53 0.69 4.36
# 6: 33.72 20.05 8.42 0.87 9.92
# dry_gonads skeleton lantern test spines maturity sex
# <num> <num> <num> <num> <num> <int> <fctr>
# 1: 1.85 14.65 0.93 6.87 6.85 1 M
# 2: 3.03 16.09 1.02 7.48 7.59 2 F
# 3: 2.87 15.67 0.91 6.94 7.82 0 M
# 4: 2.55 16.63 1.08 6.62 8.93 0 M
# 5: 1.46 NA NA NA NA 0 M
# 6: 3.35 NA NA NA NA 0 FPas d’adaptation nécessaire du code pour passer de urchin_en à urchin_fr.
# origin diameter1 diameter2 height buoyant_weight weight solid_parts
# <fctr> <num> <num> <num> <num> <num> <num>
# 1: Pêcherie 9.9 10.2 5.0 NA 0.5215 0.4777
# 2: Pêcherie 10.5 10.6 5.7 NA 0.6418 0.5891
# 3: Pêcherie 10.8 10.8 5.2 NA 0.7336 0.6770
# 4: Pêcherie 9.6 9.3 4.6 NA 0.3697 0.3438
# 5: Pêcherie 10.4 10.7 4.8 NA 0.6097 0.5587
# 6: Pêcherie 10.5 11.1 5.0 NA 0.6096 0.5509
# integuments dry_integuments digestive_tract dry_digestive_tract gonads
# <num> <num> <num> <num> <num>
# 1: 0.3658 NA 0.0525 0.0079 0
# 2: 0.4447 NA 0.0482 0.0090 0
# 3: 0.5326 NA 0.0758 0.0134 0
# 4: 0.2661 NA 0.0442 0.0064 0
# 5: 0.4058 NA 0.0743 0.0117 0
# 6: 0.4269 NA 0.0492 0.0097 0
# dry_gonads skeleton lantern test spines maturity sex
# <num> <num> <num> <num> <num> <int> <fctr>
# 1: 0 0.1793 0.0211 0.0587 0.0995 0 <NA>
# 2: 0 0.1880 0.0205 0.0622 0.1053 0 <NA>
# 3: 0 0.2354 0.0254 0.0836 0.1263 0 <NA>
# 4: 0 0.0630 0.0167 0.0180 0.0283 0 <NA>
# 5: 0 NA NA NA NA 0 <NA>
# 6: 0 NA NA NA NA 0 <NA>Le code a dû être modifié dans l’instruction sfilter() lors du passage à urchin_FR (Farm -> Pêcherie). Bien évidemment, pour un rapport plus formel en français, tout doit être traduit en français et l’option lang = "FR" accompagnée d’une vérification et une adaptation éventuelle du code est à préférer dans ce cas précis.
R permet également d’interroger des bases de données spécialisées, mais nous n’aborderons ce sujet spécifique qu’au cours de Science des Données Biologiques II en dernière année de Bachelier.↩︎
Si vous devez aussi accéder à vos données à partir d’autres langages comme Python, Java ou C++, utilisez un format commun reconnu par les différents logiciels. Le CSV fonctionne généralement bien, mais des formats binaires plus performants sont également disponibles. Parmi ces formats “inter-langages”, gardez un œil sur Apache Arrow qui offre des possibilités très intéressantes d’intercompatibilité de performance.↩︎