11.4 Facteurs hiérarchisés
Nous n’avons pas toujours la possibilité de croiser les deux facteurs. Considérons le cas d’une étude d’intercalibration. Nous avons un ou plusieurs échantillons répartis entre plusieurs laboratoires, et comme les analyses dépendent éventuellement aussi du technicien qui fait la mesure, nous demandons à chaque laboratoire de répéter les mesures avec deux de leurs techniciens. Problème : ici, il s’agit bien évidemment de techniciens différents dans chaque laboratoire. Comment faire, sachant que pour le modèle croisé, il faudrait que les deux mêmes techniciens aient fait toutes les mesures dans tous les laboratoires ?
La solution est le modèle à facteurs hiérarchisés qui s’écrit :
\[y_{ijk} = \mu + \tau1_j + \tau2_k(\tau1_j) + \epsilon_i \mathrm{\ avec\ } \ \epsilon_i \sim N(0, \sigma) \]
… et dans R, nous utiliserons la formule suivante :
\[y \sim fact1 + fact2\ \%in\%\ fact1\]
Voici un exemple concret. Un gros échantillon d’oeufs déshydratés homogène est réparti entre six laboratoires différents en vue de la détermination de la teneur en matières grasses dans cet échantillon. Le but de la manoeuvre est de déterminer si les laboratoires donnent des résultats consistants. Les deux techniciens de chaque laboratoire sont labellés one et two, mais ce sont en fait à chaque fois des techniciens différents dans chaque laboratoire54.
| Name | eggs |
| Number of rows | 48 |
| Number of columns | 4 |
| _______________________ | |
| Column type frequency: | |
| factor | 3 |
| numeric | 1 |
| ________________________ | |
| Group variables | None |
Variable type: factor
| skim_variable | n_missing | complete_rate | ordered | n_unique | top_counts |
|---|---|---|---|---|---|
| Lab | 0 | 1 | FALSE | 6 | I: 8, II: 8, III: 8, IV: 8 |
| Technician | 0 | 1 | FALSE | 2 | one: 24, two: 24 |
| Sample | 0 | 1 | FALSE | 2 | G: 24, H: 24 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| Fat | 0 | 1 | 0.39 | 0.15 | 0.06 | 0.31 | 0.37 | 0.43 | 0.8 | ▂▅▇▂▁ |
Commençons par corriger l’encodage erroné des techniciens qui ferait penser que seulement deux personnes ont travaillé dans l’ensemble des six laboratoires.
| Name | eggs |
| Number of rows | 48 |
| Number of columns | 4 |
| _______________________ | |
| Column type frequency: | |
| factor | 3 |
| numeric | 1 |
| ________________________ | |
| Group variables | None |
Variable type: factor
| skim_variable | n_missing | complete_rate | ordered | n_unique | top_counts |
|---|---|---|---|---|---|
| Lab | 0 | 1 | FALSE | 6 | I: 8, II: 8, III: 8, IV: 8 |
| Technician | 0 | 1 | FALSE | 12 | I.o: 4, II.: 4, III: 4, IV.: 4 |
| Sample | 0 | 1 | FALSE | 2 | G: 24, H: 24 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| Fat | 0 | 1 | 0.39 | 0.15 | 0.06 | 0.31 | 0.37 | 0.43 | 0.8 | ▂▅▇▂▁ |
Nous avons à présent douze techniciens notés I.one, I.two, II.one, II.two, … Nous pouvons visualiser ces données. Comme nous n’avons que quatre réplicats par technicien, nous nous limitons à la représentation des observations de départ et des moyennes.
chart(data = eggs, Fat ~ Lab %col=% Technician) +
geom_jitter(width = 0.05, alpha = 0.5) +
geom_point(data = group_by(eggs, Lab, Technician) %>.%
summarise(., means = mean(Fat, na.rm = TRUE)),
f_aes(means ~ Lab), size = 3, col = "red")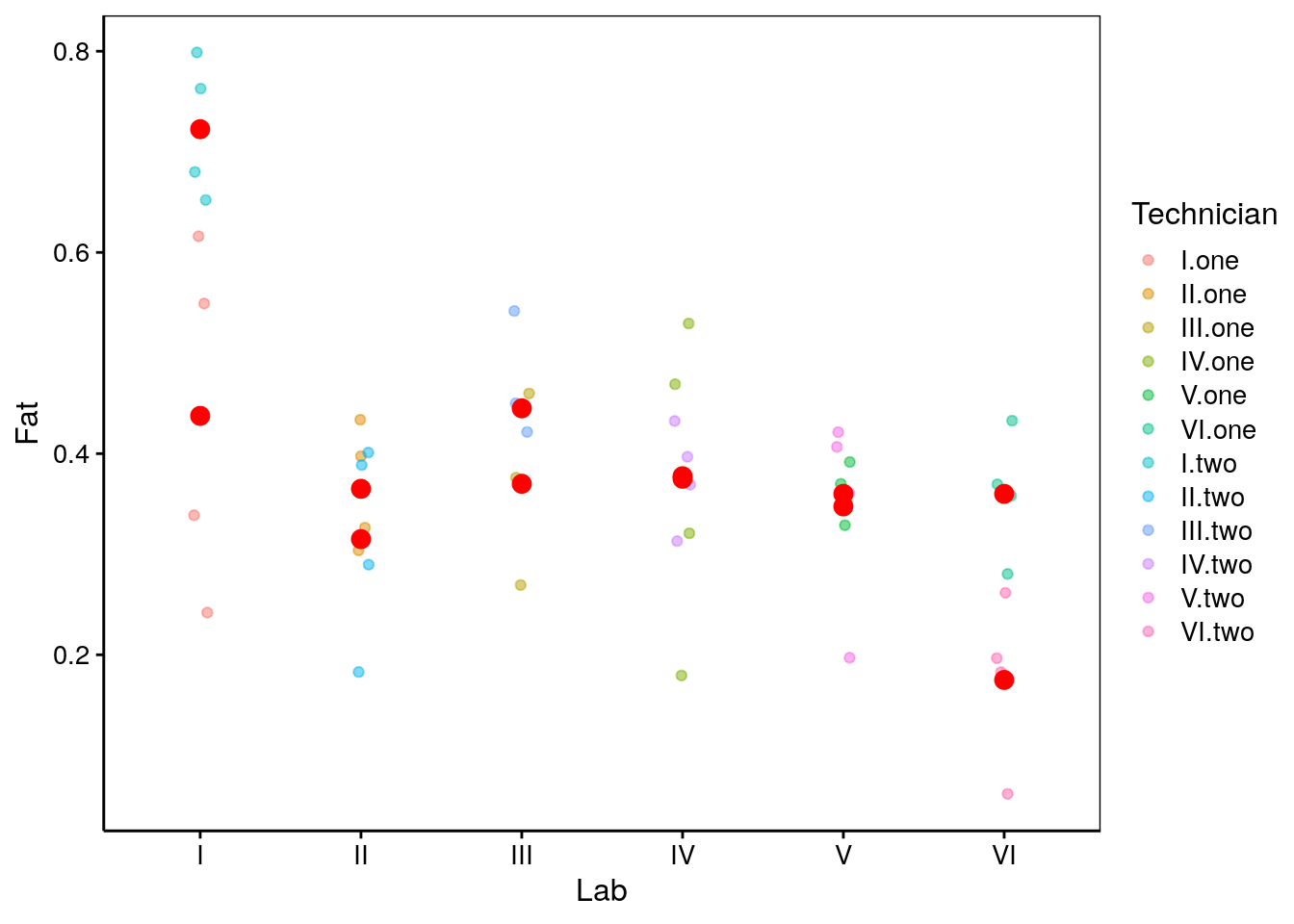
Figure 11.5: Mesures de fractions en matières grasses dans des oeufs dans six laboratoires, par douze techniciens différents. Les points rouges sont les moyennes par technicien.
Vérifions l’homoscédasticité. Ici, il suffit de considérer la variable Technician (une fois correctement encodée !) parce que dans cette forme de modèle, le facteur qui est imbriqué (Technician) est celui à partir duquel les résidus sont calculés. Nous utiliserons un seuil \(\alpha\) classique de 5% pour l’ensemble de nos tests dans cette étude.
#
# Bartlett test of homogeneity of variances
#
# data: Fat by Technician
# Bartlett's K-squared = 13.891, df = 11, p-value = 0.2391Avec une valeur p de 23,9%, nous pouvons considérer qu’il y a homoscédasticité. Voilà l’ANOVA (utilisez le “snippet” two-way ANOVA (nested model) le menu contextuel hypothesis tests: means que vous obtenez en tapant .hm).
# Analysis of Variance Table
#
# Response: Fat
# Df Sum Sq Mean Sq F value Pr(>F)
# Lab 5 0.44303 0.088605 9.5904 6.989e-06 ***
# Lab:Technician 6 0.24747 0.041246 4.4644 0.001786 **
# Residuals 36 0.33260 0.009239
# ---
# Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Nous voyons que, dans le cas présent, l’effet technicien ne peut pas être testé. Nous avons l’effet labo et les interactions entre les techniciens et les labos qui sont présentés. Les deux sont significatifs ici. Nous avons à la fois des différences significatives qui apparaissent entre labos, mais aussi, des variation d’un labo à l’autre entre techniciens (interactions).
Nous devons maintenant vérifier la distribution normale des résidus dans ce modèle (Fig. 11.6). Ici rien à redire, la distribution est conforme à nos attentes.
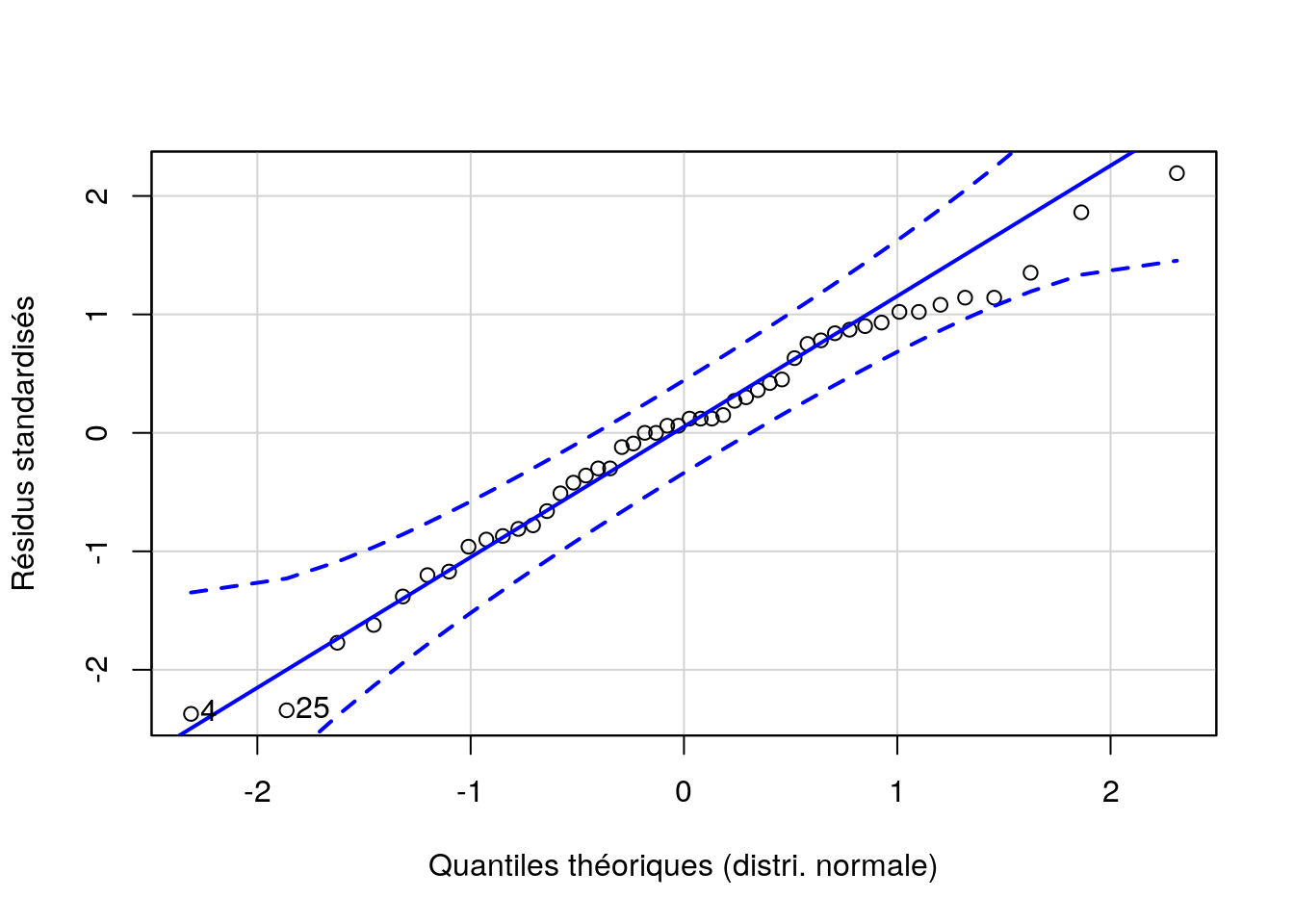
Figure 11.6: Graphique quantile-quantile des résidus pour l’ANOVA à deux facteurs hiérarchisés pour la variable Fat du jeu de données eggs.
# [1] 4 25L’effet qui nous intéresse en priorité est l’effet laboratoire. Effectuons des tests “post hoc” sur cet effet pour déterminer quel(s) laboratoire(s) diffèrent entre eux. Le code que nous utilisons habituellement ne fonctionne pas dans le cas d’un modèle hiérarchisé, mais nous pouvons utiliser la fonction TukeyHSD() à la place, en partant d’un modèle similaire créé à l’aide de la fonction aov().
# Tukey multiple comparisons of means
# 95% family-wise confidence level
#
# Fit: aov(formula = Fat ~ Lab + Technician %in% Lab, data = eggs)
#
# $Lab
# diff lwr upr p adj
# II-I -0.24000 -0.38459081 -0.095409195 0.0002088
# III-I -0.17250 -0.31709081 -0.027909195 0.0116356
# IV-I -0.20375 -0.34834081 -0.059159195 0.0019225
# V-I -0.22625 -0.37084081 -0.081659195 0.0004902
# VI-I -0.31250 -0.45709081 -0.167909195 0.0000021
# III-II 0.06750 -0.07709081 0.212090805 0.7240821
# IV-II 0.03625 -0.10834081 0.180840805 0.9733269
# V-II 0.01375 -0.13084081 0.158340805 0.9997181
# VI-II -0.07250 -0.21709081 0.072090805 0.6611505
# IV-III -0.03125 -0.17584081 0.113340805 0.9861403
# V-III -0.05375 -0.19834081 0.090840805 0.8705387
# VI-III -0.14000 -0.28459081 0.004590805 0.0624025
# V-IV -0.02250 -0.16709081 0.122090805 0.9969635
# VI-IV -0.10875 -0.25334081 0.035840805 0.2356038
# VI-V -0.08625 -0.23084081 0.058340805 0.4817411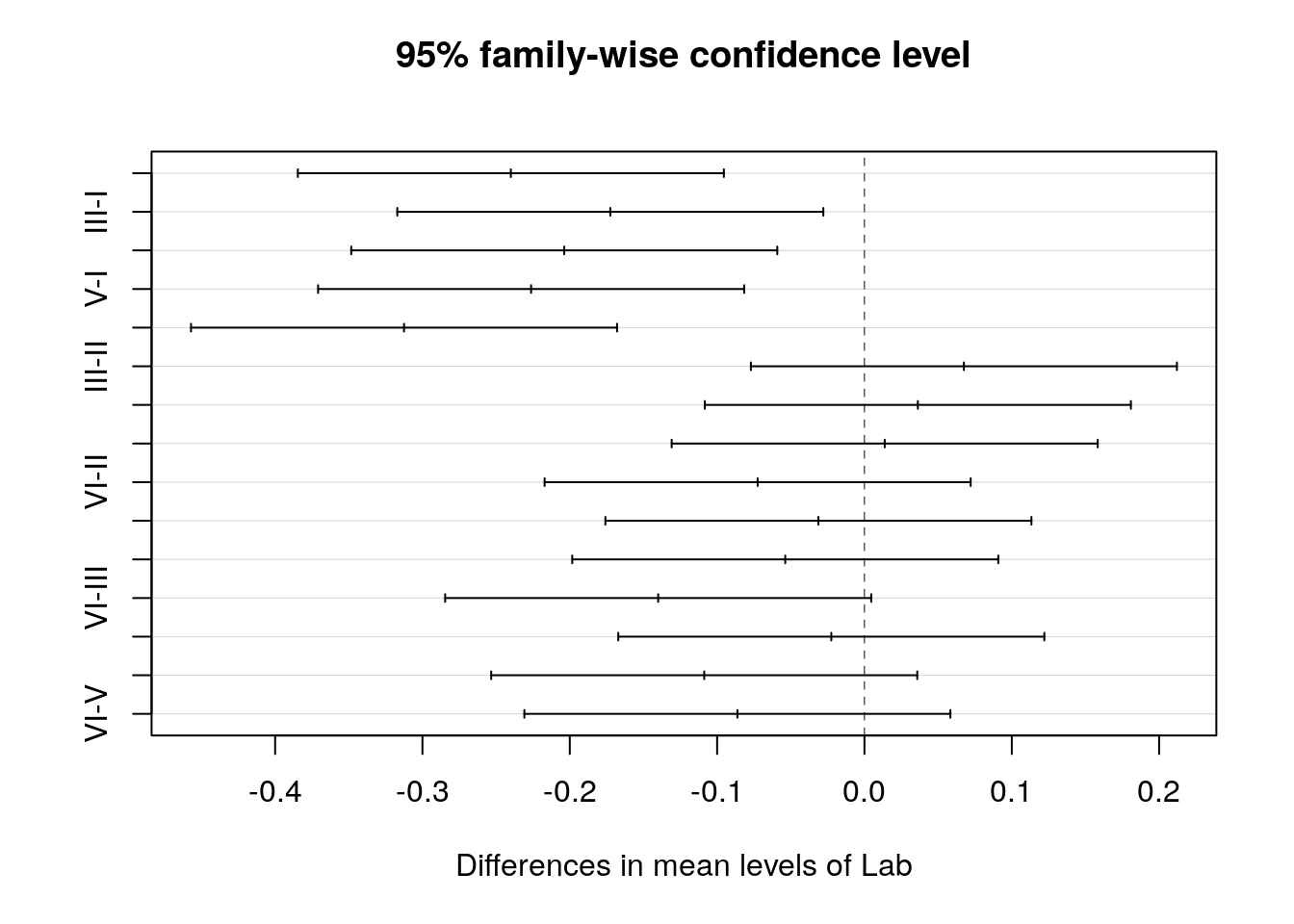
Nous pouvons observer des différences significatives au seuil \(\alpha\) de 5% entre le labo I et tous les autres labos. Les autres comparaisons n’apparaissent pas significatives.
11.4.1 Simplification du modèle
Nous pourrions être tentés de simplifier notre analyse en ne testant que l’effet laboratoire. Dans ce cas, nous tomberions dans le piège de la pseudo-réplication. Nous pourrions aussi travailler sur la mesure moyenne des mesures pour chaque technicien. Du coup, nous aurions deux valeurs par laboratoire, chaque fois réalisée par un technicien différent. Nous pourrions donc considérer que les données sont indépendantes les unes des autres et nous pourrions réduite le problème à un effet unique, celui du laboratoire.
Si nous n’avons plus que deux mesures par laboratoire au lieu de deux fois quatre, nous gagnons d’un autre côté puisque l’écart type de la moyenne d’un échantillon et l’écart type de la population divisé par la racine carré de n. Donc, l’écart type sur les mesures moyennes est alors deux fois plus faible, ce qui se répercutera de manière positive sur l’ANOVA. La distribution des résidus sera une distribution de Student, mais elle est symétrique et pas trop différente d’une distribution normale. Cela pourrait passer. Mais il se peut que la réduction de l’information soit telle que le test perde complètement sa puissance. Illustrons ce phénomène avec le jeu de données eggs. Nous créons le jeu de données eggs_means reprenant les moyennes des quatre mesures par techinicien dans la variable `Fat_mean.
eggs %>.%
group_by(., Technician) %>.%
summarise(., Fat_mean = mean(Fat), Lab = unique(Lab)) ->
eggs_means
skimr::skim(eggs_means)| Name | eggs_means |
| Number of rows | 12 |
| Number of columns | 3 |
| _______________________ | |
| Column type frequency: | |
| factor | 2 |
| numeric | 1 |
| ________________________ | |
| Group variables | None |
Variable type: factor
| skim_variable | n_missing | complete_rate | ordered | n_unique | top_counts |
|---|---|---|---|---|---|
| Technician | 0 | 1 | FALSE | 12 | I.o: 1, II.: 1, III: 1, IV.: 1 |
| Lab | 0 | 1 | FALSE | 6 | I: 2, II: 2, III: 2, IV: 2 |
Variable type: numeric
| skim_variable | n_missing | complete_rate | mean | sd | p0 | p25 | p50 | p75 | p100 | hist |
|---|---|---|---|---|---|---|---|---|---|---|
| Fat_mean | 0 | 1 | 0.39 | 0.13 | 0.18 | 0.36 | 0.37 | 0.39 | 0.72 | ▁▇▂▁▁ |
eggs_means %>.%
group_by(., Lab) %>.%
summarise(., mean = mean(Fat_mean), sd = sd(Fat_mean), count = sum(!is.na(Fat_mean)))# # A tibble: 6 x 4
# Lab mean sd count
# <fct> <dbl> <dbl> <int>
# 1 I 0.58 0.202 2
# 2 II 0.34 0.0354 2
# 3 III 0.408 0.0530 2
# 4 IV 0.376 0.00177 2
# 5 V 0.354 0.00884 2
# 6 VI 0.267 0.131 2Représentation graphique et vérification de l’homoscédasticité.
chart(eggs_means, Fat_mean ~ Lab) +
geom_point() +
geom_point(data = group_by(eggs_means, Lab) %>.%
summarise(., means = mean(Fat_mean, na.rm = TRUE)),
f_aes(means ~ Lab), size = 3, col = "red")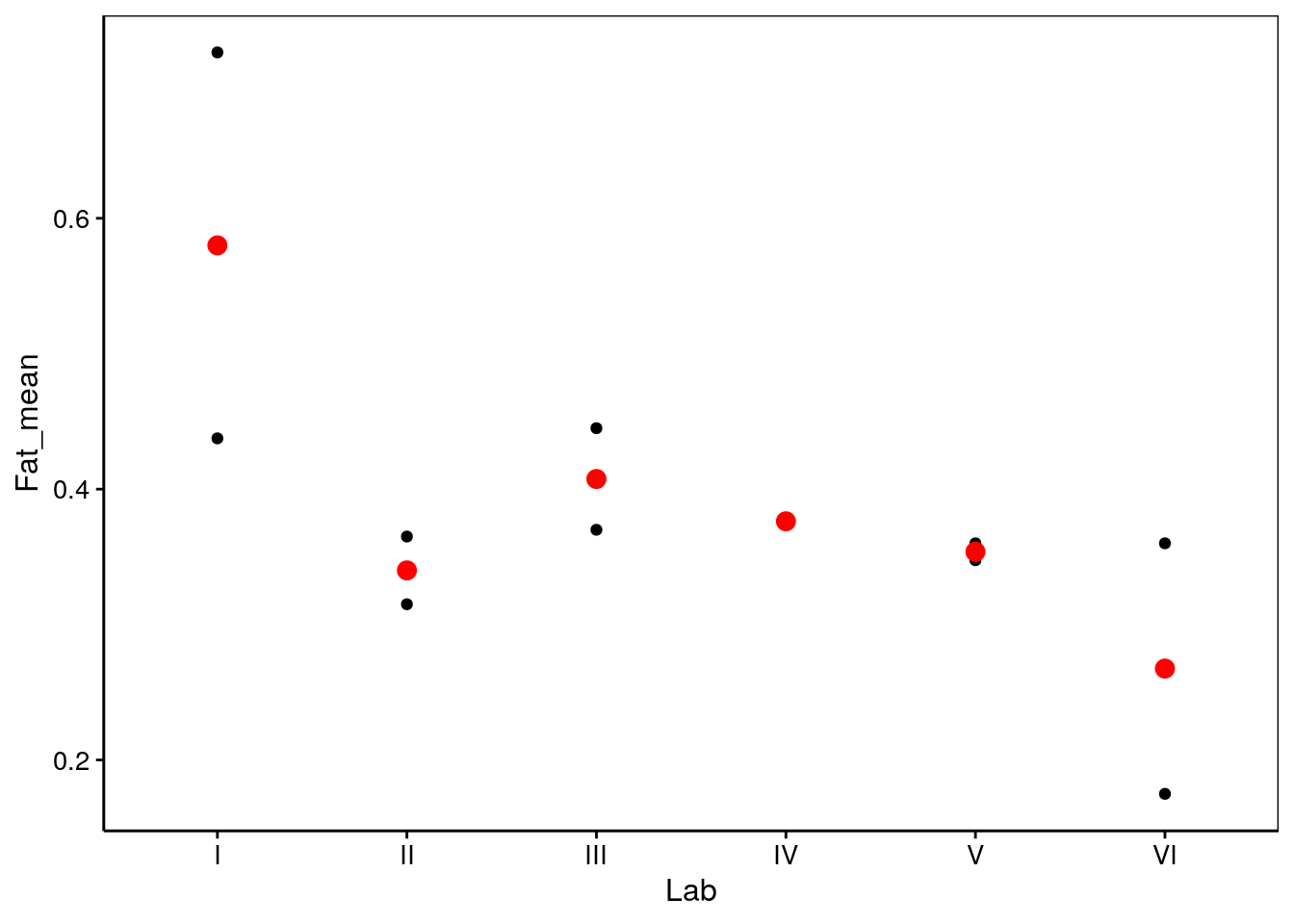
#
# Bartlett test of homogeneity of variances
#
# data: Fat_mean by Lab
# Bartlett's K-squared = 10.452, df = 5, p-value = 0.0634Analyse de variance à un facteur.
# Analysis of Variance Table
#
# Response: Fat_mean
# Df Sum Sq Mean Sq F value Pr(>F)
# Lab 5 0.110756 0.022151 2.1482 0.1895
# Residuals 6 0.061869 0.010311anova. %>.%
broom::augment(.) %>.%
car::qqPlot(.$.std.resid, distribution = "norm",
envelope = 0.95, col = "Black", xlab = "Quantiles théoriques (distri. normale)",
ylab = "Résidus standardisés")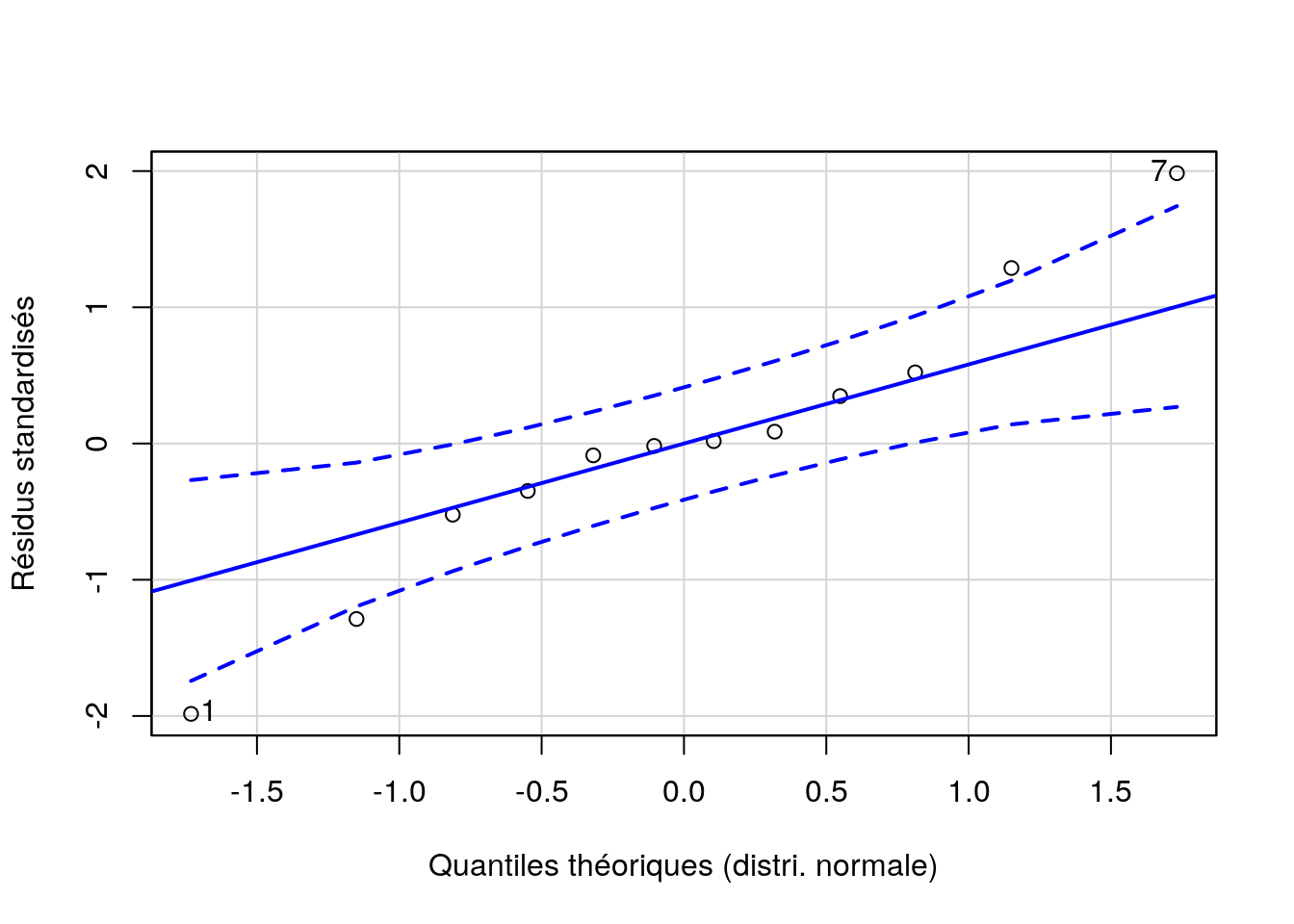
# [1] 1 7Nous n’avons plus d’effet significatif, malgré que le labo I obtient, en moyenne, une mesure beaucoup plus forte que les autres. En fait, en réduisant de la sorte nos données, nous avons perdu tellement d’information que le test a perdu toute puissance et n’est plus capable de détecter de manière significative des différences entre moyennes pourtant importantes. Si nous avions quatre techniciens par labo qui auraient tous dosés les échantillons en duplicats (également huit mesures par labo au total), nous n’aurions pas une perte d’information aussi forte en effectuant quatre moyennes de duplicats par labo, et l’analyse simplifiée aurait peut-être été utilisable. Il faut voir au cas par cas…
Conditions d’application
Les conditions d’application sont les mêmes que pour l’ANOVA à deux facteurs sans interactions, sauf qu’ici, les interactions sont bien évidemment incluses dans le modèle par construction.
La variable
SamplevalantGouHne sera pas utilisée ici. En fait, au départ, les initiateurs de l’expérience ont fait croire aux laboratoires qu’il s’agissait de deux échantillons différents alors que c’est le même en réalité.↩︎