7.1 Dates
La gestion du temps et les variables de temps peuvent sembler simples d’apparence. Nous pouvons facilement déterminer la date et l’heure qu’il est en regardant une montre et un calendrier.
À vous de jouer !
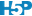
Si l’on tient compte du calendrier grégorien, des fuseaux horaires, du temps universel coordonné, de l’heure d’été, …. On s’aperçoit que le temps est complexe.
Le calendrier grégorien est le calendrier le plus utilisé actuellement dans le monde. Ce calendrier définit qu’une année comprend 4 trimestres et 12 mois. Les mois varient en longueur entre 28 et 31 jours. Une semaine comprend 7 jours. Une année comprend 365 jours à l’exception des années bissextiles. Si vous n’avez pas mal de tête, on continue.
Les fuseaux horaires ont eu pour objectif de définir une heure commune pour un espace géographique déterminé. C’est l’utilisation de chemin de fer qui a rendu la mise en place d’une heure commune indispensable.
En pratique, une échelle de temps est utilisée comme référence par la majorité des pays du globe. Il s’agit du temps universel coordonné (UTC, coordinated universal time). Le terme GMT était utilisé précédemment. Il signifiait Greenwich Mean time. À ce temps de référence, on va ajouter ou soustraire un nombre entier (avec des exceptions comme dans tout système avec l’ajout ou la soustraction d’un nombre entier plus 30 ou 45 minutes).
UTC est un compromis entre le temps atomique international et temps universel. Il faut savoir que le temps universel coordonné est synchronisé avec la rotation de la Terre qui n’est pas fixe. Le service international de la rotation terrestre et des systèmes de référence décide de l’ajout de seconde, une seconde intercalaire (leap second) à UTC comme le premier janvier 2009.
Après avoir lu ces quelques notions, pensez-vous toujours que le temps est simple ?
Comment le temps est-il géré dans R ? On retrouve les principaux objets suivants dans R pour gérer les temps : Date, POSIXct, POSIXlt, difftime. Le package {lubridate} propose en plus Period.
L’objet Date va s’intéresser au temps exprimé en jours, mois et années. Le jour 0 d’un objet Date est par convention le 1970-01-01. Il s’agit de l’heure Unix (mais nous ne détaillerons pas cette notion).
# [1] "2024-03-19"# [1] "Date"L’objet POSIXt est un objet plus précis qui tient compte de la date et de l’heure. On retrouve deux variantes des objets POSIXT qui sont POSIXct et POSIXlt. Le POSIXCT enregistre le temps depuis l’origine qui est l’heure Unix alors que le POSIXlt utilise des paramètres attributs spécifiques.
# [1] "2024-03-19 08:53:03 CET"# [1] "POSIXct" "POSIXt"Enfin les objets difftime sont des objets spécifiques aux différences entre deux dates.
# Time difference of 16228 days# Seconde manière de calculer une différence entre deux dates
diff1 <- difftime(today(), ymd("1979-10-14"))
diff == diff1 # diff et diff1 sont identiques# [1] TRUE# [1] "difftime"7.1.1 Conversion d’une chaîne de caractères en date
En pratique vous serez souvent amené à convertir des chaînes de caractères en variable de temps. Pour ce faire, le package {lubridate} comprend des fonctions très utiles pour convertir des chaînes de caractères en dates. Regardez par vous-même pour en déduire la logique simple ci-dessous :
- Les dates :
# [1] "2015-01-25"# [1] "2015-01-25"À vous de jouer !
- Les heures :
# [1] "1H 10M 0S"# [1] "20H 10M 1S"# [1] "20H 10M 1S"- Les dates et heures :
# [1] "2010-10-01 20:10:00 UTC"# [1] "2015-01-25 12:25:35 UTC"À vous de jouer !
Par défaut, ce package utilise le temps universel coordonné. Il est cependant possible de définir le fuseau horaire avec l’argument tz=.
# [1] "2024-03-19 08:53:03 CET"Ces fonctions sont capables de gérer des formats de dates différentes en même temps. Essayez cependant d’éviter ce genre d’erreur d’encodage.
ymd_hms(c("2015-Jan-25 12:25:35", "2015-01-15T122535", "2015/01/25 12:25:35"),
tz = "Europe/Brussels")# [1] "2015-01-25 12:25:35 CET" "2015-01-15 12:25:35 CET"
# [3] "2015-01-25 12:25:35 CET"Plus de détails…
Pour avoir plus de flexibilité, il est possible d’utiliser la fonction parse_date_time() qui va permettre de spécifier des formats spécifiques.
- Année
- %Y : 4 chiffres
- %y : 2 chiffres
- Mois
- %m : 2 chiffres
- %b : nom abrégé
- %B : nom complet
- Jour
- %d : 2 chiffres
- Heure
- %H : 0-23h
- %i combiné avec %p : 0-12 suivi am/pm
- %M : minutes
- %S : secondes entières
- %OS : secondes réelles
- %Z : fuseau horaire
- %z : décalage par rapport à UTC : +0100 (plus 1h)
- Valeurs à ignorer
- %. : ignore des caractères non numériques
- %* : ignore une suite de caractères non numériques
Vous serez un jour confronté à un tableau de données comme suit avec plusieurs colonnes qui renseigne sur le temps de la prise de mesure.
(df <- tibble(
annee = c(2010, 2011, 2012, 2013),
mois = c(01, 01, 01, 01),
jour = c(01, 01, 01, 01)
))# # A tibble: 4 x 3
# annee mois jour
# <dbl> <dbl> <dbl>
# 1 2010 1 1
# 2 2011 1 1
# 3 2012 1 1
# 4 2013 1 1Rassurez-vous, il n’est pas nécessaire de combiner les trois colonnes puis de les transformer en variable temporelle. La fonction make_date() permet de créer une variable qui combine nos trois colonnes. Il existe la même variante pour make_datetime().
# # A tibble: 4 x 4
# annee mois jour date
# <dbl> <dbl> <dbl> <date>
# 1 2010 1 1 2010-01-01
# 2 2011 1 1 2011-01-01
# 3 2012 1 1 2012-01-01
# 4 2013 1 1 2013-01-017.1.2 Temps biologique
Lorsque vous étudiez un phénomène biologique, il faut vous poser la question de la temporalité de votre expérience.
Est-ce que votre expérience dure quelques heures ? quelques jours ? quelques mois ? plusieurs années ?
Cette question est cruciale pour traiter vos données. Prenons trois petits cas pratiques théoriques.
- La croissance de coraux sur 30 jours.
Vous réalisez une expérience sur la croissance d’une espèce de corail durant 30 jours en aquarium. Cette expérience débute le 20 septembre 2019.
corals <- tibble::tibble(
weight = 2 + exp(0.02 * 0:29),
date = lubridate::ymd("2019-09-20") + lubridate::ddays(0:29),
nb = 0:29
)
corals <- labelise(
corals,
label = list(
weight = "Masse", date = "Date", nb = "Temps"),
units = list(
weight = "g", nb = "jour"))
a <- chart(corals, weight ~ date) +
geom_line() +
geom_point()
b <- chart(corals, weight ~ nb) +
geom_line() +
geom_point()
combine_charts(list(a,b), nrow = 2)
On comprend rapidement que le second graphique est plus intéressant. Il n’est pas nécessaire de connaitre les dates précises lorsque l’on étudie la croissance d’un organisme dans un aquarium sur 30 jours.
- Le temps perdu dans les embouteillages.
Lorsque l’on étudie le temps perdu dans les bouchons à Bruxelles. Il sera très important de spécifier les jours de la semaine. En effet, nous savons que les bouchons vont varier entre la semaine ou le week-end ou encore lors d’un jour férié. On ne peut donc pas se passer d’une date précise lorsque l’on étudie ce genre de phénomène.
- L’évolution de la température sur 20 ans.
Lorsque l’on étudie des séries temporelles en écologie, on ne s’intéresse pas aux années bissextiles, ou au mois de 29 ou 31 jours.
Le package {pastecs} propose de diviser une année en multiple de 4, 12, 48 périodes de temps, … Une fréquence de 48 correspond environ à la semaine. Le tableau ci-dessous représente le temps en biologie.
| Temps | Période |
|---|---|
| Année | 1 |
| mois | 12 |
| semaines | 48 |
| jours | 384 |
En biologie, on va s’intéresser principalement à des cycles particuliers comme le cycle circadien, la succession des saisons, le cycle lunaire,…
Pour en savoir plus
Chapter 16 Dates and times in R For Data Science : le chapitre 16 traite des variables temporelles.
À vous de jouer !
Réalisez le travail D07Ia_21M_data, partie I.
Travail individuel pour les étudiants inscrits au cours de Science des Données Biologiques IV à l’UMONS à terminer avant le 2021-10-22 23:59:59.
Initiez votre projet GitHub Classroom
Voyez les explications dans le fichier README.md, partie I.