2.2 Différents algorithmes
Les algorithmes de classification supervisée se subdivisent en trois grandes classes :
ceux qui utilisent un modèle (linéaire ou non linéaire) sous-jacent pour mettre en relation les attributs et les classes. Par exemple, l’analyse discriminante linéaire que nous connaissons déjà appartient à cette catégorie. Nous verrons également les machines à vecteurs supports et les réseaux de neurones dans cette catégorie.
ceux qui font appel à un indice de similarité calculé entre les individus (attribution de la classe correspondant aux individus les plus ressemblants à celui qu’on teste). Ce sont les techniques dites du plus proche voisin et de la quantification vectorielle (voir ci-après dans ce module).
enfin, les techniques qui définissent une suite de règle de division dichotomique du jeu de données (qui se matérialisent par un ou plusieurs arbres de décision). Il s’agit du partitionnement récursif et de la forêt aléatoire, par exemple. Nous verrons ces méthodes à la fin du module.
Nous avons déjà abordé l’analyse discriminante linéaire dans le précédent module et nous allons étudier ici quelques autres algorithmes de chaque catégorie, ainsi que dans le module suivant. Enfin, nous mettrons tout cela en musique pour étudier différents algorithmes de classification en pratique, en vue de choisir celui qui nous semble le plus adéquat pour le cas étudié.
2.2.1 Indiens diabétiques
Afin d’explorer et comparer l’utilisation de différents algorithmes de classification supervisée, nous reprendrons notre jeu de données pima concernant une population d’Amérindiens qui sont connus pour compter un haut taux d’obèses et de diabétiques. Nous avions déjà utilisé ces données pour illustrer l’ACP dans le cours de Science des Données Biologiques II. Pour rappel, le jeu de données se présente comme suit :
# # A tibble: 768 x 9
# pregnant glucose pressure triceps insulin mass pedigree age diabetes
# <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <fct>
# 1 6 148 72 35 NA 33.6 0.627 50 pos
# 2 1 85 66 29 NA 26.6 0.351 31 neg
# 3 8 183 64 NA NA 23.3 0.672 32 pos
# 4 1 89 66 23 94 28.1 0.167 21 neg
# 5 0 137 40 35 168 43.1 2.29 33 pos
# 6 5 116 74 NA NA 25.6 0.201 30 neg
# 7 3 78 50 32 88 31 0.248 26 pos
# 8 10 115 NA NA NA 35.3 0.134 29 neg
# 9 2 197 70 45 543 30.5 0.158 53 pos
# 10 8 125 96 NA NA NA 0.232 54 pos
# # … with 758 more rowsNous avons huit variables quantitatives (discrète comme pregnant, ou continues pour les autres) et une variable qualitative diabetes. Voici quelques informations sur ces différentes variables :
diabetes, variable qualitative à deux niveaux indique si l’individu est diabétique (pos) ou non (neg). C’est naturellement la variable réponse que l’on cherchera à prédire ici,pregnantest le nombre de grossesses que cette femme a eue (il s’agit uniquement d’un échantillon de femmes de 21 ans ou plus),glucoseest le taux de glucose dans le plasma sanguin (test standardisé renvoyant une valeur sans unités),pressureest la pression sanguine diastolique, en mm de mercure,tricepsest l’épaisseur mesurée d’un pli de peau au niveau du triceps en mm. Il s’agit d’une mesure permettant d’estimer l’obésité, ou en tous cas, la couche de graisse sous-cutanée à ce niveau,insulinest la détermination de la quantité d’insuline deux heures après prise orale de sucre dans un test standardisé, en µU/mL,massest en réalité l’IMC, indice de masse corporelle que vous connaissez bien (masse/ taille2), en kg/m2, un autre indice d’obésité couramment employé,pedigreeest un indice de prédisposition au diabète établi en fonction des informations sur la famille (sans unités),ageest l’âge de l’indienne exprimé en années.
En fonction de ces informations, nous pouvons labelliser notre jeu de données comme suit :
pima <- labelise(pima,
label = list(
diabetes = "Diabète",
pregnant = "Grossesses", glucose = "Test glucose",
pressure = "Pression sanguine", triceps = "Gras au triceps",
insulin = "Insuline", mass = "IMC",
pedigree = "Pedigree", age = "Âge"),
units = list(
pressure = "mm Hg", triceps = "mm",
insulin = "µU/mL", mass = "kg/m^2", age = "années"
)
)Du point de vue du balancement des observations, nous avons ceci :
#
# neg pos
# 500 268Encore une fois, nous avons plus de cas négatifs que de positifs et pourrions souhaiter balancer les deux classes. La différence n’est cependant pas à ce point dramatique et nous continuerons dans cet exemple avec les données telles quelles.
Ce jeu de données contient 768 cas, mais deux variables (triceps et insulin) ont un très grand nombre de valeurs manquantes comme nous le voyons ci-dessous.
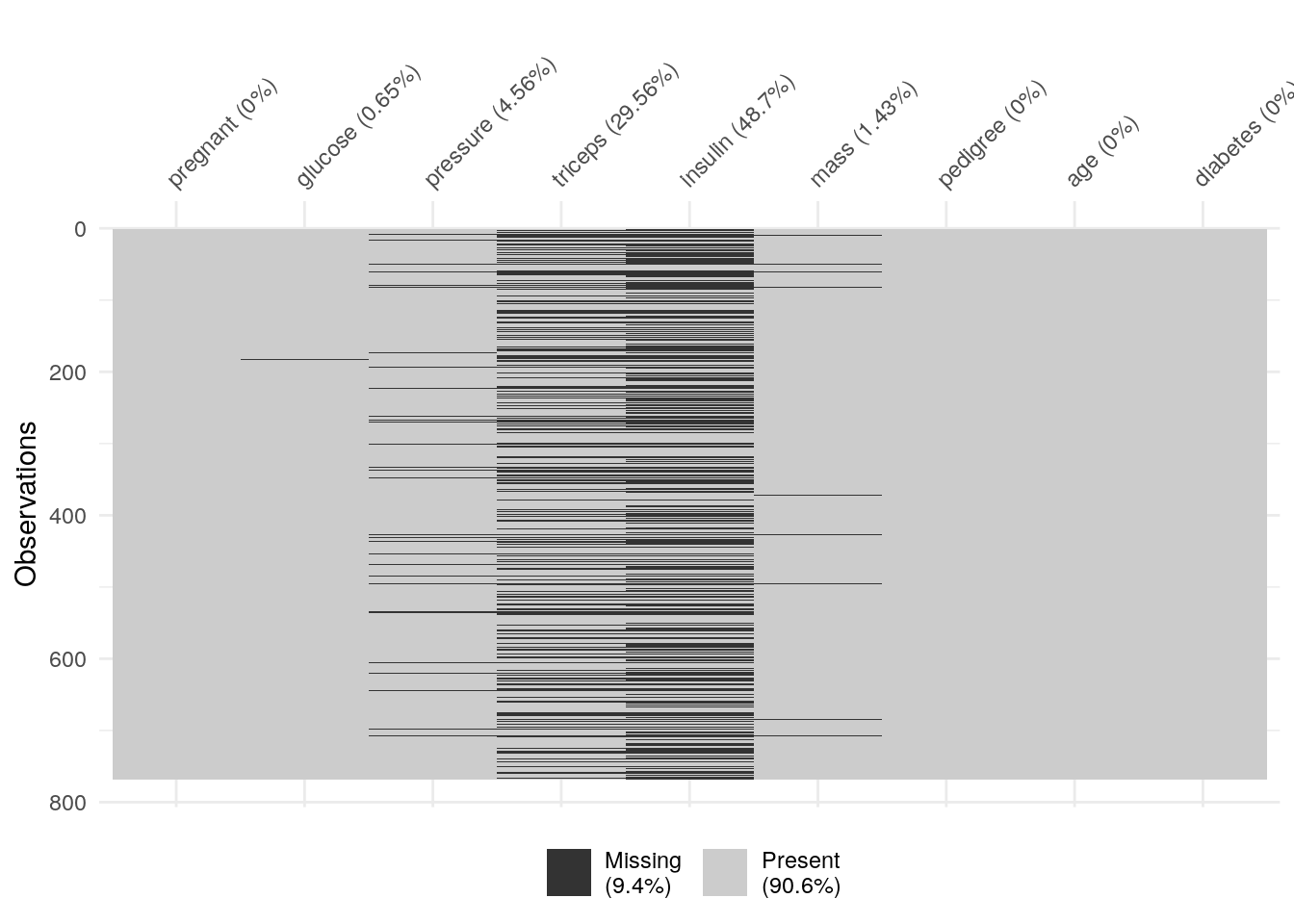
Comme pour l’ACP, nos sets d’apprentissage et de test ne peuvent pas contenir de valeurs manquantes. Si nous utilisons drop_na() sur tout le tableau, toute ligne contenant au moins une valeur manquante sera éliminée. Cela donne ceci :
# [1] 392Nous avons certes un tableau propre, mais nous avons perdu près de la moitié des données ! Or nous n’avons jamais assez de données en classification supervisée. Nous pourrions aussi considérer la possibilité de laisser tomber les colonnes qui contiennent trop de valeurs manquantes. En première approche, afin de déterminer si la perte de ces variables pourrait être préjudiciable à notre analyse, nous pourrions inspecter la matrice de corrélation.
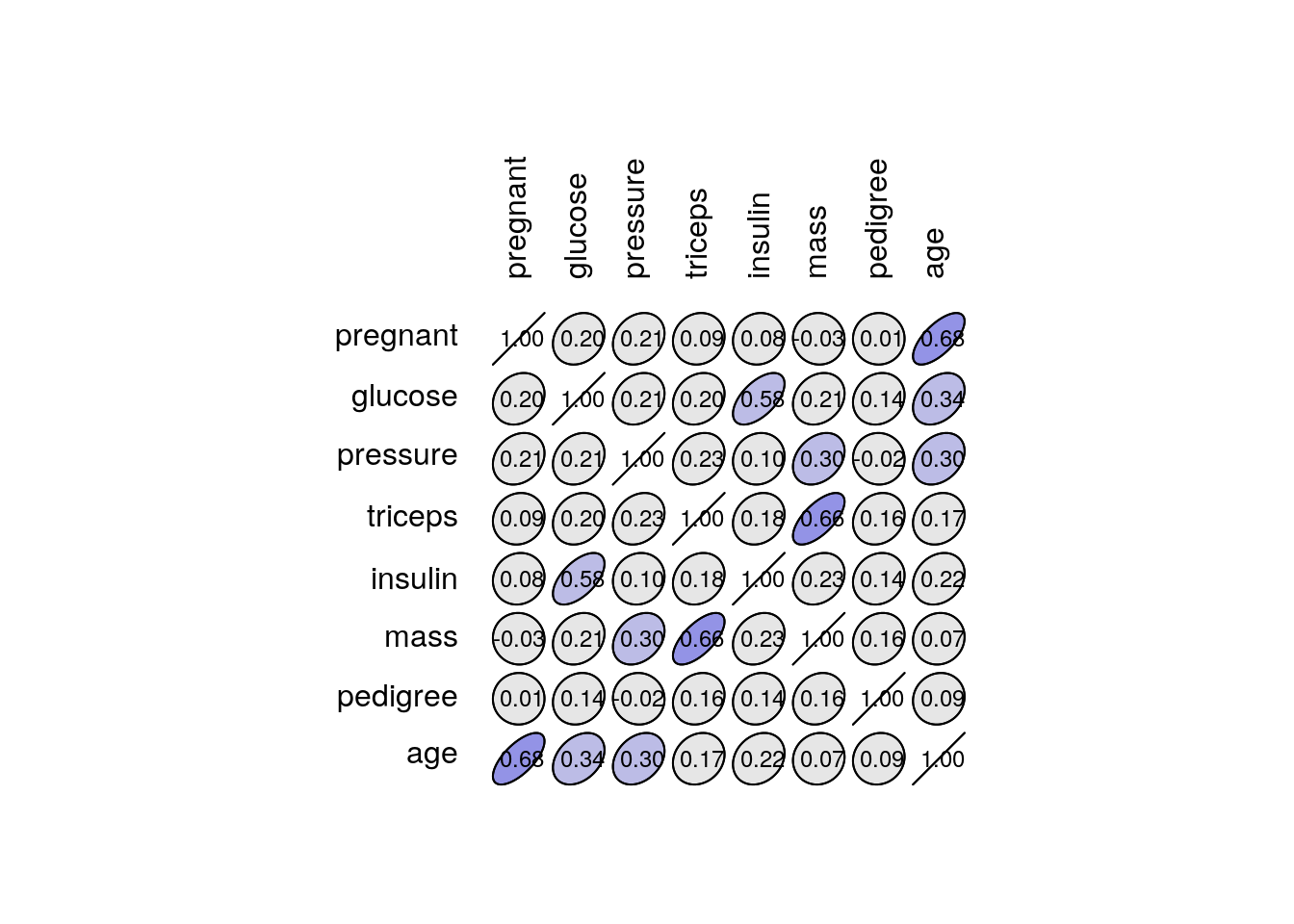
Nous observons que triceps est le plus fortement corrélé à mass, ce qui est logique puisqu’il s’agit de deux mesures différentes de l’obésité.
# `geom_smooth()` using formula 'y ~ x'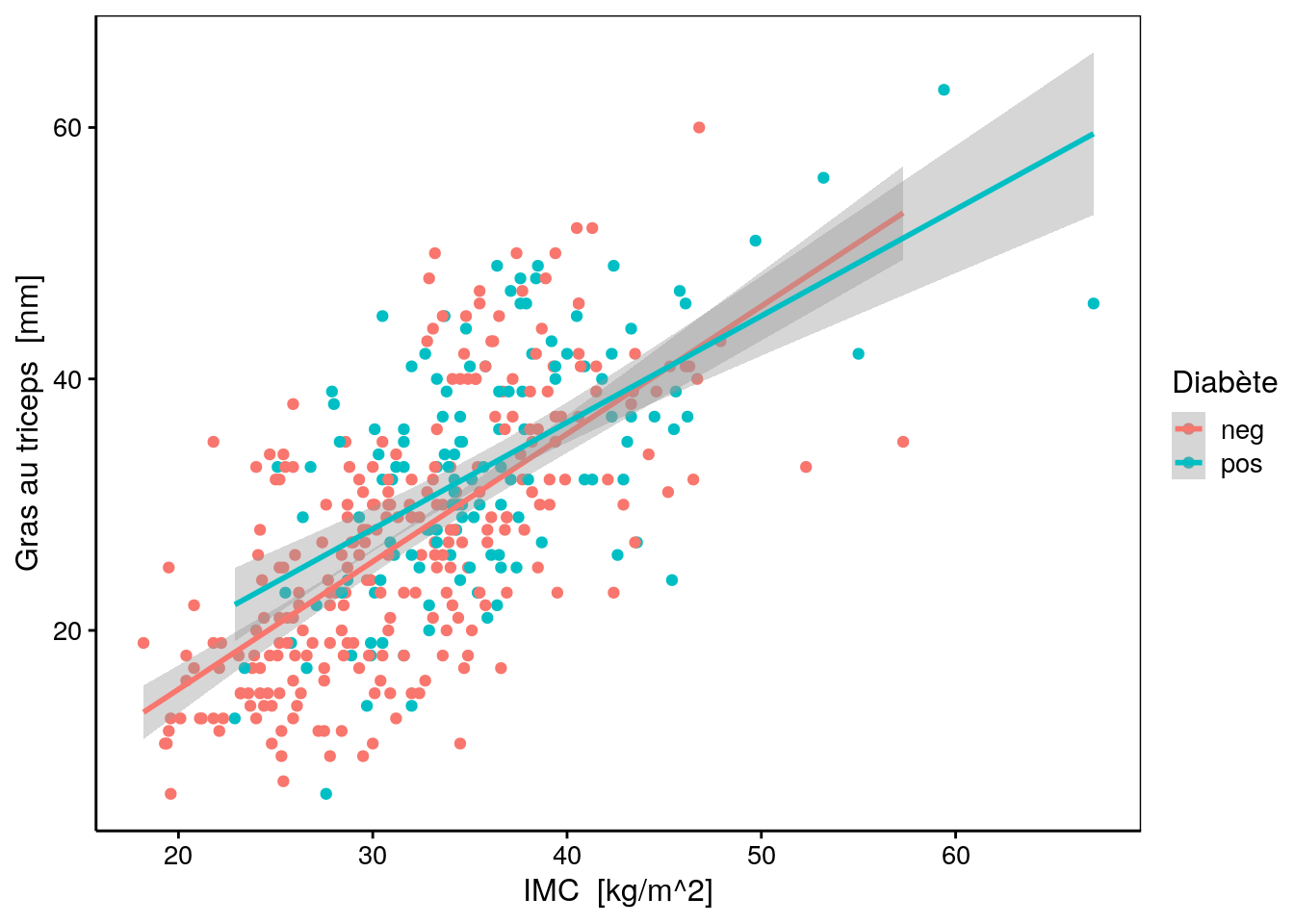
De même, insulin est corrélée à glucose, également deux tests qui étudient le profil de variation du sucre dans le sang et d’une hormone associée.
chart(data = pima1, insulin ~ glucose %color=% diabetes) +
geom_point() +
stat_smooth(method = "lm")# `geom_smooth()` using formula 'y ~ x'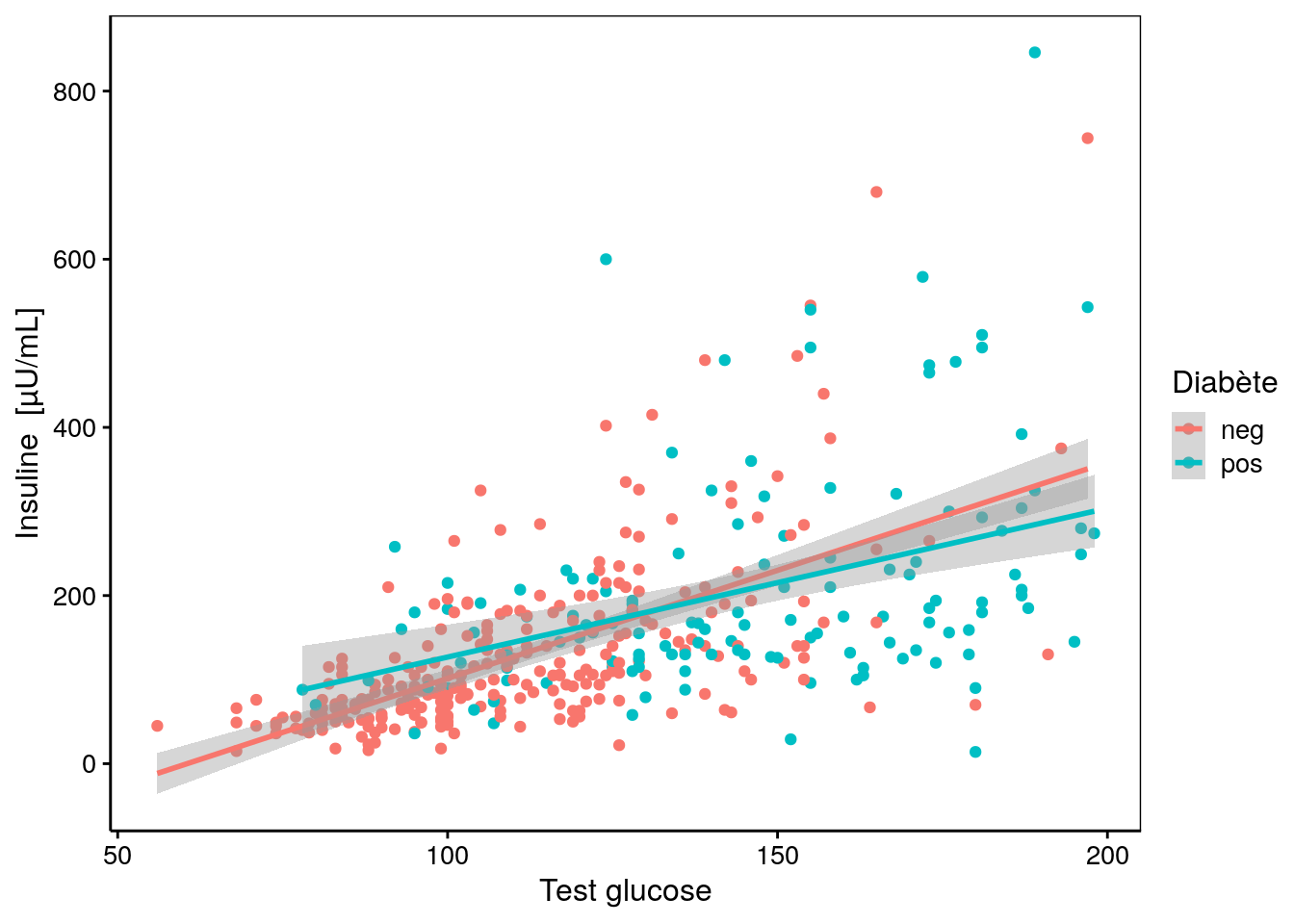
Enfin, et pour être complet, notons aussi que pregnant et age sont également corrélés (0,68). C’est assez logique que les filles moins âgées aient eu moins de grossesses que les autres.
Cependant, les corrélations de Pearson sont moyenne (0,66, 0,58, et 0,68 respectivement) et les nuages de points assez dispersés. Nous pourrions donc nous demander s’il vaut mieux garder plus de données avec moins de variables pour notre apprentissage et test… nous allons créer pima2 sans insulin et triceps et nous comparerons l’analyse faite avec pima1 (plus de variables, moins de cas) et pima2 (moins de variables, plus de cas).
# [1] 724Dans ce second jeu de données nous avons pu tout de même conserver 724 cas. L’ACP que nous avions réalisée l’an dernier sur ces données nous montrait que la variance se répartir à 53% sur deux axes, mais qu’il faut considérer 5 axes pour capturer 90% de cette variance. Ceci suggère, comme la matrice de corrélation, que les différentes variables apportent chacune un information complémentaire. Au final, nous n’observions pas de séparation nette sur le graphique des individus de l’ACP entre la sous-population diabétique et celle qui ne l’est pas. Nous allons reconsidérer la question ici à l’aide de techniques plus spécifiques visant à prédire qui est diabétique ou non en fonction des six (pima1) ou huit (pima2) attributs à disposition, et ce, à l’aide de différent algorithmes de classification supervisée. Commençons par voir ce que cela donnes avec l’ADL que nous connaissons maintenant bien, en utilisation une validation croisée dix fois.
library(mlearning)
pima1_lda <- mlLda(data = pima1, diabetes ~ .)
pima1_lda_conf <- confusion(cvpredict(pima1_lda, cv.k = 10), pima1$diabetes)
plot(pima1_lda_conf)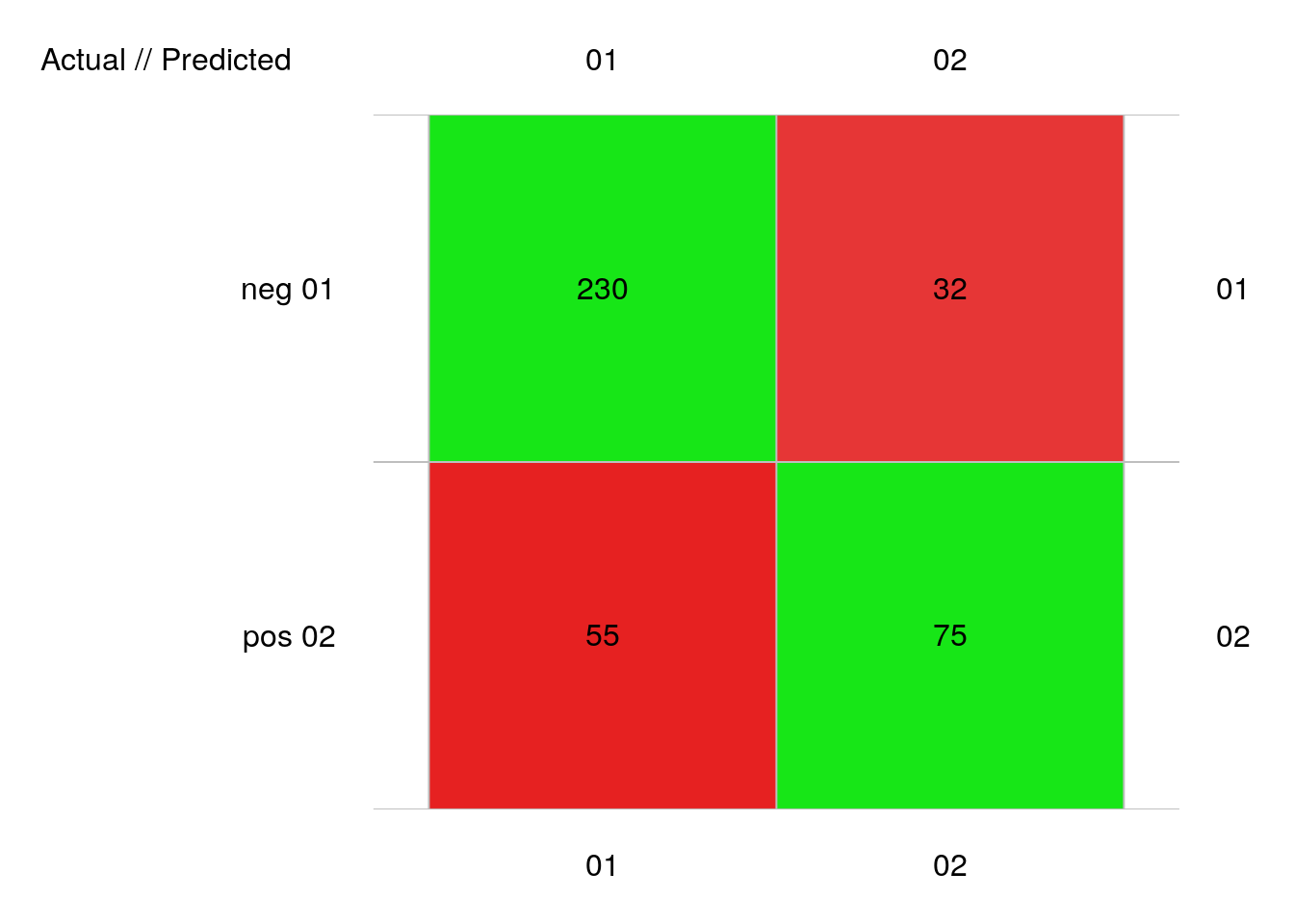
# 392 items classified with 305 true positives (error = 22.2%)
#
# Global statistics on reweighted data:
# Error rate: 22.2%, F(micro-average): 0.74, F(macro-average): 0.737
#
# Fscore Recall Precision Specificity NPV FPR FNR
# neg 0.8409506 0.8778626 0.8070175 0.5769231 0.7009346 0.4230769 0.1221374
# pos 0.6329114 0.5769231 0.7009346 0.8778626 0.8070175 0.1221374 0.4230769
# FDR FOR LRPT LRNT LRPS LRNS BalAcc
# neg 0.1929825 0.2990654 2.074948 0.2117048 2.698465 0.2753216 0.7273928
# pos 0.2990654 0.1929825 4.723558 0.4819398 3.632116 0.3705811 0.7273928
# MCC Chisq Bray Auto Manu A_M TP FP FN TN
# neg 0.4806343 90.55566 0.02933673 285 262 23 230 55 32 75
# pos 0.4806343 90.55566 0.02933673 107 130 -23 75 32 55 230Et avec pima2, cela donne :
pima2_lda <- mlLda(data = pima2, diabetes ~ .)
pima2_lda_conf <- confusion(cvpredict(pima2_lda, cv.k = 10), pima2$diabetes)
plot(pima2_lda_conf)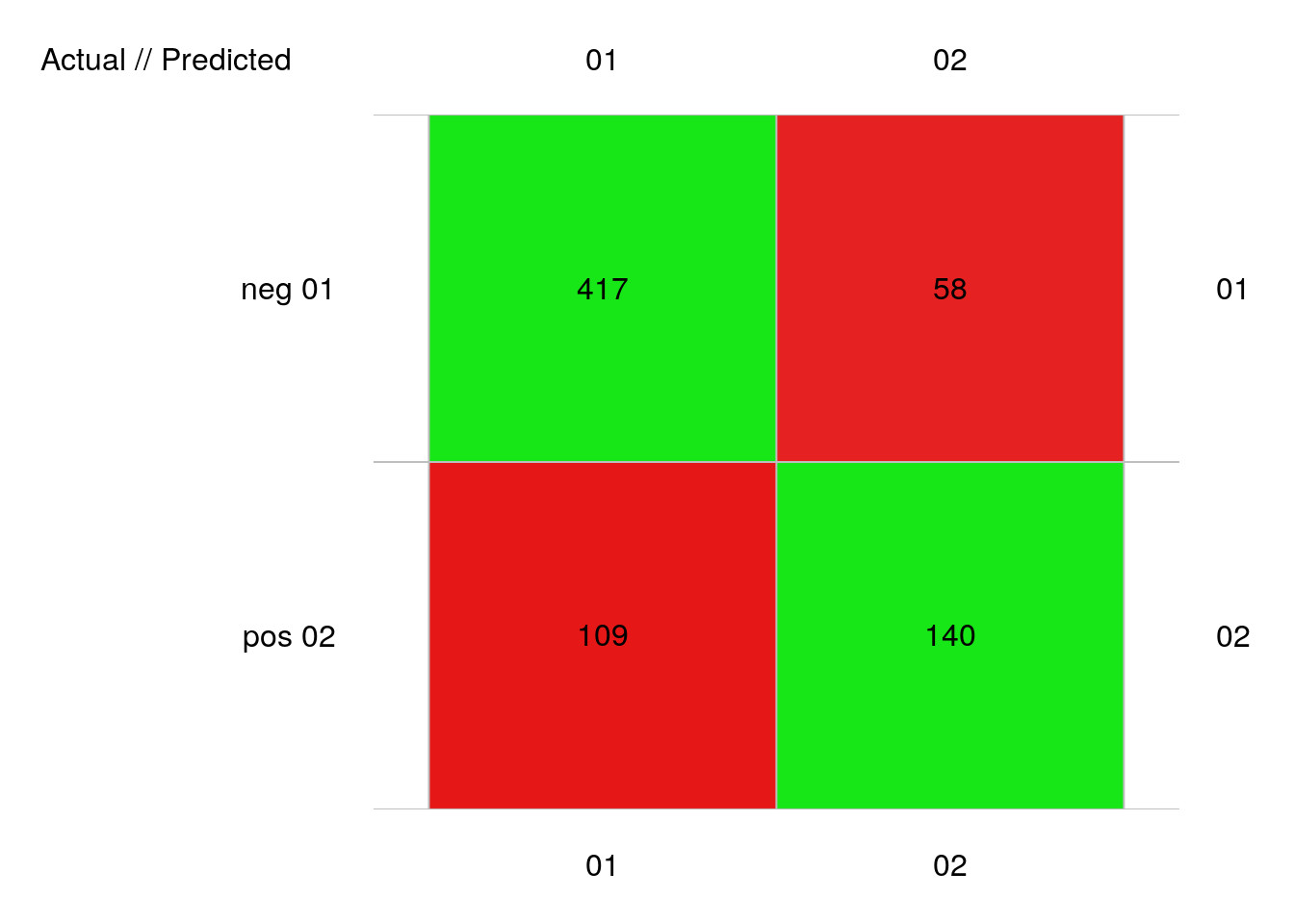
# 724 items classified with 557 true positives (error = 23.1%)
#
# Global statistics on reweighted data:
# Error rate: 23.1%, F(micro-average): 0.735, F(macro-average): 0.73
#
# Fscore Recall Precision Specificity NPV FPR FNR
# neg 0.8331668 0.8778947 0.7927757 0.5622490 0.7070707 0.4377510 0.1221053
# pos 0.6263982 0.5622490 0.7070707 0.8778947 0.7927757 0.1221053 0.4377510
# FDR FOR LRPT LRNT LRPS LRNS BalAcc
# neg 0.2072243 0.2929293 2.005466 0.2171729 2.706372 0.2930744 0.7200719
# pos 0.2929293 0.2072243 4.604625 0.4986372 3.412103 0.3694983 0.7200719
# MCC Chisq Bray Auto Manu A_M TP FP FN TN
# neg 0.4690461 159.2831 0.03522099 526 475 51 417 109 58 140
# pos 0.4690461 159.2831 0.03522099 198 249 -51 140 58 109 417Nous avons 22% d’erreur avec pima1 et 23% d’erreur avec pima2. Ces résultats se tiennent dans le cas présent. Ce n’est évidemment pas toujours le même résultat. Nous allons voir ce que cela donne avec d’autres algorithmes de classification dans la suite.