1.2 Régression linéaire simple
Nous allons découvrir les bases de la régression linéaire de façon intuitive. Nous utilisons le jeu de données trees qui rassemble la mesure du diamètre, de la hauteur et du volume de bois de cerisiers noirs Prunus serotina Ehrh.
L’objectif ici est de pouvoir prédire le volume de bois que ces arbres pourront produire si on les abattait (mais avant de le faire). Le jeu de donnée correspond à 31 arbres de différentes tailles abattus et dont il est donc possible de mesurer le volume de bois.
Nous chargeons les packages R relatifs à SciViews à l’aide de SciViews::R. Nous précisons que nous voulons aussi les packages de la section "model" pour la modélisation, ainsi que la langue par défaut en français.
Ensuite, nous importons le jeu de données trees depuis le package {datasets} à l’aide de la fonction read() comme nous en avons l’habitude.
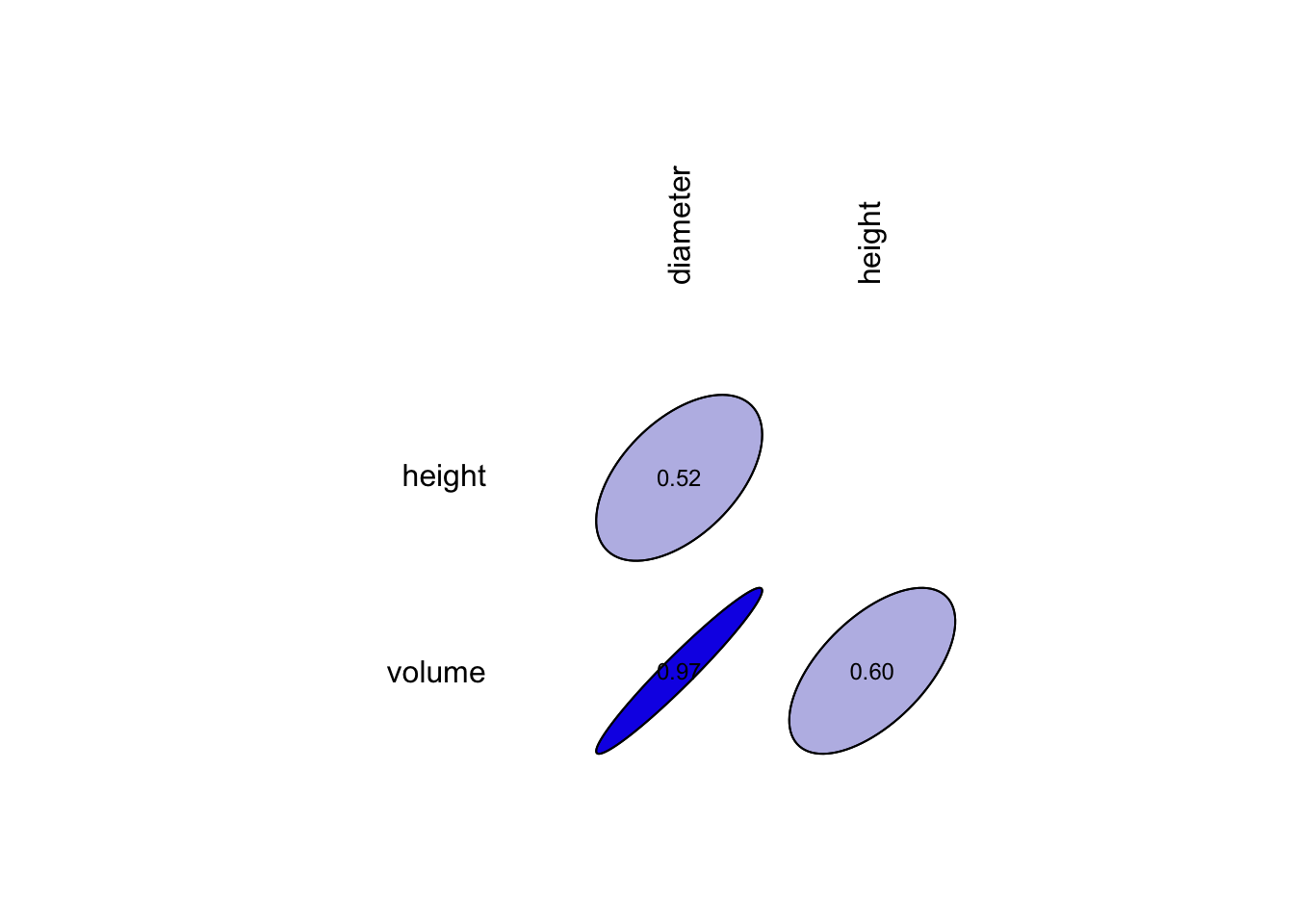
Rappelons-nous que dans le module 12 du cours de science des données 1, nous avons étudié l’association de deux variables quantitatives (ou numériques). Nous utilisons donc une matrice de corrélation afin de mettre en évidence la corrélation entre nos trois variables qui composent le jeu de donnée trees.
La fonction correlation() nous renvoie une matrice de corrélation avec l’indice de Pearson (corrélation linéaire) par défaut. C’est précisément ce coefficient qui nous intéresse dans le cadre d’une régression linéaire comme description préalable des données autant que pour servir de premier guide dans le choix des variables les plus pertinentes pour notre modèle. Nous utilisons ici tabularise() ensuite pour obtenir un tableau formaté dans notre rapport. Cela ne serait pas nécessaire si nous souhaitons seulement imprimer la matrice de corrélation à la console R.
Matrice de coefficients de corrélation de Pearson r | |||
|---|---|---|---|
| diameter | height | volume |
diameter | 1.000 | 0.519 | 0.967 |
height | 0.519 | 1.000 | 0.597 |
volume | 0.967 | 0.597 | 1.000 |
Nous pouvons également observer cette matrice sous la forme d’un graphique.
À vous de jouer !
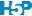
Cependant, n’oubliez pas qu’il est indispensable de visualiser le nuage de points pour ne pas tomber dans le piège mis en avant par le jeu de données artificiel appelé “quartet d’Anscombe” qui montre très bien comment des données très différentes peuvent avoir une même moyenne, une même variance et un même coefficient de corrélation. Un graphique de type matrice de nuages de points est tout indiqué ici.
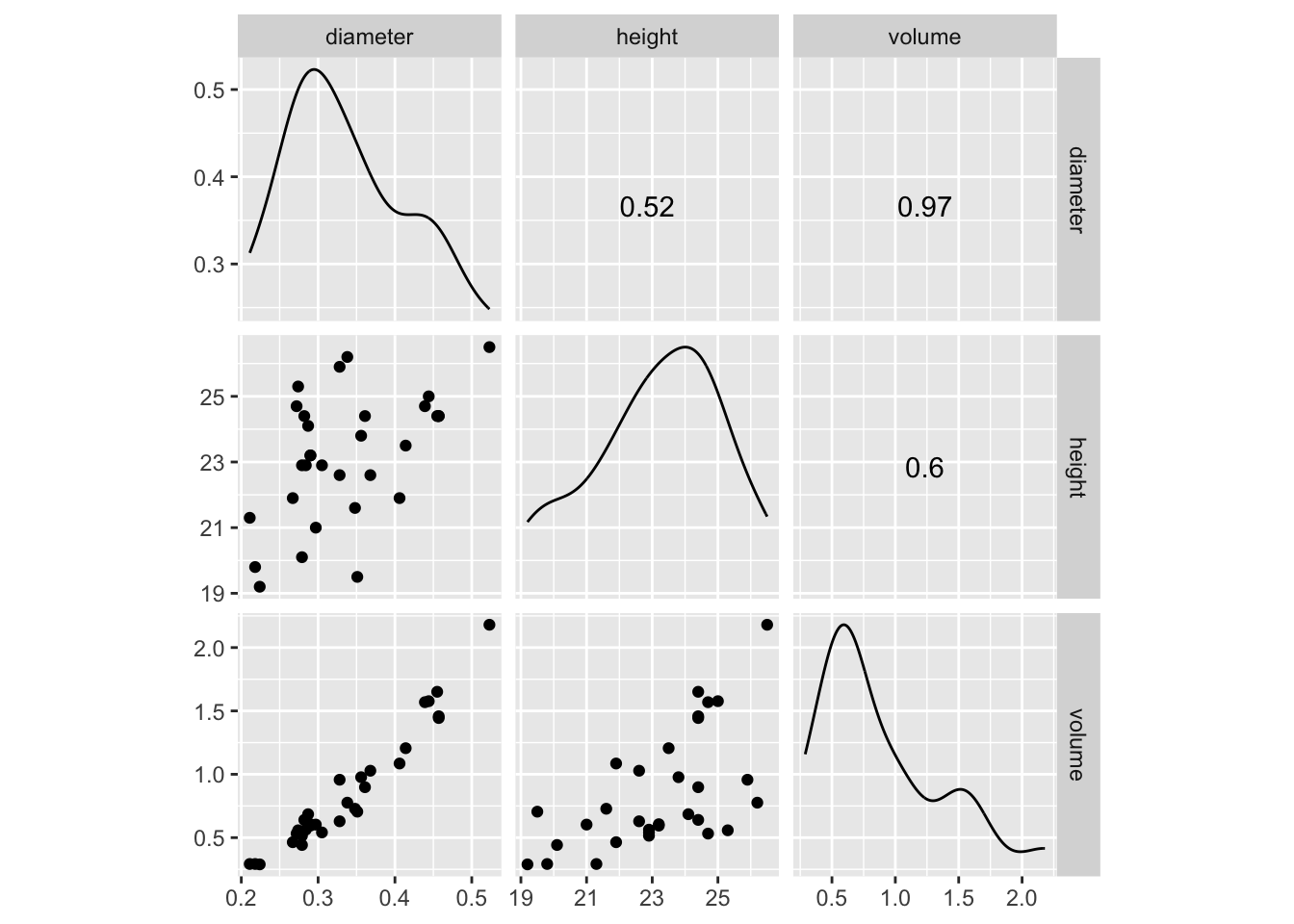
Nous observons une plus forte corrélation linéaire entre le volume et le diamètre. Intéressons-nous à cette dernière association.
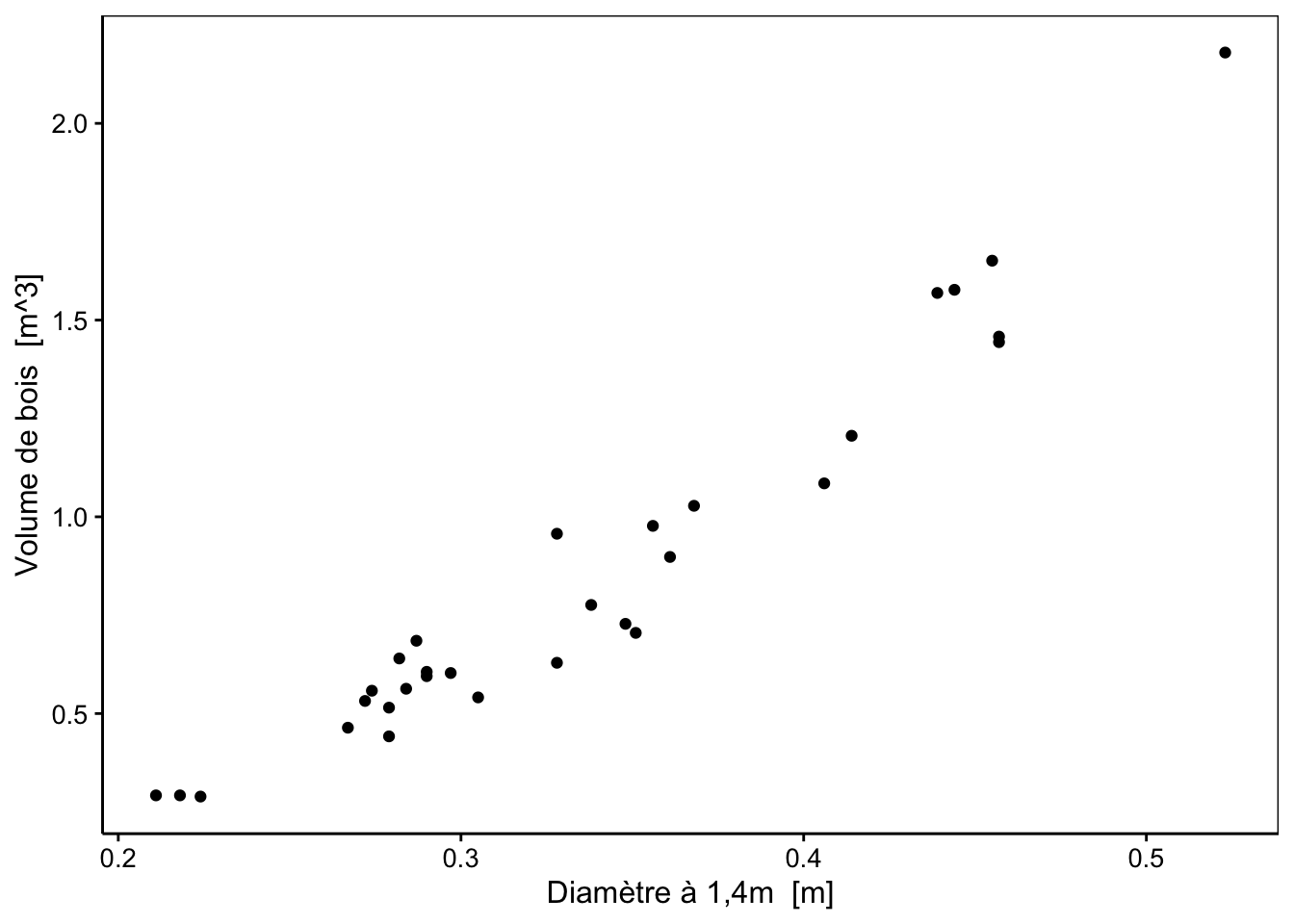
Si vous deviez ajouter une droite permettant de représenter au mieux les données, où est-ce que vous la placeriez ? Pour rappel, une droite respecte l’équation mathématique suivante :
\[y = a + b \ x\]
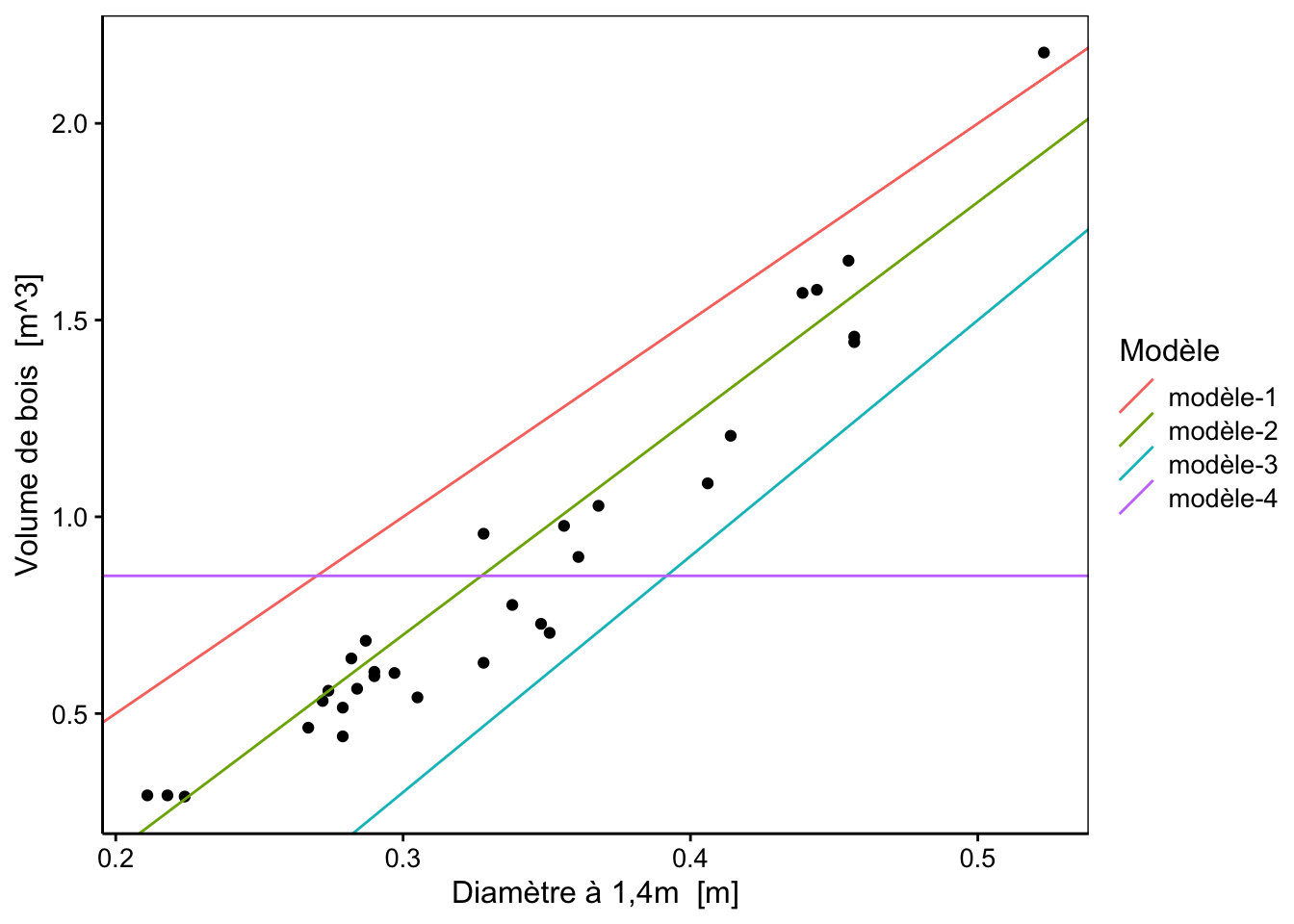
dont \(a\) est l’ordonnée à l’origine (intercept en anglais) et \(b\) la pente (slope en anglais). Voici quatre droites différentes candidates pour représenter au mieux les observations. Quelle est la meilleure d’entre elles selon vous ? Comment pourrions-nous le quantifier ?

1.2.1 Quantifier l’ajustement d’un modèle
Note : Cette partie est assez longue. Elle est utile car elle vous permet de bien comprendre le mécanisme et la logique de la régression linéaire. Par contre, c’est la suite (à partir de “La fonction lm()”) qui est véritablement importante pour bien être capable de réaliser une régression linéaire simple dans R et la diagnostiquer. Par conséquent, ne perdez pas trop de temps dans la présente partie, lisez-là attentivement et effectuez les exercices, mais conservez votre énergie mentale pour la suite !
Nous voulons identifier la meilleure droite de régression, c’est-à-dire la droite le plus proche de nos données. Nous avons besoin d’une règle qui va nous permettre de quantifier la distance de nos observations à notre modèle. La “valeur” qui quantifie cela s’appelle une métrique. La meilleure droite sera alors celle qui minimise cette métrique.
Décomposons le problème étape par étape et intéressons nous au modèle-1 (droite en rouge sur le graphique précédent).
- Nous allons calculer les valeurs de \(y_i\) prédites par le modèle que nous noterons par convention \(\hat y_i\) (prononcez “y chapeau” ou “y hat” en anglais) pour chaque observation \(i\), avec \(i\) allant de 1 à \(N\), la taille de l’échantillon. Nous allons utiliser un petit bout de code R pour faire ce calcul. Il n’est pas indispensable de le comprendre pour pouvoir suivre la suite du raisonnement. Considérez seulement que nous définissons une *fonction personnalisée**
model_predict()qui effectue ce calcul pour nous. Nous l’utilisons ensuite pour calculer les \(\hat{y_i}\) du modèle 1 rouge qui a une ordonnée à l’origine de -0.5 et une pente de 5.
# Calculer la valeur de y pour chaque valeur de x suivant le modèle souhaité
# Création de notre fonction
model_predict <- function(x, intercept, slope)
as.numeric(intercept + slope * x)
# Application de notre fonction
yhat <- model_predict(x = trees$diameter, intercept = -0.5, slope = 5)
yhat# [1] 0.555 0.590 0.620 0.835 0.860 0.870 0.895 0.895 0.910 0.920 0.935 0.950
# [13] 0.950 0.985 1.025 1.140 1.140 1.190 1.240 1.255 1.280 1.305 1.340 1.530
# [25] 1.570 1.695 1.720 1.775 1.785 1.785 2.115- Ensuite, nous calculons la distance entre les observations \(y_i\) et les prédictions par notre modèle \(\hat y_i\), soit \((y_i - \hat y_i)\).
Ces distances sont appelées les résidus du modèle et sont notées \(\epsilon_i\) (epsilon). Nous pouvons, premièrement, visualiser ces résidus graphiquement (ici en rouge par rapport à modèle-1) :
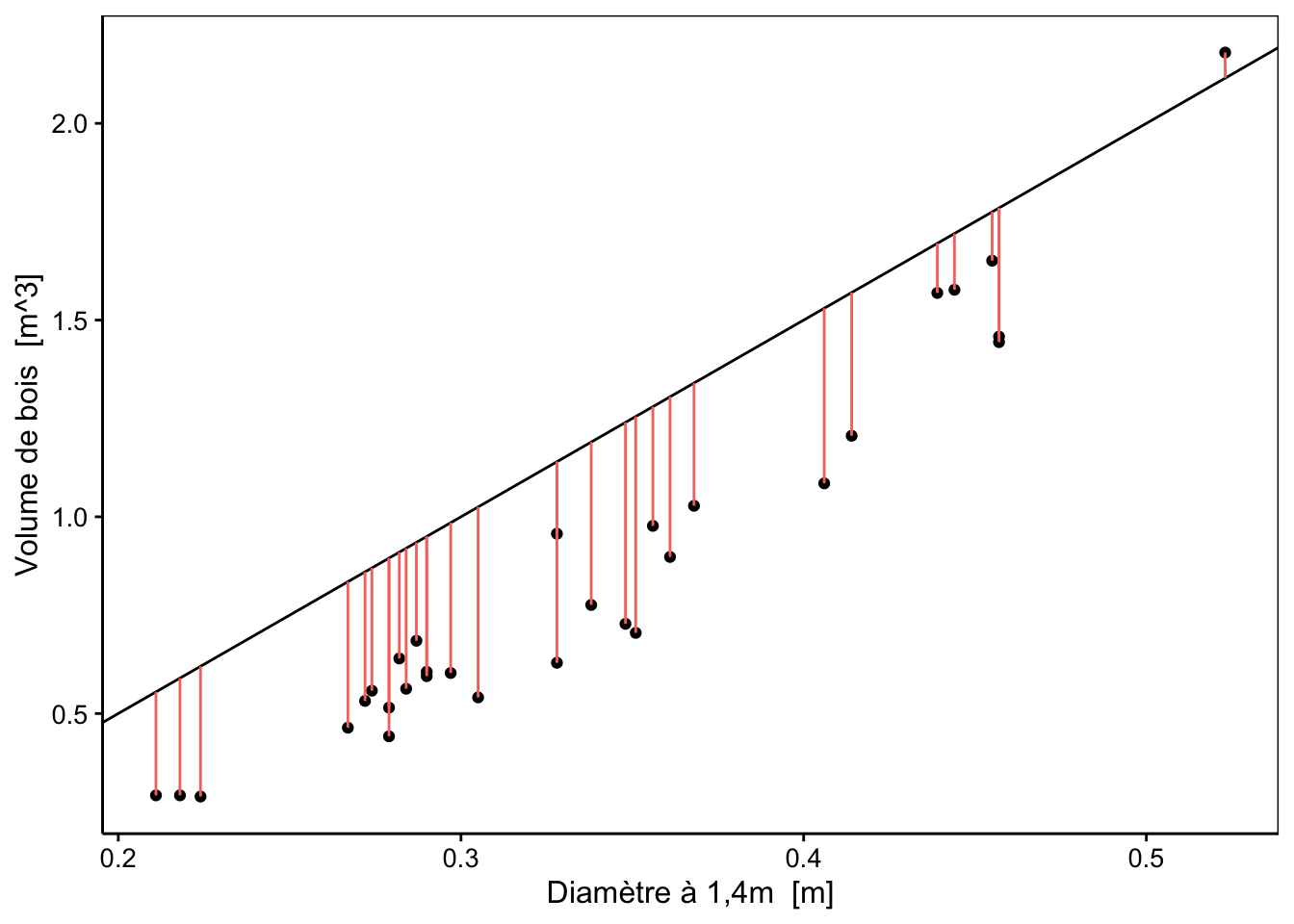
Notez que ces résidus représentent la distance des observations à la droite et donc, quantifient la part non capturée par le modèle de la variation de \(y_i\). en fonction de \(x_i\) pour chaque observation \(i\).. Nous pouvons ensuite facilement calculer leurs valeurs :
# [1] -0.263 -0.298 -0.331 -0.371 -0.328 -0.312 -0.453 -0.380 -0.270 -0.357
# [11] -0.250 -0.355 -0.344 -0.382 -0.484 -0.511 -0.183 -0.414 -0.512 -0.550
# [21] -0.303 -0.407 -0.312 -0.445 -0.364 -0.126 -0.143 -0.124 -0.327 -0.341
# [31] 0.065
# attr(,"label")
# [1] "Volume de bois"
# attr(,"units")
# [1] "m^3"- Il nous faut maintenant définir une règle pour obtenir une valeur unique qui résume l’ensemble des distances de nos observations par rapport aux prédictions du modèle. Une première idée serait de sommer nos résidus :
# [1] -10.175L’idée est ici que, lorsque cette somme tend vers zéro, cela signifierait que les résidus tendent aussi vers zéro dans l’ensemble. Donc cela voudrait dire que notre modèle décrit les données de manière optimale. Appliquons ces calculs sur nos quatre modèles afin de les comparer… Le modèle pour lequel notre métrique serait minimale serait alors considéré comme le meilleur.
modèle.1 | modèle.2 | modèle.3 | modèle.4 |
|---|---|---|---|
-10.175 | -1.441 | 10.393 | 0.135 |
Selon notre méthode, le modèle 4 est considéré comme le plus approprié parce que la valeur de notre métrique est la plus faible en valeur absolue. Qu’en pensez-vous ?
À vous de jouer !
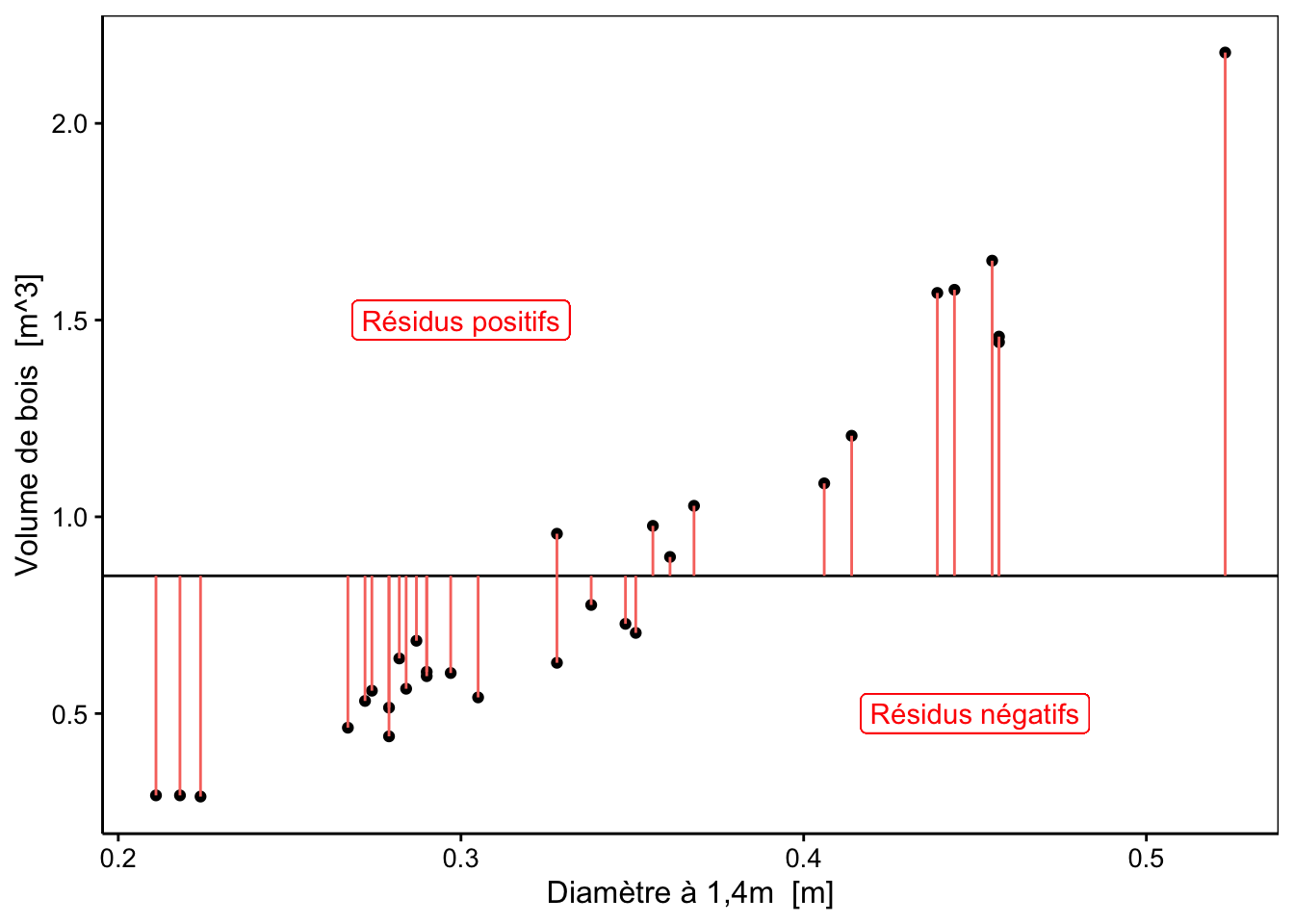
Ainsi avec notre première méthode naïve de somme des résidus, il suffit d’avoir autant de résidus positifs que négatifs pour avoir un résultat proche de zéro pour la métrique. Mais cela n’implique pas que les observations soient proches de la droite pour autant. Avez-vous une autre idée que de sommer les résidus ?
- Sommer le carré des résidus aurait des propriétés intéressantes, car d’une part les carrés de nombres positifs et négatifs sont tous positifs, et d’autre part, plus une observation est éloignée plus sa distance au carré pèse fortement dans la somme3.
Nous obtenons les résultats suivants :
modèle.1 | modèle.2 | modèle.3 | modèle.4 |
|---|---|---|---|
3.842211 | 0.4931095 | 3.929195 | 6.498511 |
- Sommer les valeurs absolues des résidus mène également à des contributions toutes positives, mais sans pénaliser outre mesure les observations les plus éloignées.
Voici ce que cela donne :
modèle.1 | modèle.2 | modèle.3 | modèle.4 |
|---|---|---|---|
10.305 | 3.186 | 10.393 | 11.525 |
Nous avons découvert deux métriques intéressantes pour quantifier la distance de notre droite par rapport à nos observations. En effet, dans les deux cas, la valeur minimale est obtenue pour le modèle-2 (en vert sur le graphique) qui est aussi visuellement le meilleur des quatre.
La méthode utilisant les carrés des résidus s’appelle une régression par les moindres carrés. Notre objectif est donc de trouver les meilleures valeurs des paramètres \(a\) et \(b\) de la droite pour minimiser cette métrique. Cette approche est la plus utilisée.
La méthode utilisant la somme de la valeur absolue des résidus est également intéressante (et elle est d’ailleurs préférable en présence de valeurs extrêmes potentiellement suspectes). Elle s’apppelle régression par la médiane ou régression par les moindres valeurs absolues des résidus, un cas particulier de la régression quantile, une technique intéressante dans le cas de non Normalité des résidus et/ou de présence de valeurs extrêmes suspectes.La fonction qui sert à calculer notre métrique dépend des variables \(x\) et \(y\) et des paramètres du modèle \(a\) et \(b\). On l’appelle la fonction objective. Pour la régression par les moindres carrés, la fonction objective est :
\[f_{obj}(x_i, y_i, a, b) = \sum_{i = 1}^n \epsilon_i^2 = \sum_{i = 1}^n (y_i - \hat y_i)^2 = \sum_{i = 1}^n [y_i - (a + b \ x_i)]^2\] Pour la régression par les médianes, elle est :
\[f_{obj}(x_i, y_i, a, b) = \sum_{i = 1}^n |\epsilon_i| = \sum_{i = 1}^n |y_i - \hat y_i| = \sum_{i = 1}^n |y_i - (a + b \ x_i)|\]
À vous de jouer !
Attention : dans l’application ci-dessous, le nom des paramètres est inversé par rapport au texte. La pente est a et l’ordonnée à l’origine est b. Ce sera corrigé dans la prochaine version de l’app.
 Cliquez pour lancer ou exécutez dans RStudio
Cliquez pour lancer ou exécutez dans RStudio BioDataScience2::run_app("B01Sa_reglin").
Nous pouvons nous demander si notre modèle 2 qui est la meilleure droite parmi nos quatre modèles est le meilleur modèle possible dans l’absolu. En réalité, le meilleur modèle est celui qui minimise la fonction objective, donc pour la régression par les moindres carrés (par la suite, nous ne traiterons plus que de ce cas) :
\[\min f_{obj}(x_i, y_i, a, b) = \min \sum_{i = 1}^n [y_i - (a + b \ x_i)]^2\] Pour ce faire, nous allons devoir définir une technique d’optimisation qui nous permette de déterminer quelle est la droite qui minimise notre fonction objective. Imaginons que nous n’avons pas quatre, mais 5000 modèles linéaires avec des pentes et des ordonnées à l’origine différentes. Quelle est la meilleure droite ?
set.seed(34643)
models1 <- dtx(
intercept = runif(5000, -5, 4),
slope = runif(5000, -5, 15))
chart(data = trees, volume ~ diameter) +
geom_point() +
geom_abline(aes(intercept = intercept, slope = slope),
data = models1, alpha = 1/6) 
Nous voyons sur le graphique qu’un grand nombre de droites différentes sont testées et qu’elles couvrent toute la surface du graphique explorant ainsi un grand nombre de possibilités, mais nous ne distinguons pas grand-chose de plus. Cependant, sur ces 5000 modèles, nous pouvons maintenant calculer la valeur de notre fonction objective et ensuite déterminer quel est le meilleur d’entre eux en retenant celui qui la minimise (ici une fois de plus, il n’est pas indispensable de comprendre le détail du code utilisé pour pouvoir suivre le raisonnement).
# Fonction de calcul de la somme des carrés des résidus (fonction objective)
f_obj <- function(x, y, intercept, slope) {
ybar <- x * slope + intercept
resid <- y - ybar
sum(resid^2)
}
# Test de la fonction
#f_obj(x = trees$diameter, y = trees$volume, intercept = 0.85, slope = 0)
# Fonction adaptée pour être employée avec purrr:map2_dbl(),
# une fonction qui permet de distribuer le calcul sur tous nos modèles
f_obj_trees <- function(intercept, slope) {
f_obj(x = trees$diameter, y = trees$volume,
intercept = intercept, slope = slope)
}
models1 <- smutate(models1, fobj = purrr::map2_dbl(intercept, slope, f_obj_trees))Si nous réalisons un graphique de valeurs d’ordonnées à l’origine, de pentes et de la valeur de la fonction objective en couleur, nous obtenons le graphique suivant :
p <- chart(data = models1, slope ~ intercept %col=% fobj) +
geom_point() +
geom_point(data = sfilter(models1, frank(fobj) <= 10),
shape = 1, color = 'red', size = 3) +
labs(x = "Ordonnée à l'origine", y = "Pente", color = "f_obj") +
scale_color_viridis_c(direction = -1)
plotly::ggplotly(p)Les dix valeurs les plus faibles sont mises en évidence sur le graphique par des cercles rouges. Le modèle optimal que nous recherchons se trouve dans cette région.
Nous pouvons afficher les 10 meilleurs modèles sur notre graphique :
chart(data = trees, volume ~ diameter) +
geom_abline(data = best_models,
aes(slope = slope, intercept = intercept, color = fobj), alpha = 3/4) +
geom_point() +
labs(color = "F(obj)") +
scale_color_viridis_c(direction = -1)
En résumé, nous avons maintenant une fonction objective qui quantifie la qualité d’ajustement de la droite à tous nos points. La meilleure droite est celle qui minimise cette fonction objective. Il nous manque encore une technique pour trouver rapidement et efficacement la droite qui répond à ce critère.
Dans le cas particulier de la régression linéaire par les moindres carrés, les mathématiciens nous expliquent que la solution à notre problème s’obtient très facilement par calcul. En effet, les paramètres \(a\) et \(b\) de la droite que nous recherchons sont directement calculables comme suit :
\[b = \frac{\operatorname{cov}_{x, y}}{\operatorname{var}_x} \ \ \ \textrm{et} \ \ \ a = \bar y - b \ \bar x\]
où \(\bar x\) et \(\bar y\) sont les moyennes pour les deux variables \(x\) et \(y\), \(\operatorname{cov}\) est la covariance et \(\operatorname{var}\) est la variance.
Dans R, vous vous doutez bien que nous n’allons pas refaire tous ces calculs à chaque fois que nous voulons ajuster une droite dans un nuage de points. La fonction lm() réalise, en effet, ce calcul pour nous :
#
# Call:
# lm(formula = volume ~ diameter, data = trees)
#
# Coefficients:
# (Intercept) diameter
# -1.047 5.652Nous pouvons reporter ces valeurs sur notre graphique afin d’observer ce résultat par rapport à nos modèles choisis au hasard. Nous avons en rouge la droite calculée par lm().
chart(data = trees, volume ~ diameter) +
geom_abline(data = best_models,
aes(slope = slope, intercept = intercept, color = fobj), alpha = 3/4) +
geom_point() +
geom_abline(
aes(intercept = trees_lm$coefficients[1], slope = trees_lm$coefficients[2]),
color = "red", linewidth = 1.5) +
labs( color = "F(obj)") +
scale_color_viridis_c(direction = -1)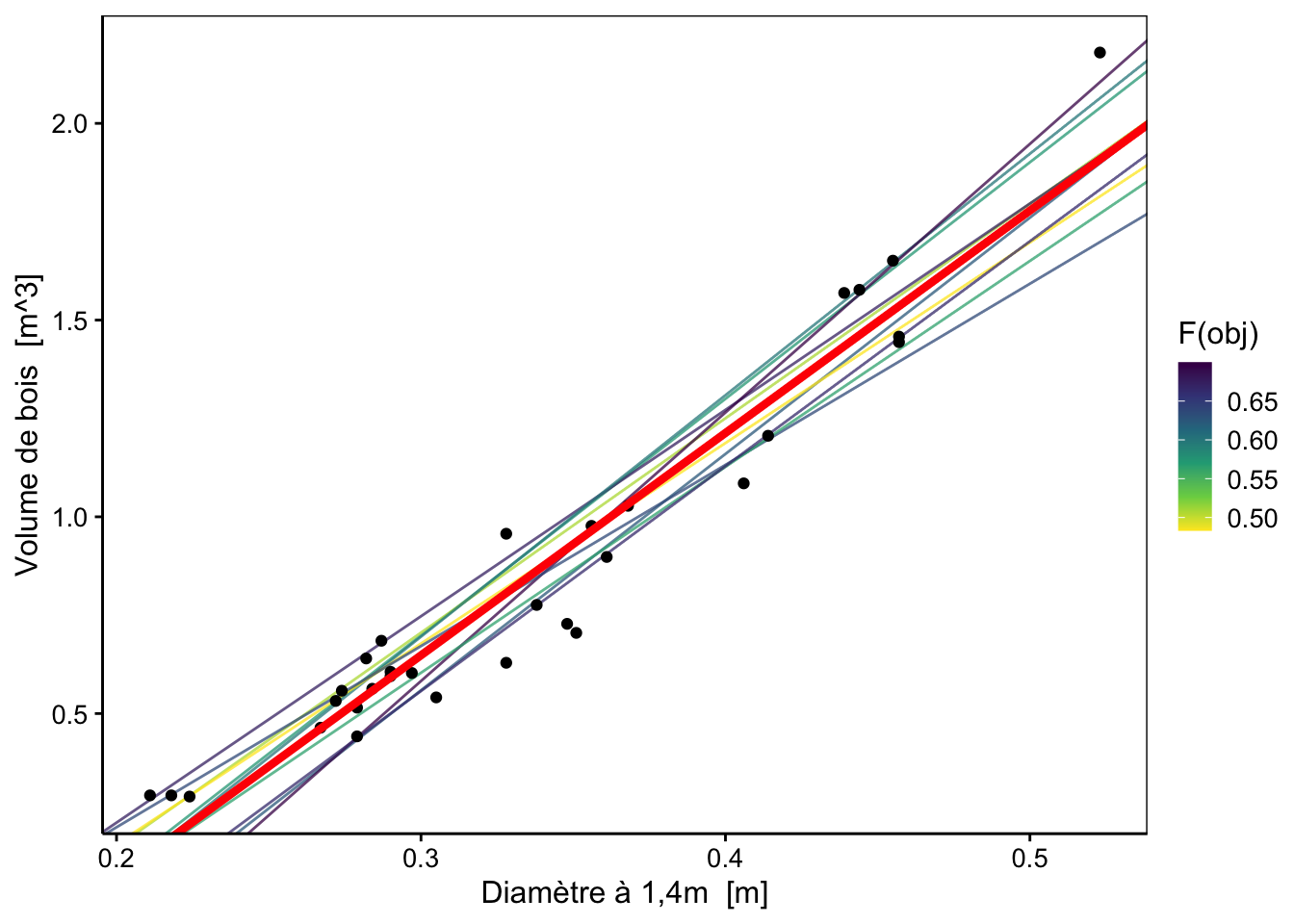
1.2.2 La fonction lm()
Suite à notre découverte de manière intuitive de la régression linéaire simple, nous pouvons récapituler quelques points clés :
- Une droite correspond à l’équation mathématique suivante :
\[y = a + b \ x\]
dont \(a\) est l’ordonnée à l’origine (intercept en anglais) et \(b\) est la pente (slope en anglais), tous deux les paramètres du modèle, alors que \(x\) et \(y\) en sont les variables. En statistique, on a l’habitude de représenter les paramètres généraux avec des lettres grecques pour réserver les lettres latines aux valeurs de l’échantillon observé. Nous utiliserons donc plutôt l’équation :
\[y = \alpha + \beta \ x\]
où \(\alpha\) et \(\beta\) correspondent respectivement aux paramètres \(a\) et \(b\) utilisés jusqu’ici.
- Les distances entre les valeurs observées \(y_i\) et les valeurs prédites \(\hat y_i\) se nomment les résidus (\(\epsilon_i\)) et se mesurent toujours parallèlement à l’axe des ordonnées \(y\). Cela revient à considérer que toute l’erreur du modèle se situe sur \(y\) et non sur \(x\). Cela donne l’équation complète de notre modèle statistique :
\[y_i = \alpha + \beta \ x_i + \epsilon_i\]
avec \(y_i\) qui sont les valeurs mesurées pour le point i sur l’axe y, \(\alpha\) est l’ordonnée à l’origine, \(x_i\) sont les valeurs mesurées pour le point i sur l’axe x, \(\beta\) est la pente et \(\epsilon_i\) sont les résidus (partie non expliquée par le modèle). L’équation s’écrit aussi souvent de manière simplifiée en se débarrassant des indices i et en considérant \(Y = y_i\) et \(X = x_i\) :
\[Y = \alpha + \beta \ X + \epsilon\]
On peut montrer –nous ne le ferons pas ici pour limiter les développements mathématiques– que le choix des moindres carrés des résidus comme fonction objective s’accompagne d’une distribution Normale de nos résidus centrée autour de zéro et avec une variance \(\sigma^2\) constante :
\[\epsilon_i \approx N(0, \sigma^2)\]
Dans le cas de la régression linéaire simple, la meilleure droite s’obtient très facilement. En effet, l’estimateur de la pente, noté \(\hat\beta\) est \(\hat \beta = \operatorname{cov}_{x, y}/\operatorname{var}_x\) et l’estimateur de l’ordonnée à l’origine noté \(\hat \alpha\) est \(\hat \alpha = \bar y - \hat \beta \ \bar x\).
La fonction
lm()permet de faire ce calcul rapidement dans R.
# Régression linéaire
trees_lm <- lm(data = trees, volume ~ diameter)
# Graphique de nos observations et de la droite obtenue avec la fonction lm()
# accompagnée d'une enveloppe de confiance à 95%
chart(trees_lm)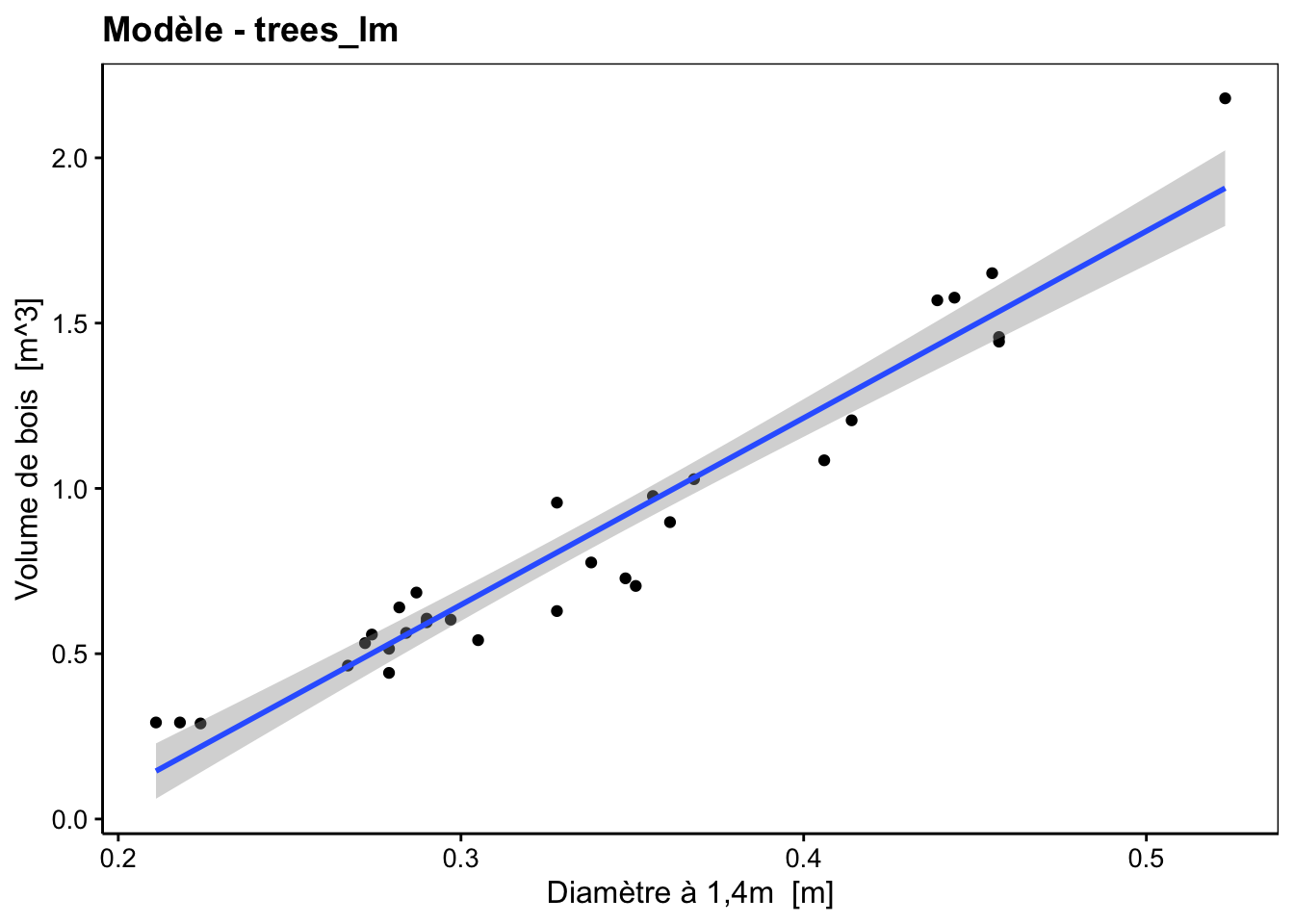
Le graphique chart() par défaut d’un objet lm est le nuage de points auquel la droite de régression est ajoutée ainsi qu’une zone grisée représentant l’enveloppe de confiance à 95%, c’est-à-dire que selon le modèle et si la distribution des résidus est Normale et de variance constante (= homoscédasticité), la droite de régression de la population statistique a 95% de chances de se trouver à l’intérieur de cette enveloppe. Cela permet de visualiser tout de suite la précision avec laquelle l’échantillon permet d’estimer la droite que l’on cherche (sachant que la droite bleue est celle relative à notre échantillon, mais pas celle de la population statistique étudiée, on dit que c’est l’estimateur de la droite de la population). Nous reviendrons à cette représentation et à l’enveloppe de confiance à la fin de ce module.
La fonction lm() crée un objet spécifique qui contient de nombreuses informations pour pouvoir ensuite analyser plus en détail notre modèle linéaire. Afin de déterminer de quel objet il s’agit, vous pouvez utiliser class(). Ainsi, nous voyons que trees_lm est un objet de classe lm :
# [1] "lm"Pour la régression par les moindres carrés (des résidus), le fait de considérer que toute l’erreur porte sur la variable dépendante Y est une décision importante qui a de nombreuses conséquences. Outre un calcul simplifié de la meilleure droite et des contraintes sur la distribution des résidus (qui doit être Normale), nous avons également une asymétrie qui apparait entre les deux variables X et Y.
Ainsi, la régression \(Y = \alpha + \beta \ X\) ne correspond pas à la même droite que \(X = \alpha' + \beta' \ Y'\). En effet, dans le premier cas, l’erreur est entièrement reportée sur Y tandis que dans le second cas, elle est entièrement reportée sur X. C’est pour cela que l’on distingue la variable réponse ou dépendante à la gauche de l’équation de la variable explicative ou indépendante à sa droite.
Décider quel variable est réponse dans l’équation n’est donc pas anodin. Utilisez les critères suivants :
- Si une des deux variables contient beaucoup plus d’erreur et/ou de variabilité inter-individuelle, placez-là à la gauche de l’équation comme variable réponse. En effet, votre modèle sera plus en adéquation avec la réalité, puisque dans le modèle toute l’erreur porte sur cette variable.
- Si une des deux variables est fixée dans une expérience. Par exemple, vous faites grandir différents lots de plantes à des intensités lumineuses différentes, alors la variable intensité lumineuse sera plutôt variable explicative à la droite de l’équation et la mesure de la croissance de vos plantes sera variable réponse dans le terme de gauche.
- Si une variable devra être prédite ultérieurement (quelle sera la croissance de mes plantes pour telle ou telle illumination, dans notre exemple précédent), cette variable à prédire sera plus facile à traiter si elle est variable réponse à la gauche de l’équation.
1.2.3 Résumé avec summary()
Avec summary() nous obtenons un résumé condensé des informations les plus utiles pour chaque objet dans R. Dans le cas de l’objet lm, l’information renvoyée est très utile pour interpréter notre régression linéaire.
#
# Call:
# lm(formula = volume ~ diameter, data = trees)
#
# Residuals:
# Min 1Q Median 3Q Max
# -0.231211 -0.087021 0.003533 0.100594 0.271725
#
# Coefficients:
# Estimate Std. Error t value Pr(>|t|)
# (Intercept) -1.04748 0.09553 -10.96 7.85e-12 ***
# diameter 5.65154 0.27649 20.44 < 2e-16 ***
# ---
# Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#
# Residual standard error: 0.1206 on 29 degrees of freedom
# Multiple R-squared: 0.9351, Adjusted R-squared: 0.9329
# F-statistic: 417.8 on 1 and 29 DF, p-value: < 2.2e-16- Call :
Il s’agit de la formule employée dans la fonction lm(). C’est-à-dire le volume en fonction du diamètre du jeu de données trees.
- Residuals :
La tableau nous fournit un résumé via les cinq nombres de l’ensemble des résidus (que vous pouvez par ailleurs récupérer à partir de residuals(trees_lm)).
# 20 7 12 9 31
# -0.231210947 -0.087020695 0.003532709 0.100594122 0.271724973- Coefficients :
Il s’agit des résultats associés à la pente et à l’ordonnée à l’origine, dont les valeurs estimées des paramètres (Estimate). Les mêmes valeurs peuvent être obtenues à partir de coef(trees_lm) :
# (Intercept) diameter
# -1.047478 5.651535Nous retrouvons également les écarts-types calculés sur ces valeurs (Std. Error) qui donnent une indication de la précision de leur estimation, les valeurs des distributions t de Student sur ces estimations (t value) et enfin les valeurs p (PR(>|t|)) liées à un test t de Student pour déterminer si le paramètre correspondant est significativement différent de zéro (avec \(H_0: p = 0\) et \(H_a: p \neq 0\)).
Pour l’instant, nous nous contenterons d’interpréter et d’utiliser les informations issues de summary() dans sa partie supérieure. Le contenu des trois dernières lignes sera détaillé dans le module suivant.
Comme la sortie de summary() pour un objet lm, bien que très utile, fait brut de décoffrage dans un rapport, vous pouvez utiliser ici aussi tabularise() pour mettre cela en forme :
Modèle linéaire | |||||
|---|---|---|---|---|---|
| |||||
Terme | Valeur estimée | Ecart type | Valeur de t | Valeur de p | |
| -1.05 | 0.0955 | -11.0 | 7.85·10-12 | *** |
| 5.65 | 0.2765 | 20.4 | < 2·10-16 | *** |
0 <= '***' < 0.001 < '**' < 0.01 < '*' < 0.05 | |||||
Etendue des résidus : [-0.2312, 0.2717] | |||||
Ecart type des résidus : 0.1206 pour 29 degrés de liberté | |||||
R2 multiple : 0.9351 - R2 ajusté : 0.9329 | |||||
Statistique F : 417.8 pour 1 et 29 ddl - valeur de p : < 2.22e-16 | |||||
En prime, on a l’équation du modèle (que l’on peut ne pas indiquer avec l’argument equation = FALSE si on préfère). Les variables génériques X et Y dans l’équation sont remplacées par celles du modèle. La notation \(\alpha\) et \(\beta\) pour les paramètres est également respectée. Cela rend le tableau plus lisible.
1.2.4 Paramétrisation du modèle
À partir des données de ce résumé, nous pouvons maintenant paramétrer l’équation de notre modèle :
\[\operatorname{Volume\ de\ bois \ [m^{3}]} = \alpha + \beta_{}(\operatorname{Diamètre\ à\ 1,4m \ [m]}) + \epsilon\]
devient4 :
\[\operatorname{\widehat{Volume\ de\ bois
\ [m^{3}]}} = -1.05 + 5.65(\operatorname{Diamètre\ à\ 1,4m
\ [m]})\] Notez que nous avons laissé tombé \(\epsilon\) ici, ce qui nous oblige à écrire \(\widehat{\mathrm{Volume\ de\ bois\ [m^3]}}\) car \(\widehat{\mathrm{Volume\ de\ bois\ [m^3]}} = \mathrm{Volume\ de\ bois\ [m^3]} - \epsilon\). Vous apprendrez à extraire les équations du modèle de manière automatisée avec SciViews::R dans le learnr ci-dessous.
À vous de jouer !
Effectuez maintenant les exercices du tutoriel B01La_reg_lin (Régression linéaire simple).
BioDataScience2::run("B01La_reg_lin")Utiliser le carré des résidus a aussi d’autres propriétés statistiques intéressantes qui rapprochent ce calcul de la variance (somme de la distance au carré à la moyenne pour une seule variable numérique).↩︎
Lors de la paramétrisation du modèle, pensez à arrondir la valeur des paramètres à un nombre de chiffres significatifs raisonnables. Inutile de garder cinq, ou même trois chiffres derrière la virgule si vous n’avez que quelques dizaines d’observations pour ajuster votre modèle et que les mesures ont été faites avec une précision moyenne, elle-même à deux chiffres, par exemple.↩︎