2.5 Modèle empirique et mécaniste
Un modèle empirique est issu de l’observation. Nous collectons des données et nous cherchons à les prédire via un modèle statistique. L’important ici est d’obtenir une bonne prédiction, c’est-à-dire, une erreur quadratique moyenne du modèle la plus faible possible. Cependant, la forme mathématique du modèle n’a, éventuellement, rien à voir avec le mécanisme qui génère les données. C’est typiquement le cas pour un modèle polynomial d’ordre élevé.
Outre les prédictions pour lesquelles ce modèle est taillé par construction, un modèle linéaire simple ou multiple peut s’avérer également utile pour révéler les relations qui existent entre les variables impliquées… tout en restant très prudent concernant la colinéarité qui peut faire penser que des variables fortement corrélées entre elles aient faussement un pouvoir explicatif.
Dans le cas de trees, nos différents essais suggèrent que le diamètre du tronc pourrait être une variable explicative lorsqu’elle est élevée au carré. La hauteur est également utile, sous forme non transformée.
Un modèle mécaniste vise à expliciter le mécanisme qui est responsable de la relation observée. De tels modèles sont plus courants en physique, voire en chimie qu’en biologie, mais il y en a aussi. Le modèle mécaniste est plus difficile à définir, car il implique une compréhension profonde des mécanismes impliqués. Dans le cas de trees, il ne faut pas être un génie pour se rendre compte que le tronc de l’arbre peut être assimilé, en première approximation, à un cône. Par conséquent, le volume de bois peut être approximé par la formule suivante :
\[volume = \frac{1}{3} \cdot \pi \cdot rayon^2 \cdot hauteur\]
Avec notre diamètre au carré et notre hauteur, nous n’étions pas loin, à ceci près que les deux variables sont additionnées, et non multipliées dans notre modèle. La régression linéaire, et comme nous le verrons au chapitre suivant, le modèle linéaire sont fondamentalement des modèles additifs où les différents termes s’additionnent. Cependant, nous pouvons faire intervenir un terme qui multiplie nos deux variables explicatives par l’intermédiaire des interactions comme pour l’ANOVA à deux facteurs, voir section 11.3 de SDD I. Afin de nous rapprocher de la formule du volume d’un cône, calculons à présent le carré du rayon et ajustons un modèle linéaire multiple avec interactions :
trees <- smutate(trees, radius2 = labelise((diameter/2)^2, "Rayon^2", units = "m^2"))
trees_lm7 <- lm(data = trees, volume ~ radius2 * height) # Forme compacte
# Forme développée, équivalente à la forme compacte
trees_lm7 <- lm(data = trees, volume ~ radius2 + height + radius2:height)
summary(trees_lm7) |>
tabularise()Modèle linéaire | |||||
|---|---|---|---|---|---|
| |||||
Terme | Valeur estimée | Ecart type | Valeur de t | Valeur de p | |
| -0.04178 | 0.3391 | -0.123 | 0.9028 |
|
| -1.25193 | 12.4130 | -0.101 | 0.9204 |
|
| 0.00173 | 0.0145 | 0.119 | 0.9061 |
|
| 1.26682 | 0.5074 | 2.497 | 0.0189 | * |
0 <= '***' < 0.001 < '**' < 0.01 < '*' < 0.05 | |||||
Etendue des résidus : [-0.1398, 0.1208] | |||||
Ecart type des résidus : 0.07299 pour 27 degrés de liberté | |||||
R2 multiple : 0.9779 - R2 ajusté : 0.9754 | |||||
Statistique F : 397.5 pour 3 et 27 ddl - valeur de p : < 2.22e-16 | |||||
Ce modèle est le suivant :
\[\operatorname{\widehat{Volume\ de\ bois \ [m^{3}]}} = -0.042 - 1.25(\operatorname{Rayon^{2} \ [m^{2}]}) + 0.0017(\operatorname{Hauteur \ [m]}) + 1.27(\operatorname{Rayon^{2} \ [m^{2}]} \times \operatorname{Hauteur \ [m]})\]
Or, le résumé du modèle nous indique clairement que \(\alpha\), \(\beta_1\) et \(\beta_2\) sont tous les trois non significativement différents de zéro. Nous pouvons donc simplifier notre modèle à :
\[volume = \beta \cdot radius^2 \cdot height + \epsilon\]
… qui est mathématiquement équivalent à la formule qui calcule le volume d’un cône. Cela donne :
Modèle linéaire | |||||
|---|---|---|---|---|---|
| |||||
Terme | Valeur estimée | Ecart type | Valeur de t | Valeur de p | |
| 1.21 | 0.0157 | 77.4 | < 2·10-16 | *** |
0 <= '***' < 0.001 < '**' < 0.01 < '*' < 0.05 | |||||
Etendue des résidus : [-0.1327, 0.1239] | |||||
Ecart type des résidus : 0.06956 pour 30 degrés de liberté | |||||
R2 multiple : 0.995 - R2 ajusté : 0.9949 | |||||
Statistique F : 5990 pour 1 et 30 ddl - valeur de p : < 2.22e-16 | |||||
# [1] -74.31119# [1] 0.06842465Vous aurez certainement noté un problème dans l’affichage de l’équation. C’est clairement un bug. Actuellement, le code qui crée l’équation du modèle n’est pas capable de le faire lorsque les termes qui apparaissent dans des interactions n’apparaissent pas également sous forme classique dans l’équation. Nous sommes donc ici obligés de fournir le code LaTeX de l’équation dans l’argument equation= de tabularise() pour avoir l’équation correcte (une astuce consiste à examiner et retravailler la sortie de equation(trees_lm7) à la console R) :
trees_lm8 <- lm(data = trees, volume ~ 0*radius2 + 0*height + radius2:height + 0)
summary(trees_lm8) |>
tabularise(equation = "\\operatorname{Volume\\ de\\ bois \\ [m^{3}]} =
\\alpha + \\beta_{}(\\operatorname{Rayon^{2} \\ [m^{2}]} \\times
\\operatorname{Hauteur \\ [m]}) + \\epsilon")Modèle linéaire | |||||
|---|---|---|---|---|---|
| |||||
Terme | Valeur estimée | Ecart type | Valeur de t | Valeur de p | |
radius2:height | 1.21 | 0.0157 | 77.4 | < 2·10-16 | *** |
0 <= '***' < 0.001 < '**' < 0.01 < '*' < 0.05 | |||||
Etendue des résidus : [-0.1327, 0.1239] | |||||
Ecart type des résidus : 0.06956 pour 30 degrés de liberté | |||||
R2 multiple : 0.995 - R2 ajusté : 0.9949 | |||||
Statistique F : 5990 pour 1 et 30 ddl - valeur de p : < 2.22e-16 | |||||
# [1] -74.31119# [1] 0.06842465Avec ce dernier modèle, nous avons une équation très simple pour laquelle un seul paramètre doit être évalué. L’estimateur de ce paramètre vaut 1,21. Il n’est pas très éloigné de la valeur théorique \(1/3 \cdot \pi = 1.05\). Souvenons-nous que le tronc de nos cerisiers noirs a une forme plus complexe qu’un cône et que le diamètre est mesuré à 4 pieds 6 pouces = 1,37m au-dessus du sol (voir ?trees) alors que la formule du cône utilise le rayon à la base du cône, sachant que le bûcheron coupe son arbre quand même plus bas que cela.
Avec ce modèle, nous obtenons le critère d’Akaiké et l’erreur quadratique moyenne les plus faibles aussi. Tout indique que c’est le meilleur modèle, et en plus, il est mécaniste en ce sens qu’il fait intervenir la géométrie pour expliquer un volume à partir de deux dimensions dont l’une est élevée au carré. D’ailleurs, une analyse dimensionnelle (comparaison des unités intervenant à gauche et à droite dans l’équation) montre aussi qu’il est cohérent de ce point de vue : un volume en m3 s’obtient bien par la multiplication d’une dimension en m2 (le rayon au carré) par une dimension en m (la hauteur). Voici pour finir à quoi ressemblent les graphiques d’analyse des résidus de ce dernier modèle :
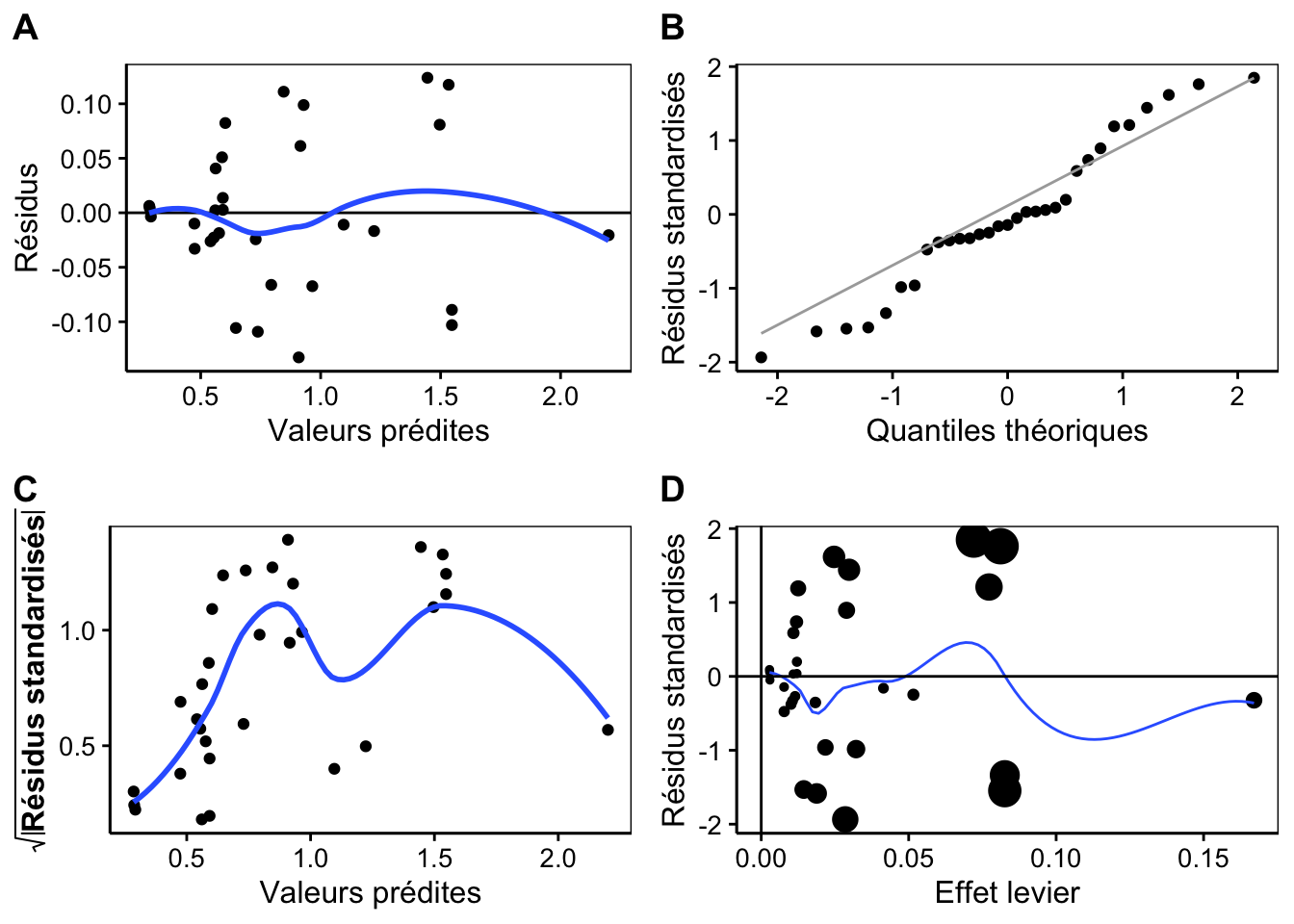
Clairement, nous aurions pu nous économiser de nombreux essais si nous étions partis directement sur cette piste-là. Malheureusement, la biologie fait intervenir souvent un ensemble de mécanismes (bio)chimiques et physiques qui rendent la compréhension mécanistique du phénomène difficile, voire impossible. Heureusement, les modèles empiriques sont également utiles à condition d’être conscient de leurs limites qui impliquent de ne pas chercher à interpréter les termes et les paramètres comme autant d’indicateurs des mécanismes responsables de la distribution des données et sans tirer de conclusions au-delà de la relation entre variables (pas d’établissement de causalité sur base d’une régression, jamais).
À vous de jouer !
Le projet suivant est un travail par groupe de quatre. Attendez les instructions de vos enseignants qui vont nommer des capitaines d’équipes qui créerons les différents “teams” avant toute chose.
Réalisez en groupe le travail B02Ga_models, partie I.
Travail en groupe de 4 pour les étudiants inscrits au cours de Science des Données Biologiques II à l’UMONS (Q1 : modélisation) à terminer avant le 2023-12-19 23:59:59.
Initiez votre projet GitHub Classroom
Voyez les explications dans le fichier README.md, partie I.