2.3 Régression linéaire polynomiale
Pour rappel, un polynome est une expression mathématique du type (notez la ressemblance avec l’équation de la régression multiple) :
\[ a_0 + a_1 . x + a_2 . x^2 + ... + a_n . x^n \]
Un polynome d’ordre 2 (terme jusqu’au \(x^2\)) correspond à une parabole dans le plan xy. Que se passe-t-il si nous calculons une variable diametre2 qui est le carré du diamètre et que nous prétendons faire une régression multiple en utilisant à la fois diamètre et diamètre2/ ?
trees %>.%
mutate(., diameter2 = diameter^2) -> trees
summary(lm(data = trees, volume ~ diameter + diameter2))#
# Call:
# lm(formula = volume ~ diameter + diameter2, data = trees)
#
# Residuals:
# Min 1Q Median 3Q Max
# -0.157938 -0.068324 -0.009027 0.060611 0.214988
#
# Coefficients:
# Estimate Std. Error t value Pr(>|t|)
# (Intercept) 0.3111 0.3180 0.978 0.336293
# diameter -2.3718 1.8381 -1.290 0.207489
# diameter2 11.2363 2.5563 4.396 0.000144 ***
# ---
# Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#
# Residual standard error: 0.09441 on 28 degrees of freedom
# Multiple R-squared: 0.9616, Adjusted R-squared: 0.9589
# F-statistic: 350.5 on 2 and 28 DF, p-value: < 2.2e-16Il semble que R ait pu réaliser cette analyse. Cette fois-ci, nous n’avons cependant pas une droite ou un plan ajusté, mais par ce subterfuge, nous avons pu ajuster une courbe dans les données ! Nous pourrions augmenter le degré du polynome (ajouter un terme en diameter^3, voire encore des puissance supérieures). Dans ce cas, nous obtiendrons une courbe de plus en plus flexible, toujours dans le plan xy. Ceci illustre parfaitement d’ailleurs l’ambiguité de la complexité du modèle qui s’ajuste de mieux en mieux dans les données, mais qui ce faisant, perd également progressivement son pouvoir explicatif. En effet, on sait qu’il existe toujours une droite qui passe entre deux points dans le plan. De même, il existe toujours une parabole qui passe par 3 points quelconques dans le plan. Et par extension, il existe une courbe correspondant à un polynome d’ordre n - 1 qui passe par n’importe quel ensemble de n points dans le plan. Un modèle construit à l’aide d’un tel polynome aura toujours un R2 égal à un, … mais en même temps ce modèle ne sera d’aucune utilité car il ne contient plus aucune information pertinente. C’est ce qu’on appelle le surajustement (overfitting en anglais). La figure ci-dessous (issue d’un article écrit par Anup Bhande ici) illuste bien ce phénomène.

Devoir calculer les différentes puissance des variables au préalable devient rapidement fastidieux. Heureusement, R autorise de glisser ce cacul directement dans la formule, mais à condition de lui indiquer qu’il ne s’agit pas du nom d’une variable diameter^2, mais d’un calcul effectué sur diameter en utilisant la fonction, d’identité I(). Ainsi, sans rien calculer au préalable, nous pouvons utiliser la formule volume ~ diameter + I(diameter^2). Un snippet est d’ailleurs disponible pour ajuster un polynome d’ordre 2 ou d’ordre 3, et il est accompagné du code nécessaire pour représenter également graphiquement cette régression polynomiale. Le code ci-dessous qui construit le modèle lm3 l’utilise.
summary(lm3 <- lm(data = trees,
volume ~ diameter + I(diameter^2)))#
# Call:
# lm(formula = volume ~ diameter + I(diameter^2), data = trees)
#
# Residuals:
# Min 1Q Median 3Q Max
# -0.157938 -0.068324 -0.009027 0.060611 0.214988
#
# Coefficients:
# Estimate Std. Error t value Pr(>|t|)
# (Intercept) 0.3111 0.3180 0.978 0.336293
# diameter -2.3718 1.8381 -1.290 0.207489
# I(diameter^2) 11.2363 2.5563 4.396 0.000144 ***
# ---
# Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#
# Residual standard error: 0.09441 on 28 degrees of freedom
# Multiple R-squared: 0.9616, Adjusted R-squared: 0.9589
# F-statistic: 350.5 on 2 and 28 DF, p-value: < 2.2e-16lm3 %>.% (function(lm, model = lm[["model"]], vars = names(model))
chart(model, aes_string(x = vars[2], y = vars[1])) +
geom_point() +
stat_smooth(method = "lm", formula = y ~ x + I(x^2)))(.)
Remarquez sur le graphique comment, à présent, la courbe s’ajuste bien mieux dans le nuage de point et comme l’arbre le plus grand avec un diamètre supérieur à 0,5m est à présent presque parfaitement ajusté par le modèle. Faites donc très attention que des points influents ou extrêmes peuvent apparaitre également comme tel à cause d’un mauvais choix de modèle !
L’analyse des résidus nous montre aussi un comportement plus sain.
#plot(lm3, which = 1)
lm3 %>.%
chart(broom::augment(.), .resid ~ .fitted) +
geom_point() +
geom_hline(yintercept = 0) +
geom_smooth(se = FALSE, method = "loess", formula = y ~ x) +
labs(x = "Fitted values", y = "Residuals") +
ggtitle("Residuals vs Fitted") 
#plot(lm3, which = 2)
lm3 %>.%
chart(broom::augment(.), aes(sample = .std.resid)) +
geom_qq() +
geom_qq_line(colour = "darkgray") +
labs(x = "Theoretical quantiles", y = "Standardized residuals") +
ggtitle("Normal Q-Q") 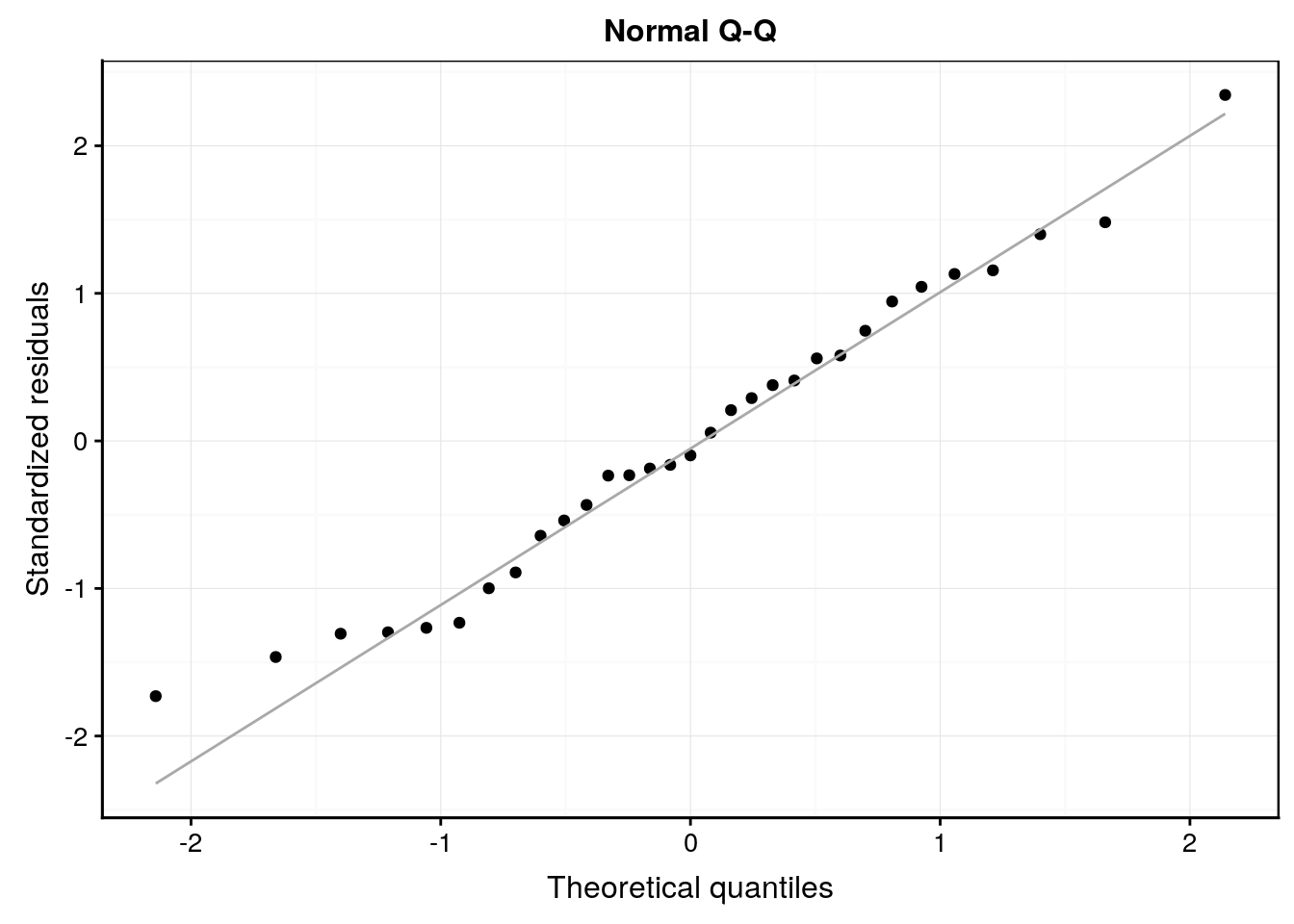
#plot(lm3, which = 3)
lm3 %>.%
chart(broom::augment(.), sqrt(abs(.std.resid)) ~ .fitted) +
geom_point() +
geom_smooth(se = FALSE, method = "loess", formula = y ~ x) +
labs(x = "Fitted values",
y = expression(bold(sqrt(abs("Standardized residuals"))))) +
ggtitle("Scale-Location") 
#plot(lm3, which = 4)
lm3 %>.%
chart(broom::augment(.), .cooksd ~ seq_along(.cooksd)) +
geom_bar(stat = "identity") +
geom_hline(yintercept = seq(0, 0.1, by = 0.05), colour = "darkgray") +
labs(x = "Obs. number", y = "Cook's distance") +
ggtitle("Cook's distance") 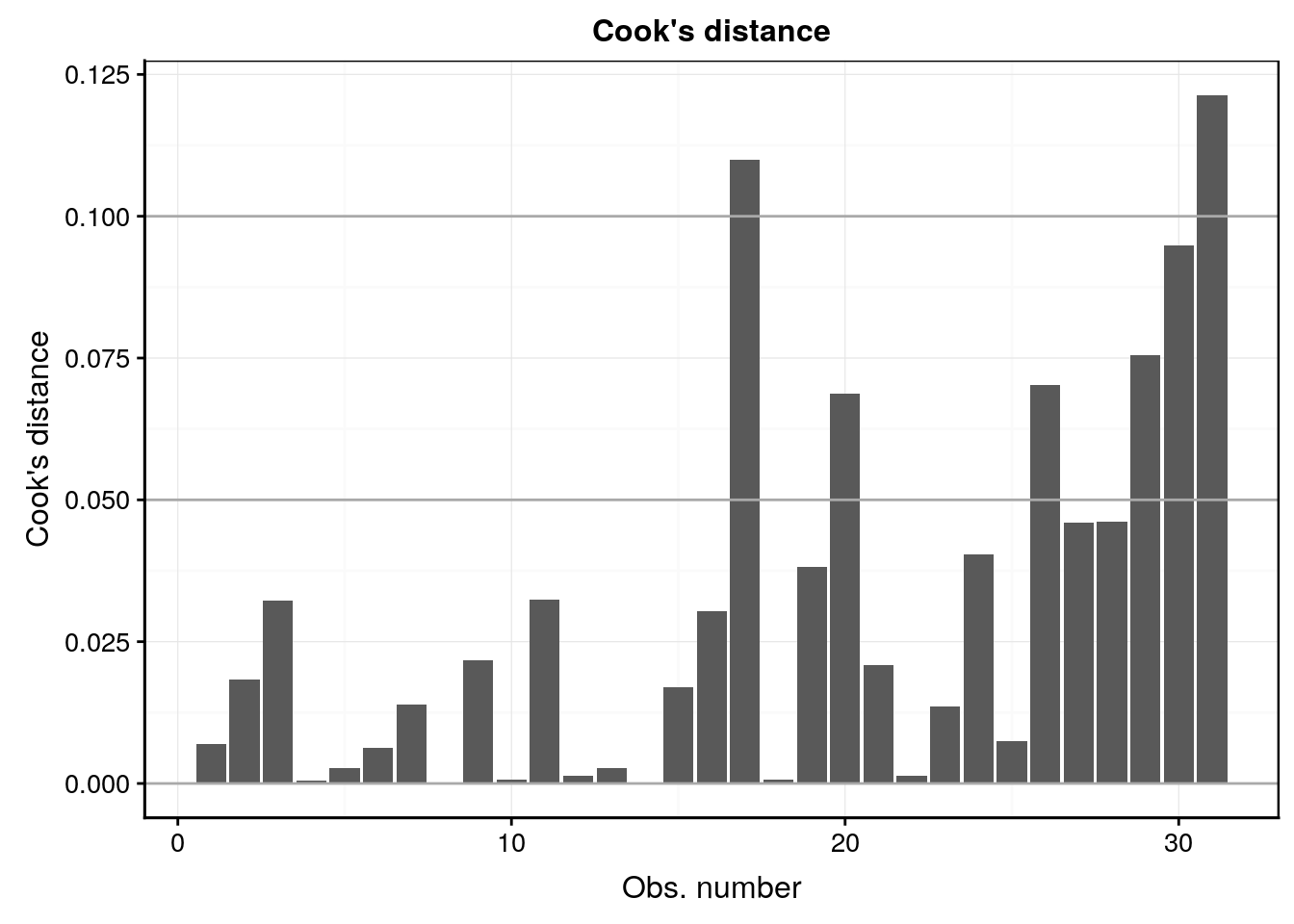
Revenons un instant sur le résumé de ce modèle.
summary(lm3)#
# Call:
# lm(formula = volume ~ diameter + I(diameter^2), data = trees)
#
# Residuals:
# Min 1Q Median 3Q Max
# -0.157938 -0.068324 -0.009027 0.060611 0.214988
#
# Coefficients:
# Estimate Std. Error t value Pr(>|t|)
# (Intercept) 0.3111 0.3180 0.978 0.336293
# diameter -2.3718 1.8381 -1.290 0.207489
# I(diameter^2) 11.2363 2.5563 4.396 0.000144 ***
# ---
# Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#
# Residual standard error: 0.09441 on 28 degrees of freedom
# Multiple R-squared: 0.9616, Adjusted R-squared: 0.9589
# F-statistic: 350.5 on 2 and 28 DF, p-value: < 2.2e-16La pente relative au diameter nécessite quelques éléments d’explication. En effet, que signifie une pente pour une courbe dont la dérivée première (“pente locale”) change constamment ? En fait, il faut comprendre ce paramètre comme étant la pente de la courbe au point x = 0.
Si le modèle est très nettement significatif (ANOVA, valeur p <<< 0,001), et si le R2 ajusté grimpe maintenant à 0,959, seul le paramètre relatif au diamètre2 est significatif cette fois-ci. Ce résultat suggère que ce modèle pourrait êtrte simplifié en considérant que l’ordonnée à l’origine et la pente pour le terme diameter valent zéro. Cela peut être tenté, mais à condition de refaire l’analyse. On ne peut jamais laisser tomber un paramètre dans une analyse et considérer que les autres sont utilisable tels quels. tous les paramètres caculés sont interconnectés.
Voyons ce que cela donne (la formule devient volume ~ I(diameter^2) - 1 ou volume ~ I(diameter^2) + 0, ce qui est identique) :
summary(lm4 <- lm(data = trees, volume ~ I(diameter^2) - 1))#
# Call:
# lm(formula = volume ~ I(diameter^2) - 1, data = trees)
#
# Residuals:
# Min 1Q Median 3Q Max
# -0.19474 -0.07234 -0.04120 0.04522 0.18240
#
# Coefficients:
# Estimate Std. Error t value Pr(>|t|)
# I(diameter^2) 7.3031 0.1397 52.26 <2e-16 ***
# ---
# Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#
# Residual standard error: 0.1027 on 30 degrees of freedom
# Multiple R-squared: 0.9891, Adjusted R-squared: 0.9888
# F-statistic: 2731 on 1 and 30 DF, p-value: < 2.2e-16Notez bien que quand on réajuste un modèle simplifié, les paramètres restants doivent être recalculés. En effet, le paramètre relatif au diamètre2 vallait 11,2 dans le modèle lm3 plus haut. Un fois les autres termes éliminés, ce paramètre devient 7,30 dans ce modèle lm4 simplifié.
Le modèle lm4 revient aussi (autre point de vue) à transformer d’abord le diamètre en diamètre2 et à effectuer ensuite une régression linéaire simple entre deux variables, volume et diametre2 :
summary(lm4bis <- lm(data = trees, volume ~ diameter2 - 1))#
# Call:
# lm(formula = volume ~ diameter2 - 1, data = trees)
#
# Residuals:
# Min 1Q Median 3Q Max
# -0.19474 -0.07234 -0.04120 0.04522 0.18240
#
# Coefficients:
# Estimate Std. Error t value Pr(>|t|)
# diameter2 7.3031 0.1397 52.26 <2e-16 ***
# ---
# Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#
# Residual standard error: 0.1027 on 30 degrees of freedom
# Multiple R-squared: 0.9891, Adjusted R-squared: 0.9888
# F-statistic: 2731 on 1 and 30 DF, p-value: < 2.2e-16Notez qu’on obtient bien évidemment exactement les mêmes résultats si nous transformons d’abord les données ou si nous intégrons le calcul à l’intérieur de la formule qui décrit le modèle.
Faites bien attention de ne pas comparer le R2 acvec ordonnée à l’origine fixée à zéro ici dans notre modèle lm4 avec les R2 des modèles lm. ou lm3 qui ont ce paramètre estimé. Rappelez-vous que le R2 est calculé différemment dans les deux cas ! Donc, nous voilà une fois de plus face à un nouveau modèle pour lequel il nous est difficile de décider s’il est meilleur que les précédents. Avant de comparer, élaborons un tout dernier modèle, le plus complexe, qui reprend à la fois notre régression polynomiale d’ordre 2 sur le diamètre et la hauteur. Autrement dit, une régression à la fois multiple et polynomiale.
summary(lm5 <- lm(data = trees, volume ~ diameter + I(diameter^2) + height))#
# Call:
# lm(formula = volume ~ diameter + I(diameter^2) + height, data = trees)
#
# Residuals:
# Min 1Q Median 3Q Max
# -0.12169 -0.04806 -0.00237 0.05156 0.12559
#
# Coefficients:
# Estimate Std. Error t value Pr(>|t|)
# (Intercept) -0.267097 0.284895 -0.938 0.356798
# diameter -3.281421 1.463401 -2.242 0.033354 *
# I(diameter^2) 11.891724 2.019252 5.889 2.83e-06 ***
# height 0.034788 0.008169 4.259 0.000223 ***
# ---
# Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#
# Residual standard error: 0.07436 on 27 degrees of freedom
# Multiple R-squared: 0.977, Adjusted R-squared: 0.9745
# F-statistic: 382.8 on 3 and 27 DF, p-value: < 2.2e-16Ah ha, ceci est bizarre ! Le R2 ajusté nous indique que le modèle serait très bon puisqu’il grimpe à 0,975. Le terme en diamètre2 reste très significatif, … mais la pente relative à la hauteur est maintenant elle aussi très significative alors que dans le modèle multiple lm2 ce n’était pas le cas. De plus, la pente à l’origine en face du diamètre semble devenir un peu plus significative. Bienvenue dans les instabilités liées aux intercorrelations entre paramètres dans les modèles linéaires complexes.