4.4 Figures composées
Il arrive fréquemment de vouloir combiner plusieurs graphiques dans une même figure. Plusieurs fonctions sont à votre disposition pour cela. Il faut tout d’abord distinguer deux types de figures multi-graphiques :
- Soit il s’agit d’un seul graphique que vous souhaitez subdiviser par rapport à une ou plusieurs variables facteurs.
- Soit il s’agit de graphiques indépendants que vous souhaitez assembler dans une même figure parce que les données ont un lien entre elles, ou parce que ces graphiques sont complémentaires pour comprendre les données.
Dans le premier cas, les fonctions facet_XXX() comme facet_grid() peuvent être employées (ou l’opérateur | dans une formule). Dans le second cas, la fonction combine_charts() est l’une des alternatives possibles.
4.4.1 Facettes
L’une des règles les plus importantes que vous devez impérativement garder à l’esprit lors de la réalisation de vos graphiques est la simplicité. Plus votre graphique contiendra d’information, plus il sera compliqué à décoder par vos lecteurs.
# Importation de données relative à la croissance de poulets
(chick_weight <- read("ChickWeight", package = "datasets", lang = "fr"))# weight time chick diet
# 1: 42 0 1 1
# 2: 51 2 1 1
# 3: 59 4 1 1
# 4: 64 6 1 1
# 5: 76 8 1 1
# ---
# 574: 175 14 50 4
# 575: 205 16 50 4
# 576: 234 18 50 4
# 577: 264 20 50 4
# 578: 264 21 50 4# Réalisation du graphique (points semi-transparents)
chart(data = chick_weight, weight ~ time %col=% diet) +
geom_point(alpha = 0.5)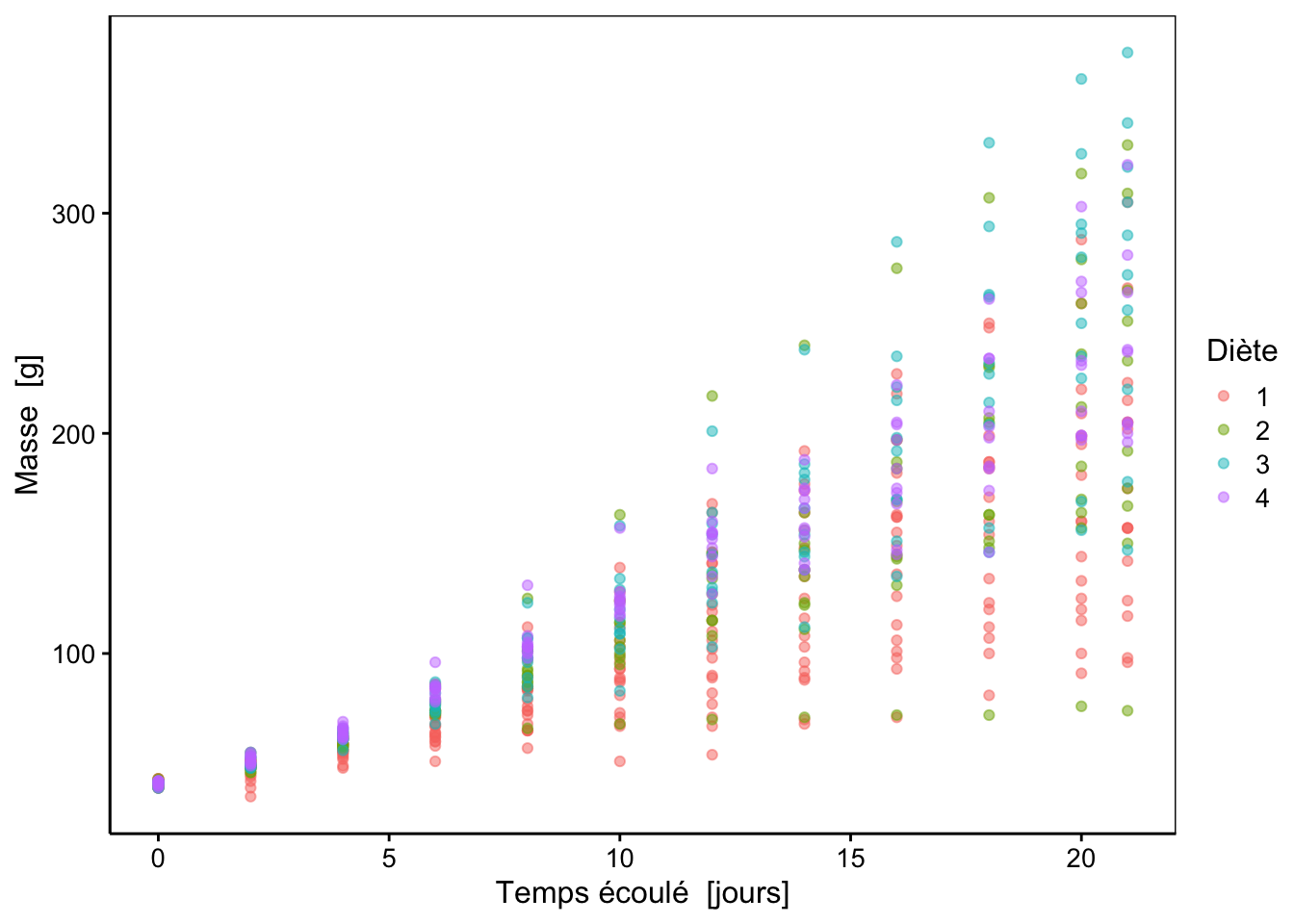
Figure 4.20: Croissance de poulets en utilisant quatre aliments différents.
Le graphique à la Fig. 4.20 est mal adapté pour montrer les différences entre les quatre aliments : tous les points sont entremêlés. Il peut typiquement être simplifié en utilisant des facettes pour représenter les résultats relatifs aux différents régimes alimentaires sur des graphiques séparés. L’information est la même, mais la lecture est beaucoup plus aisée.
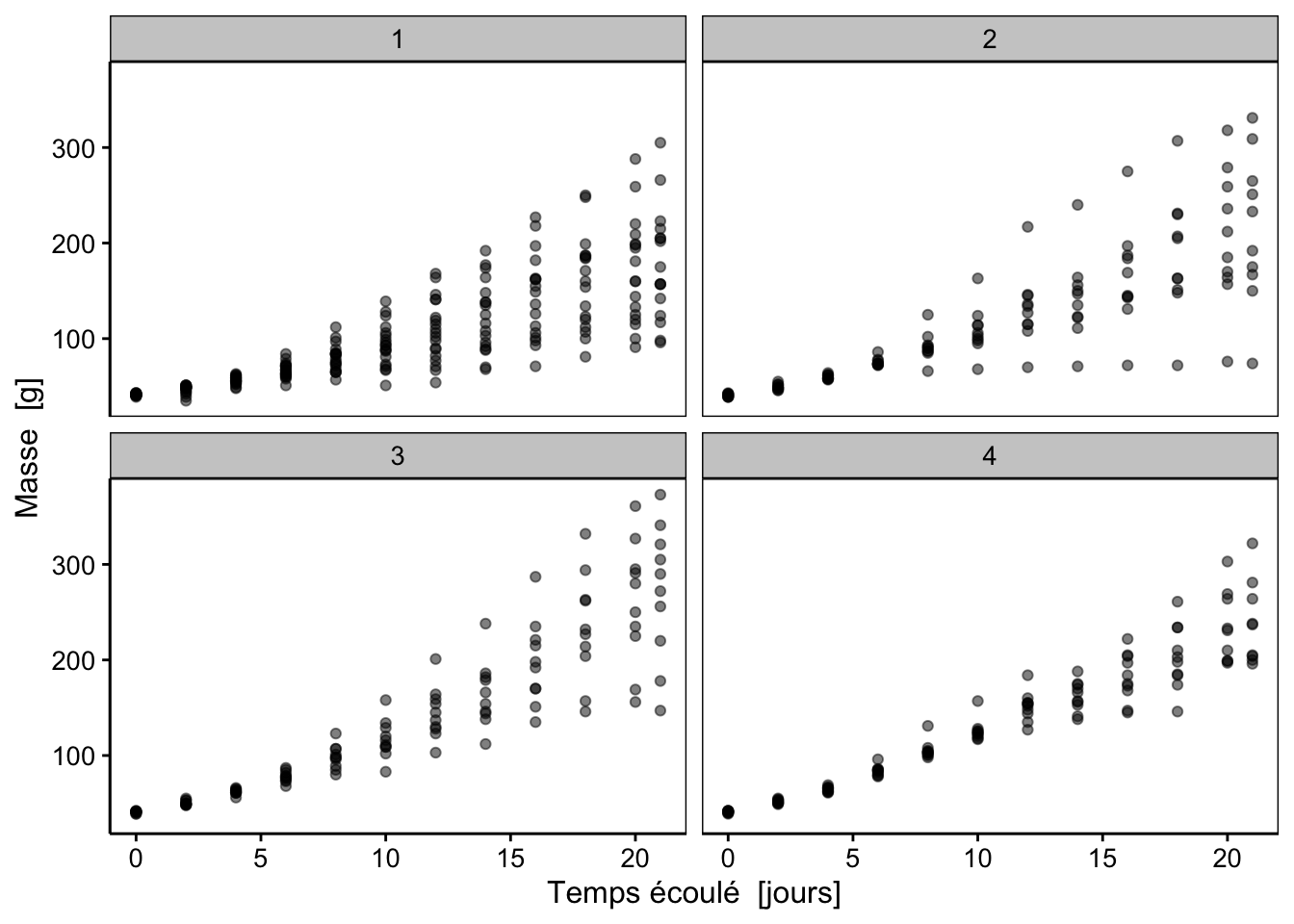
Figure 4.21: Croissance de poulets en utilisant quatre aliments différents (1-4).
Vous observez que les échelles en abscisse et en ordonnée sont similaires sur tous les graphiques. Cela permet une meilleure comparaison. Notez toutefois que, plus le nombre de facettes augmente, plus chaque graphique individuel devient petit. Faites attention à ne pas finir avec des graphiques individuels tellement petits qu’ils en deviennent illisibles !
4.4.2 Graphiques assemblés
La fonction combine_charts() permet de combiner plusieurs graphiques dans une figure unique. Nous l’avons déjà utilisée à plusieurs reprises. Cette fonction attend une liste (list()) de graphiques de type chart() à assembler, et il est possible d’en combiner les légendes à l’aide de common.legend = TRUE.
# Importation des données
urchin <- read("urchin_bio", package = "data.io", lang = "FR")
# Réalisation des graphiques
a <- chart(data = urchin, weight ~ height %col=% origin) +
geom_point()
b <- chart(data = urchin, weight ~ solid_parts %col=% origin) +
geom_point()
# Combinaison des graphiques dans une même figure
combine_charts(list(a, b), common.legend = TRUE)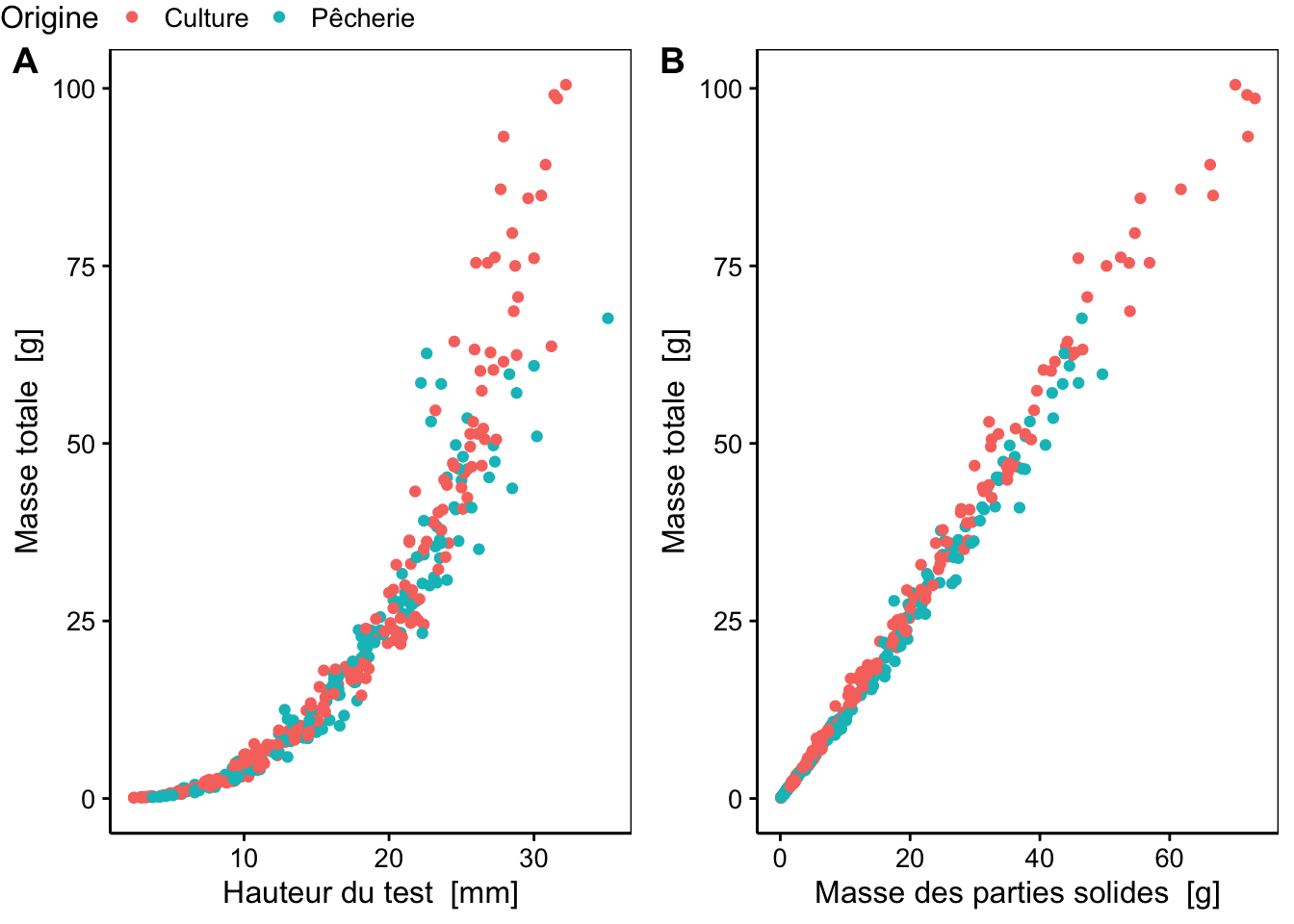
Figure 4.22: A) Masse d’oursins en fonction de leur taille et de leur origine. B) Masse totale en fonction de la masse des parties solides de ces mêmes oursins.
Il existe d’autres fonctions permettant de combiner plusieurs graphiques comme cowplot::plot_grid()ou encore, les fonctions du package {patchwork}, mais avec combine_charts() vous pourrez déjà faire beaucoup. De plus, un libellé sous forme d’une lettre majuscule est automatiquement associé à chaque sous-région de la figure composée. Cela permet d’y faire plus facilement référence dans le texte et/ou dans la légende.
À vous de jouer !
Effectuez maintenant les exercices du tutoriel A04Lc_comp_fig (Graphiques composés).
BioDataScience1::run("A04Lc_comp_fig")Réalisez le travail A04Ia_graphe_avance.
Travail individuel pour les étudiants inscrits au cours de Science des Données Biologiques I : visualisation à l’UMONS à terminer avant le 2023-11-20 23:59:59.
Initiez votre projet GitHub Classroom
Voyez les explications dans le fichier README.md.
Pour en savoir plus
Partitionnement des graphiques en facettes. Différentes options sont présentées ici.
Figures composées à l’aide de
cowplot::plot_grid()avec les différentes options, aussi disponibles aveccombine_charts().Seconde possibilité pour des figures composées à l’aide de
ggpubr::ggarrange().combine_charts()fait la même chose, mais avec des valeurs par défaut légèrement différentes (labels = "auto"par défaut pour ce dernier, maislabels = NULLpourggpubr::ggarrange()).Site du package {patchwork}. Une façon puissante et intuitive de réaliser des graphiques composites dans R.