7.3 Accès aux bases de données
Puisque nous traitons de jeux de données multivariés potentiellement très gros, il devient important de pouvoir accéder aux données stockées de manière plus structurée que dans un fichier au format CSV ou Excel, par exemple. Les bases de données, et notamment les bases de données relationnelles, sont prévue pour stocker de grandes quantités de données de manière structurée et pour pouvoir en extraire la partie qui nous intéresse à l’aide de requêtes. Nous allons ici étudier les rudiments indispensables pour nous permettre de réaliser de telles requêtes depuis R.
auteur d’une liste de livres dans la table Livres renvoie (est lié à) vers le champs nom d’une autre table Ecrivains qui fournit plus de détails sur chaque auteur de livres.
Il existe différents moteurs de bases de données relationnelles. Les plus courants sont : SQLite, MySQL/MariaDB, PosgreSQL, SQL Server, Oracle, … La plupart de ces solutions nécessitent d’installer un serveur de base de données centralisé. Cependant, SQLite, est une solution légère qui permet d’explorer le language SQL (prononcez “S.Q.L.” ou “Sequel”), y compris avec des petites bases de données test en mémoire ou contenues dans un fichier.
7.3.1 Installation de SQLite
Dans la SciViews Box, les drivers SQLite pour la version 2 et la version 3 sont préinstallés. Sous R, vous pouvez utiliser le package RSQLite pour accéder à des bases de données qui sont de simples fichiers sur le disque. Cependant, l’onglet Connections dans RStudio n’est pas compatible avec RSQLite. Il fonctionne, par contre avec les drivers odbc qui sont un format commun de drivers pour différentes bases de donnes dont SQLite. Les drivers SQLite, MySQL et PosgreSQL sont préinstallés dans la SciViews Box. Nous utiliserons également une interfaces graphique vers SQLite : DB Browser for SQLite disponible depuis le menu principal Applications, dans la section Development.
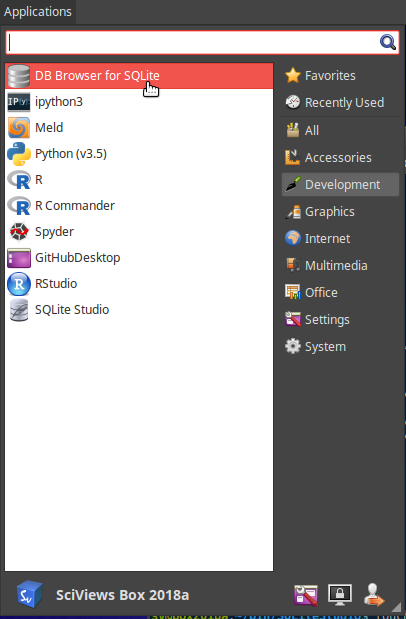
7.3.2 Base de données en mémoire
La simplicité de SQLite tient au fait qu’il n’est pas nécessaire d’installer un serveur de bases de données pour l’utiliser. La version la plus simple permet même de travailler directement en mémoire. Ainsi, nous pouvons facilement placer le contenu d’un jeu de données comme mtcars et tester ensuite des requêtes SQL (l’équivalent des fonctions d’extraction et de remaniement de tableau dans dplyr) sur ces données en mémoire.
Voici un petit aperçu qui vous montre comment créer, et puis manipuler une base de données SQLite en mémoire depuis R.
library('RSQLite')
# Base de données en mémoire
con <- dbConnect(RSQLite::SQLite(), dbname = ":memory:")
# Ne contient encore rien
dbListTables(con)# character(0)# [1] "mtcars"# [1] "mpg" "cyl" "disp" "hp" "drat" "wt" "qsec" "vs" "am" "gear"
# [11] "carb"# mpg cyl disp hp drat wt qsec vs am gear carb
# 1 21.0 6 160.0 110 3.90 2.620 16.46 0 1 4 4
# 2 21.0 6 160.0 110 3.90 2.875 17.02 0 1 4 4
# 3 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1
# 4 21.4 6 258.0 110 3.08 3.215 19.44 1 0 3 1
# 5 18.7 8 360.0 175 3.15 3.440 17.02 0 0 3 2
# 6 18.1 6 225.0 105 2.76 3.460 20.22 1 0 3 1
# 7 14.3 8 360.0 245 3.21 3.570 15.84 0 0 3 4
# 8 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
# 9 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
# 10 19.2 6 167.6 123 3.92 3.440 18.30 1 0 4 4
# 11 17.8 6 167.6 123 3.92 3.440 18.90 1 0 4 4
# 12 16.4 8 275.8 180 3.07 4.070 17.40 0 0 3 3
# 13 17.3 8 275.8 180 3.07 3.730 17.60 0 0 3 3
# 14 15.2 8 275.8 180 3.07 3.780 18.00 0 0 3 3
# 15 10.4 8 472.0 205 2.93 5.250 17.98 0 0 3 4
# 16 10.4 8 460.0 215 3.00 5.424 17.82 0 0 3 4
# 17 14.7 8 440.0 230 3.23 5.345 17.42 0 0 3 4
# 18 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
# 19 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
# 20 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1
# 21 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1
# 22 15.5 8 318.0 150 2.76 3.520 16.87 0 0 3 2
# 23 15.2 8 304.0 150 3.15 3.435 17.30 0 0 3 2
# 24 13.3 8 350.0 245 3.73 3.840 15.41 0 0 3 4
# 25 19.2 8 400.0 175 3.08 3.845 17.05 0 0 3 2
# 26 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1
# 27 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2
# 28 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2
# 29 15.8 8 351.0 264 4.22 3.170 14.50 0 1 5 4
# 30 19.7 6 145.0 175 3.62 2.770 15.50 0 1 5 6
# 31 15.0 8 301.0 335 3.54 3.570 14.60 0 1 5 8
# 32 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2# Effectuer une requête SQL sur la table
res <- dbSendQuery(con, "SELECT * FROM mtcars WHERE cyl = 4")
dbFetch(res)# mpg cyl disp hp drat wt qsec vs am gear carb
# 1 22.8 4 108.0 93 3.85 2.320 18.61 1 1 4 1
# 2 24.4 4 146.7 62 3.69 3.190 20.00 1 0 4 2
# 3 22.8 4 140.8 95 3.92 3.150 22.90 1 0 4 2
# 4 32.4 4 78.7 66 4.08 2.200 19.47 1 1 4 1
# 5 30.4 4 75.7 52 4.93 1.615 18.52 1 1 4 2
# 6 33.9 4 71.1 65 4.22 1.835 19.90 1 1 4 1
# 7 21.5 4 120.1 97 3.70 2.465 20.01 1 0 3 1
# 8 27.3 4 79.0 66 4.08 1.935 18.90 1 1 4 1
# 9 26.0 4 120.3 91 4.43 2.140 16.70 0 1 5 2
# 10 30.4 4 95.1 113 3.77 1.513 16.90 1 1 5 2
# 11 21.4 4 121.0 109 4.11 2.780 18.60 1 1 4 2dbClearResult(res)
# On peut aussi récupérer les données morceau par morceau
res <- dbSendQuery(con, "SELECT * FROM mtcars WHERE cyl = 4")
while (!dbHasCompleted(res)) {
chunk <- dbFetch(res, n = 5)
print(nrow(chunk))
}# [1] 5
# [1] 5
# [1] 17.3.3 Base de données dans un fichier
La SciViews Box contient une base de données test dans ~/shared/database.sqlite. Vous pouvez facilement vous connecter dessus via l’onglet Connections de RStudio. Vous pouvez également vous y connecter via du code R directement. Notez que la même syntaxe est utilisée pour créer une nouvelle base de données si le fichier n’existe pas encore au moment de la connexion.
À vous de jouer !
Voici quelques instructions typiques pour interroger cette base de données depuis R :
# [1] "iris" "mtcars" "versicolor"# Extraction de données à l'aide d'une requête SQL
(setosa <- dbGetQuery(con, "SELECT * FROM iris WHERE Species is 'setosa'"))# Sepal.Length Sepal.Width Petal.Length Petal.Width Species
# 1 5.1 3.5 1.4 0.2 setosa
# 2 4.9 3.0 1.4 0.2 setosa
# 3 4.7 3.2 1.3 0.2 setosa
# 4 4.6 3.1 1.5 0.2 setosa
# 5 5.0 3.6 1.4 0.2 setosa
# 6 5.4 3.9 1.7 0.4 setosa
# 7 4.6 3.4 1.4 0.3 setosa
# 8 5.0 3.4 1.5 0.2 setosa
# 9 4.4 2.9 1.4 0.2 setosa
# 10 4.9 3.1 1.5 0.1 setosa
# 11 5.4 3.7 1.5 0.2 setosa
# 12 4.8 3.4 1.6 0.2 setosa
# 13 4.8 3.0 1.4 0.1 setosa
# 14 4.3 3.0 1.1 0.1 setosa
# 15 5.8 4.0 1.2 0.2 setosa
# 16 5.7 4.4 1.5 0.4 setosa
# 17 5.4 3.9 1.3 0.4 setosa
# 18 5.1 3.5 1.4 0.3 setosa
# 19 5.7 3.8 1.7 0.3 setosa
# 20 5.1 3.8 1.5 0.3 setosa
# 21 5.4 3.4 1.7 0.2 setosa
# 22 5.1 3.7 1.5 0.4 setosa
# 23 4.6 3.6 1.0 0.2 setosa
# 24 5.1 3.3 1.7 0.5 setosa
# 25 4.8 3.4 1.9 0.2 setosa
# 26 5.0 3.0 1.6 0.2 setosa
# 27 5.0 3.4 1.6 0.4 setosa
# 28 5.2 3.5 1.5 0.2 setosa
# 29 5.2 3.4 1.4 0.2 setosa
# 30 4.7 3.2 1.6 0.2 setosa
# 31 4.8 3.1 1.6 0.2 setosa
# 32 5.4 3.4 1.5 0.4 setosa
# 33 5.2 4.1 1.5 0.1 setosa
# 34 5.5 4.2 1.4 0.2 setosa
# 35 4.9 3.1 1.5 0.2 setosa
# 36 5.0 3.2 1.2 0.2 setosa
# 37 5.5 3.5 1.3 0.2 setosa
# 38 4.9 3.6 1.4 0.1 setosa
# 39 4.4 3.0 1.3 0.2 setosa
# 40 5.1 3.4 1.5 0.2 setosa
# 41 5.0 3.5 1.3 0.3 setosa
# 42 4.5 2.3 1.3 0.3 setosa
# 43 4.4 3.2 1.3 0.2 setosa
# 44 5.0 3.5 1.6 0.6 setosa
# 45 5.1 3.8 1.9 0.4 setosa
# 46 4.8 3.0 1.4 0.3 setosa
# 47 5.1 3.8 1.6 0.2 setosa
# 48 4.6 3.2 1.4 0.2 setosa
# 49 5.3 3.7 1.5 0.2 setosa
# 50 5.0 3.3 1.4 0.2 setosaLa dernière instruction nécessite quelques explications supplémentaires. La fonction dbGetQuery() envoie une requête sur la base en langage SQL. Ici, nous indiquons les colonnes que nous souhaitons récupérer avec le mot clé SELECT. L’utilisation de * indique que nous voulons toutes les colonnes, sinon, on nomme celles que l’on veut à la place. Ensuite, le mot clé FROM nous indique depuis qulle table, ici celle nommée iris, et enfin, le mot clé WHERE introduit une expression de condition qui va filtrer les lignes de la table à récupérer, par exemple 'Sepal.Length' > 1.5 ou comme ici Species is 'setosa'.
Il est également possible d’effectuer une requête SQL directement dans un chunk. A la place d’indiquer ```{r}, on indiquera ```{sql, connection=con}, et nous pourrons alors directement indiquer la requête SQL dans le chunk :
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | Species |
|---|---|---|---|---|
| 5.1 | 3.5 | 1.4 | 0.2 | setosa |
| 4.9 | 3.0 | 1.4 | 0.2 | setosa |
| 4.7 | 3.2 | 1.3 | 0.2 | setosa |
| 4.6 | 3.1 | 1.5 | 0.2 | setosa |
| 5.0 | 3.6 | 1.4 | 0.2 | setosa |
| 5.4 | 3.9 | 1.7 | 0.4 | setosa |
| 4.6 | 3.4 | 1.4 | 0.3 | setosa |
| 5.0 | 3.4 | 1.5 | 0.2 | setosa |
| 4.4 | 2.9 | 1.4 | 0.2 | setosa |
| 4.9 | 3.1 | 1.5 | 0.1 | setosa |
Par défaut, cette requête est imprimée dans le document, et son résultat est perdu ensuite. Il est cependant possible de l’enregistrer sous un nom avec l’option de chunk output.var=. Dans ce cas, rien n’est imprimé, mais comme le résultat de la requête est contenu dans l’objet créé, il est facile de le manipuler dans R ensuite plus loin dans notre document.
Ensuite, dans un chunk R, vous pouvez manipuler la table contenue dans virginica :
# [1] 50# Sepal.Length Sepal.Width Petal.Length Petal.Width
# Min. :4.900 Min. :2.200 Min. :4.500 Min. :1.400
# 1st Qu.:6.225 1st Qu.:2.800 1st Qu.:5.100 1st Qu.:1.800
# Median :6.500 Median :3.000 Median :5.550 Median :2.000
# Mean :6.588 Mean :2.974 Mean :5.552 Mean :2.026
# 3rd Qu.:6.900 3rd Qu.:3.175 3rd Qu.:5.875 3rd Qu.:2.300
# Max. :7.900 Max. :3.800 Max. :6.900 Max. :2.500
# Species
# Length:50
# Class :character
# Mode :character
#
#
# Ne pas oublier de se déconnecter de la base de données une fois terminé.
7.3.4 Driver ODBC dans RStudio
RStudio facilite l’utilisation de bases de données à condition d’utiliser un driver “compatible”. Nous avons installé un tel driver ODBC pour les bases de données SQLite. Pour nous connecter à /home/sv/shared/database.sqlite depuis RStudio, nous entrons dans l’onglet Connections et nous cliquons sur le bouton New Connection, ou nous cliquons sur la connection correspondante si elle est déjà créée dans la liste.
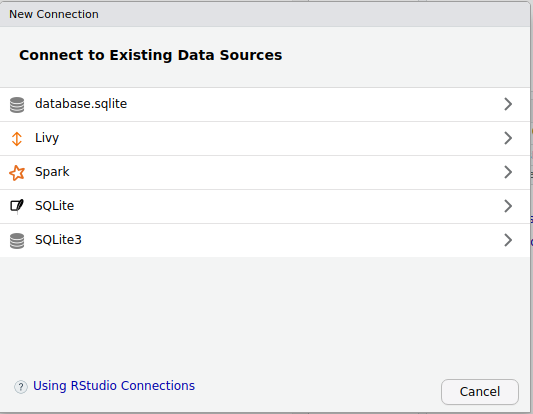
Pour créer une nouvelle connection, sélectionnons SQLite3. Ensuite, nous rentrons Database=/home/sv/shared/database.sqlite comme paramètre dans la fenêtre suivante.
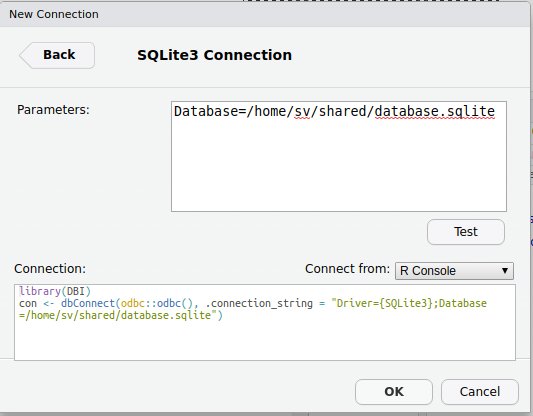
Le bouton Test permet de vérifier que R/RStudio peut se connecter à cette base de données. Ensuite, dans Connect from:, vous pouvez choisir où vous voulez placer l’instruction de connexion. L’option Clipboard est intéressante. Elle place l’instruction dans le presse-papier et vous pouvez alors décider vous-même où la placer. Nous la placerons dans un chunk R dans notre notebook.
library(odbc)
con <- dbConnect(odbc::odbc(), .connection_string = "Driver={SQLite3};Database=/home/sv/shared/database.sqlite")Une fois connecté, vous pouvez voir le contenu de la base de données dans l’onglet Connections. Pour ajouter une table que nous remplissons à partir des données issues du jeu de données mtcars, nous écririons dans R :
A partir de ce moment, vous pouvez voir votre table mtcars (il faut peut être cliquer sur le bouton en forme de flèche qui se mord la queue pour rafraichir l’affichage). Comme dans Environnement, si vous cliquez sur la flèche dans un rond bleu devant le nom de la table, vous pouvez voir les colonnes qu’elle contient. En cliquant sur l’icône tableau à droite, vous visualisez la table directement dans RStudio.
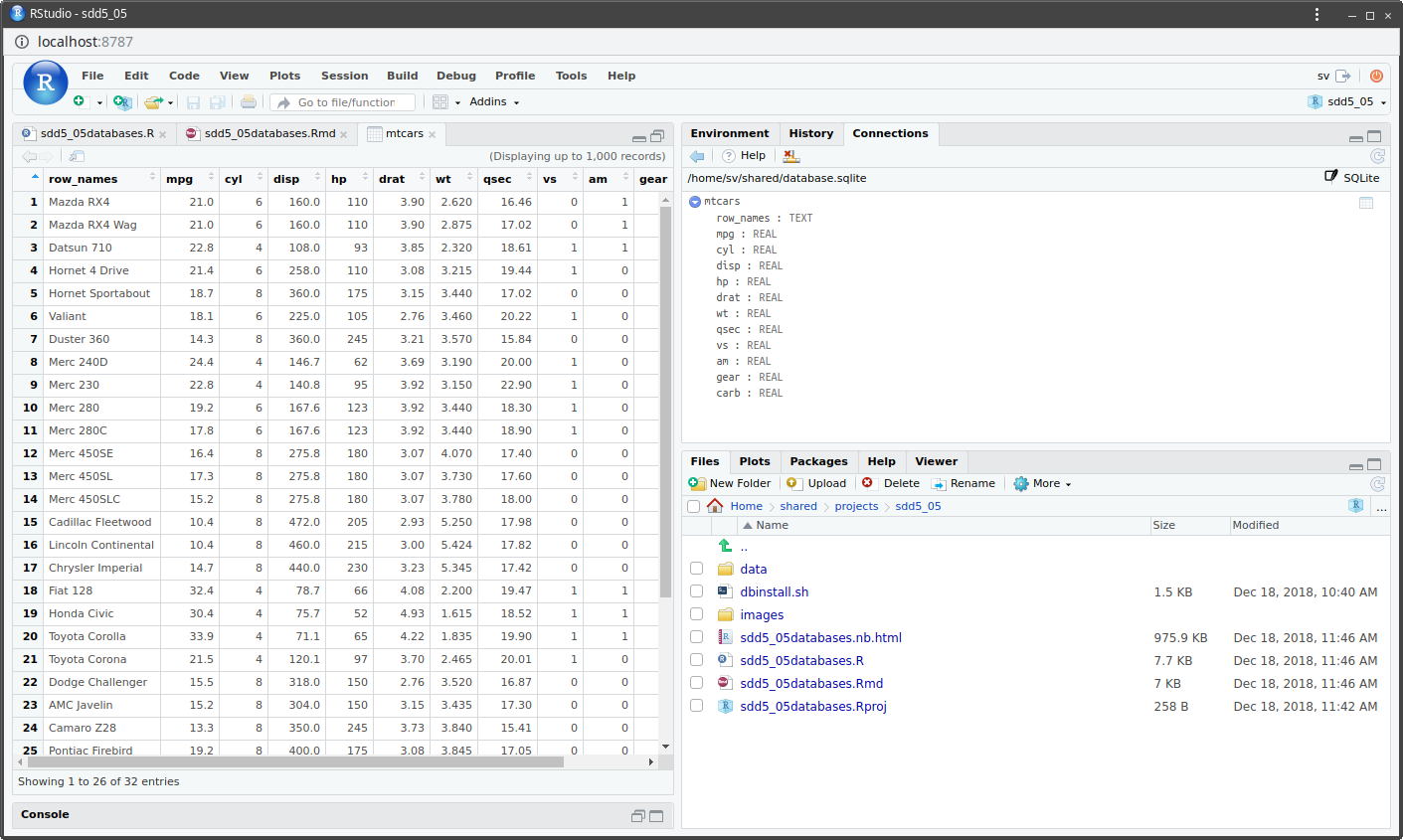
A part cela, vous travaillez avec cette base de données dans R en utilisant l’objet con comme d’habitude, et vous pouvez aussi utiliser directement des chunks sql.
7.3.5 Utilisation de DB Browser
Lancez DB Browser. Connectez-vous à /home/sv/shared/database.sqlite Vous avez un accès visuel à votre base de données. Explorez les différentes possibilités du logiciel.
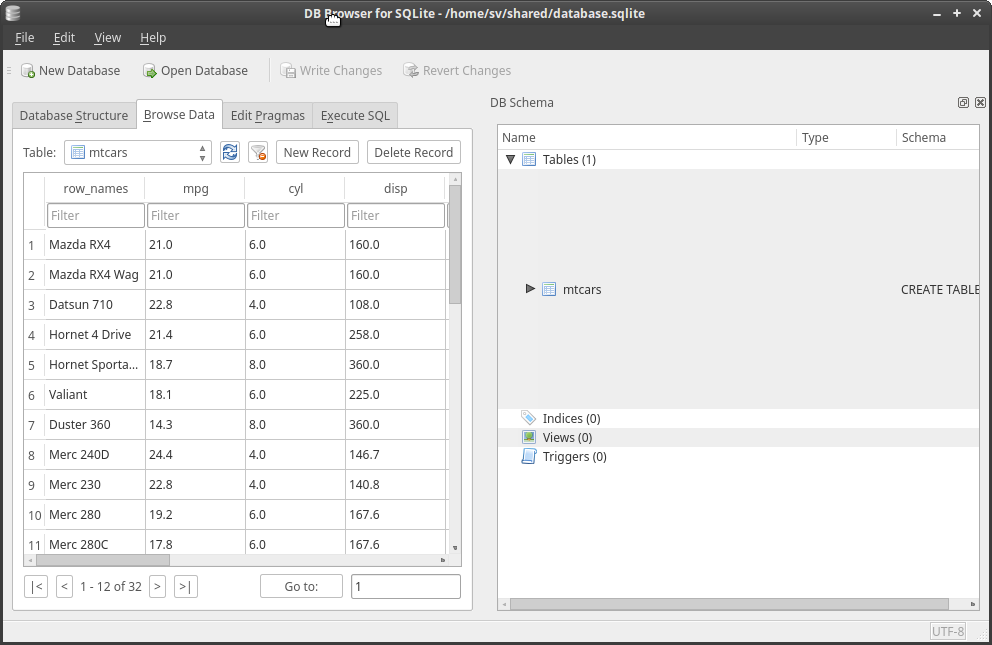
DB Browser for SQLite
7.3.6 Utilisation de {dplyr}
Les fonctions du package {dplyr} fonctionnent aussi très bien sur des bases de données. Les commandes sont converties en interne en requêtes SQL. Il suffit d’utiliser collect() à la fin pour exécuter la requête.
dbfile <- "~/shared/database.sqlite"
my_db <- src_sqlite(dbfile) # Utiliser create = TRUE pour la créer
my_db# src: sqlite 3.30.1 [/media/sf_shared/database.sqlite]
# tbls: iris, mtcars, versicolor# # A tibble: 150 x 5
# Sepal.Length Sepal.Width Petal.Length Petal.Width Species
# <dbl> <dbl> <dbl> <dbl> <chr>
# 1 5.1 3.5 1.4 0.2 setosa
# 2 4.9 3 1.4 0.2 setosa
# 3 4.7 3.2 1.3 0.2 setosa
# 4 4.6 3.1 1.5 0.2 setosa
# 5 5 3.6 1.4 0.2 setosa
# 6 5.4 3.9 1.7 0.4 setosa
# 7 4.6 3.4 1.4 0.3 setosa
# 8 5 3.4 1.5 0.2 setosa
# 9 4.4 2.9 1.4 0.2 setosa
# 10 4.9 3.1 1.5 0.1 setosa
# # … with 140 more rowsVoici maintenant ce que cela donne en utilisant les verbes de {dplyr}. La fonction explain() permet d’expliquer ce qui est fait.
# # Source: lazy query [?? x 4]
# # Database: sqlite 3.30.1 [/media/sf_shared/database.sqlite]
# Sepal.Width Petal.Length Petal.Width Species
# <dbl> <dbl> <dbl> <chr>
# 1 3.5 1.4 0.2 setosa
# 2 3 1.4 0.2 setosa
# 3 3.2 1.3 0.2 setosa
# 4 3.1 1.5 0.2 setosa
# 5 3.6 1.4 0.2 setosa
# 6 3.9 1.7 0.4 setosa
# 7 3.4 1.4 0.3 setosa
# 8 3.4 1.5 0.2 setosa
# 9 2.9 1.4 0.2 setosa
# 10 3.1 1.5 0.1 setosa
# # … with more rows# # Source: lazy query [?? x 5]
# # Database: sqlite 3.30.1 [/media/sf_shared/database.sqlite]
# Sepal.Length Sepal.Width Petal.Length Petal.Width Species
# <dbl> <dbl> <dbl> <dbl> <chr>
# 1 5.4 3.9 1.7 0.4 setosa
# 2 4.8 3.4 1.6 0.2 setosa
# 3 5.7 3.8 1.7 0.3 setosa
# 4 5.4 3.4 1.7 0.2 setosa
# 5 5.1 3.3 1.7 0.5 setosa
# 6 4.8 3.4 1.9 0.2 setosa
# 7 5 3 1.6 0.2 setosa
# 8 5 3.4 1.6 0.4 setosa
# 9 4.7 3.2 1.6 0.2 setosa
# 10 4.8 3.1 1.6 0.2 setosa
# # … with more rows# Réarranger les lignes par longueur de pétale croissante et largeur de sépale décroissant
arrange(my_table, Petal.Length, desc(Sepal.Width))# # Source: SQL [?? x 5]
# # Database: sqlite 3.30.1 [/media/sf_shared/database.sqlite]
# # Ordered by: Petal.Length, desc(Sepal.Width)
# Sepal.Length Sepal.Width Petal.Length Petal.Width Species
# <dbl> <dbl> <dbl> <dbl> <chr>
# 1 4.6 3.6 1 0.2 setosa
# 2 4.3 3 1.1 0.1 setosa
# 3 5.8 4 1.2 0.2 setosa
# 4 5 3.2 1.2 0.2 setosa
# 5 5.4 3.9 1.3 0.4 setosa
# 6 5.5 3.5 1.3 0.2 setosa
# 7 5 3.5 1.3 0.3 setosa
# 8 4.7 3.2 1.3 0.2 setosa
# 9 4.4 3.2 1.3 0.2 setosa
# 10 4.4 3 1.3 0.2 setosa
# # … with more rows# # Source: lazy query [?? x 6]
# # Database: sqlite 3.30.1 [/media/sf_shared/database.sqlite]
# Sepal.Length Sepal.Width Petal.Length Petal.Width Species logPL
# <dbl> <dbl> <dbl> <dbl> <chr> <dbl>
# 1 5.1 3.5 1.4 0.2 setosa 0.146
# 2 4.9 3 1.4 0.2 setosa 0.146
# 3 4.7 3.2 1.3 0.2 setosa 0.114
# 4 4.6 3.1 1.5 0.2 setosa 0.176
# 5 5 3.6 1.4 0.2 setosa 0.146
# 6 5.4 3.9 1.7 0.4 setosa 0.230
# 7 4.6 3.4 1.4 0.3 setosa 0.146
# 8 5 3.4 1.5 0.2 setosa 0.176
# 9 4.4 2.9 1.4 0.2 setosa 0.146
# 10 4.9 3.1 1.5 0.1 setosa 0.176
# # … with more rowsIl est possible de réaliser des choses plus complexes ! On peut naturellement chaîner tout cela avec le pipe %>.%, ou combiner les requêtes comme on veut. Rien n’est fait avant de collect()er les résultats.
my_table %>.%
filter(., Petal.Length > 1.5) %>.%
select(., Petal.Length, Sepal.Width, Species) %>.%
mutate(., logPL = log10(Petal.Length)) -> query1
query2 <- arrange(query1, Petal.Length, desc(Sepal.Width))
query2# # Source: lazy query [?? x 4]
# # Database: sqlite 3.30.1 [/media/sf_shared/database.sqlite]
# # Ordered by: Petal.Length, desc(Sepal.Width)
# Petal.Length Sepal.Width Species logPL
# <dbl> <dbl> <chr> <dbl>
# 1 1.6 3.8 setosa 0.204
# 2 1.6 3.5 setosa 0.204
# 3 1.6 3.4 setosa 0.204
# 4 1.6 3.4 setosa 0.204
# 5 1.6 3.2 setosa 0.204
# 6 1.6 3.1 setosa 0.204
# 7 1.6 3 setosa 0.204
# 8 1.7 3.9 setosa 0.230
# 9 1.7 3.8 setosa 0.230
# 10 1.7 3.4 setosa 0.230
# # … with more rows# # A tibble: 113 x 4
# Petal.Length Sepal.Width Species logPL
# <dbl> <dbl> <chr> <dbl>
# 1 1.6 3.8 setosa 0.204
# 2 1.6 3.5 setosa 0.204
# 3 1.6 3.4 setosa 0.204
# 4 1.6 3.4 setosa 0.204
# 5 1.6 3.2 setosa 0.204
# 6 1.6 3.1 setosa 0.204
# 7 1.6 3 setosa 0.204
# 8 1.7 3.9 setosa 0.230
# 9 1.7 3.8 setosa 0.230
# 10 1.7 3.4 setosa 0.230
# # … with 103 more rowsEnfin, la fonction sql_translate() du package {dbply} va indiquer comment une instruction R est convertie en code SQL équivalent. C’est très pratique aussi pour apprendre SQL quand on connait R !
# <SQL> POWER(`x`, 3.0) < 15.0 OR `y` > 20.0# Warning: Missing values are always removed in SQL.
# Use `AVG(x, na.rm = TRUE)` to silence this warning
# This warning is displayed only once per session.# <SQL> AVG(`x`) OVER ()# <SQL> AVG(`x`) OVER ()# Tout ne fonctionne pas, car R offre plus de possibilités que SQL
dbplyr::translate_sql(plot(x)) #???# <SQL> plot(`x`)# Error in mean(x, trim = TRUE): unused argument (trim = TRUE)Voilà pour ce très rapide tour d’horizon des différentes façons de manipuler des bases de données avec R et RStudio. En pratique, revenez sur cette section, et approfondissez vos connaissances via les ressources proposées ci-dessous lorsque vous serez confrontés “en vrai” à des données présentées dans une base de données relationnelle.
Pour en savoir plus
RStudio et bases de données : tout un site web dédié à l’accès aux bases de données depuis RStudio (en anglais).
La documentation de DB Browser for SQLite (en anglais).
Une introduction des requêtes SQL dans R un peu plus développée (en anglais).
Un tutorial SQL avec des exercices (en anglais).
Un cours en ligne sur SQL par vidéos par la Kahn Academy (en anglais).
Un autre tutorial complet sur SQL. Remarquez qu’il en existe beaucoup. Faites une recherche via Google et choisissez le tutoriel qui vous plaît le plus.
Les dix commandements d’une base de données réussie. Il s’agit ici plutôt de la création d’une base de données que de la requête sur une base de données existante… mais tôt ou tard, vous créerez vos propres bases de données et ces conseils vous seront alors utiles.
À vous de jouer !
Note: Ce projet est un travail par groupe de 4 personnes. Ce projet est transmodule, ce qui signifie que vous reviendrez dessus à plusieurs reprises au fur et à mesure de votre progression dans la matière afin de compléter votre analyse de ces données.
Réalisez en groupe l’assignation B07Ga_doubs.
Si vous êtes un utilisateur non enregistré ou que vous travaillez en dehors d’un cours, faites un “fork” de ce dépôt.
Voyez les explications dans le fichier README.md.